Содержание
- 2. HLA типирование - исследование главного комплекса гистосовместимости человека - HLA комплекса. Это образование включает в себя
- 3. Задачи Биологическая идентификация (HLA-тип наследуется вместе с родительскими генами), определение предрасположенности к различным заболеваниям, подбор доноров
- 4. HLA-типирование методом секвенирования По Сэнгеру NGS: Roche/454 Life Sciences Illumina/Solexa Applied Biosystems/SOLiD Одномолекулярное секвенирование Helicos Biosciences
- 5. HLA-типирование основанное на технологии секвенирования по Сэнгеру HLA-секвенирование по Сэнгеру является золотым стандартом в высокоразрешающем HLA-типировании
- 6. В классическом варианте метода Сэнгера одна из цепочек анализируемой ДНК выступает в качестве матрицы для синтеза
- 11. На сегодняшний день секвенирование ДНК по Сэнгеру полностью автоматизировано и проводится на специальных приборах, секвенаторах. Использование
- 12. HLA-типирование методом секвенирования следующего поколения (NGS) Секвенирование нового поколения— техника определения нуклеотидной последовательности ДНК и РНК
- 13. Основные принципы всех методов Все основные принципы работы технологий СНП базируются на секвенировании ДНК-чипов, используя интерактивные
- 14. Этапы Первый этап секвенирования — создание библиотеки случайных последовательностей ДНК, которые можно будет сшить с общедоступными
- 16. Roche/454 Life Sciences Первая эффективно используемая на коммерческой основе платформа СНП. Компания 454 Life Sciences основана
- 18. Illumina/Solexa Авторы метода — британские химики Шанкар Баласубраманиан и Дэвид Кленерман. Этот метод секвенирования использует прикреплённые
- 20. Applied Biosystems/SOLiD Платформа SOLiD (Supported Oligonucleotide Ligation and Detection System 2.0), разработанная Applied Biosystems — технология
- 22. Одномолекулярное секвенирование Helicos Biosciences Первый метод секвенирования единичных молекул, разработанный HeliScope имеет производительность около 1Gb/день. Принцип
- 23. Одномолекулярное секвенирование в реальном времени Pacific Biosciences Метод одномолекулярного секвенирование в реальном времени (SMRT) основан на
- 25. Ion Torrent Sequencing Метод основан на связи между химической и цифровой информацией, что позволяет быстрее и
- 28. Нанопоровое секвенирование Метод основан на измерении тока ионов через единичную нанопору в непроводящей мембране. При прохождении
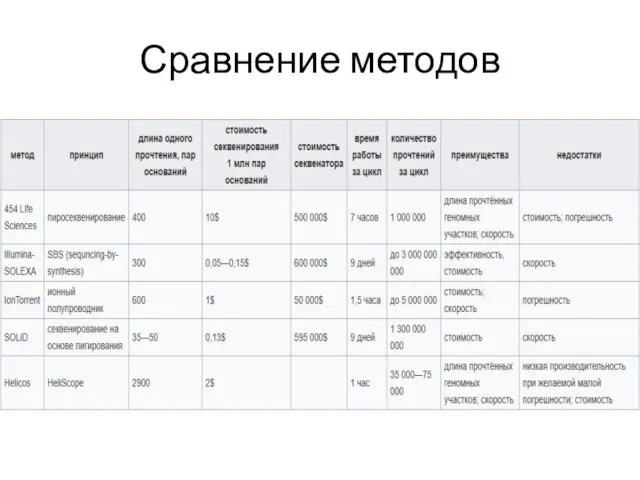
- 30. Сравнение методов
- 32. Скачать презентацию




























 Отряд приматы
Отряд приматы Птицы тундры. Выполнила: Серых Виктория, 6 класс МКОУ Пензинская ООШ Новосибирская область Барабинский район Руководитель:
Птицы тундры. Выполнила: Серых Виктория, 6 класс МКОУ Пензинская ООШ Новосибирская область Барабинский район Руководитель:  Презентация на тему "Лист – часть побега. Значение листа для растения" - скачать презентации по Биологии
Презентация на тему "Лист – часть побега. Значение листа для растения" - скачать презентации по Биологии Презентация на тему Семейство Папоротниковые
Презентация на тему Семейство Папоротниковые Аттестационная работа. Методическая разработка по выполнению проекта Подгруппа углерода для 9 класса по химии и экологии
Аттестационная работа. Методическая разработка по выполнению проекта Подгруппа углерода для 9 класса по химии и экологии Дощові черві
Дощові черві Экологическое исследование почвы на территории Белоярского района
Экологическое исследование почвы на территории Белоярского района Творчество Виктора Петровича Астафьева
Творчество Виктора Петровича Астафьева Рудольф Вирхов – отец патологии
Рудольф Вирхов – отец патологии Учение Чарльза Дарвина
Учение Чарльза Дарвина  Презентация на тему: «Исчезающие виды растений». Красота растений не в букетах, а там, где они растут! Не рви растения! Лучше научис
Презентация на тему: «Исчезающие виды растений». Красота растений не в букетах, а там, где они растут! Не рви растения! Лучше научис Абиотические и биотические факторы среды
Абиотические и биотические факторы среды Внутренняя среда организма. Значение крови. 4 класс
Внутренняя среда организма. Значение крови. 4 класс Формирование дидактической системы учителя средствами ИКТ
Формирование дидактической системы учителя средствами ИКТ Эволюция жизни на Земле в палеозойскую эру
Эволюция жизни на Земле в палеозойскую эру Урок биологии в 8 классе. Тема: Среда обитания и внешнее строение млекопитающих.
Урок биологии в 8 классе. Тема: Среда обитания и внешнее строение млекопитающих.  Презентация на тему "Дельфинотерапия как нетрадиционный метод психотерапии" - скачать презентации по Биологии
Презентация на тему "Дельфинотерапия как нетрадиционный метод психотерапии" - скачать презентации по Биологии Біологічний прогрес і біологічний регрес.
Біологічний прогрес і біологічний регрес.  Физиология центральной нервной системы
Физиология центральной нервной системы Сказочный гербарий
Сказочный гербарий Рекомендации по созданию презентации на уроках биологии
Рекомендации по созданию презентации на уроках биологии Сальтационная теория эволюции
Сальтационная теория эволюции Презентация на тему "Бобовые" - скачать презентации по Биологии
Презентация на тему "Бобовые" - скачать презентации по Биологии Класс брюхоногие
Класс брюхоногие Муниципальное общеобразовательное учреждение средняя общеобразовательная школа № 2 г. Будённовска Урок биологии: Влияние асоци
Муниципальное общеобразовательное учреждение средняя общеобразовательная школа № 2 г. Будённовска Урок биологии: Влияние асоци экологических факторах. Основные среды жизни.
экологических факторах. Основные среды жизни.  Выращивание узамбарской фиалки в домашних условиях
Выращивание узамбарской фиалки в домашних условиях Презентация на тему "Удивительные плоды" - скачать презентации по Биологии
Презентация на тему "Удивительные плоды" - скачать презентации по Биологии