Содержание
- 2. Раздел дисциплины Методы изучения генома, основные понятия и определения. Биоинформационные подходы, позволяющие планировать эксперименты. Дизайн праймеров.
- 3. Основная литература: 1. И.В. Бабкин, Н.В. Тикунова, С.В. Нетесов. Компьютерные методы анализа нуклеотидных последовательностей. Новосибирский государственный
- 4. Полезные Интернет-сайты: 1. httphttp://http://wwwhttp://www.http://www.ncbihttp://www.ncbi.http://www.ncbi.nlmhttp://www.ncbi.nlm.http://www.ncbi.nlm.nihhttp://www.ncbi.nlm.nih.http://www.ncbi.nlm.nih.govhttp://www.ncbi.nlm.nih.gov/ 2. httphttp://http://wwwhttp://www.http://www.ebihttp://www.ebi.http://www.ebi.achttp://www.ebi.ac.http://www.ebi.ac.ukhttp://www.ebi.ac.uk/ 3. http://web.expasy.org/ 4. httphttp://http://evolutionhttp://evolution.http://evolution.geneticshttp://evolution.genetics.http://evolution.genetics.washingtonhttp://evolution.genetics.washington.http://evolution.genetics.washington.eduhttp://evolution.genetics.washington.edu/http://evolution.genetics.washington.edu/phyliphttp://evolution.genetics.washington.edu/phylip/http://evolution.genetics.washington.edu/phylip/softwarehttp://evolution.genetics.washington.edu/phylip/software.http://evolution.genetics.washington.edu/phylip/software.htmlhttp://evolution.genetics.washington.edu/phylip/software.html#http://evolution.genetics.washington.edu/phylip/software.html#methods 5. httphttp://http://blasthttp://blast.http://blast.ncbihttp://blast.ncbi.http://blast.ncbi.nlmhttp://blast.ncbi.nlm.http://blast.ncbi.nlm.nihhttp://blast.ncbi.nlm.nih.http://blast.ncbi.nlm.nih.govhttp://blast.ncbi.nlm.nih.gov/http://blast.ncbi.nlm.nih.gov/Blasthttp://blast.ncbi.nlm.nih.gov/Blast.http://blast.ncbi.nlm.nih.gov/Blast.cgi 6. httphttp://http://maffthttp://mafft.http://mafft.cbrchttp://mafft.cbrc.http://mafft.cbrc.jphttp://mafft.cbrc.jp/http://mafft.cbrc.jp/alignmenthttp://mafft.cbrc.jp/alignment/http://mafft.cbrc.jp/alignment/serverhttp://mafft.cbrc.jp/alignment/server/http://mafft.cbrc.jp/alignment/server/indexhttp://mafft.cbrc.jp/alignment/server/index.http://mafft.cbrc.jp/alignment/server/index.html 7. httphttp://http://unafoldhttp://unafold.http://unafold.rnahttp://unafold.rna.http://unafold.rna.albanyhttp://unafold.rna.albany.http://unafold.rna.albany.eduhttp://unafold.rna.albany.edu/?http://unafold.rna.albany.edu/?qhttp://unafold.rna.albany.edu/?q=http://unafold.rna.albany.edu/?q=mfoldhttp://unafold.rna.albany.edu/?q=mfold/http://unafold.rna.albany.edu/?q=mfold/DNAhttp://unafold.rna.albany.edu/?q=mfold/DNA-http://unafold.rna.albany.edu/?q=mfold/DNA-Foldinghttp://unafold.rna.albany.edu/?q=mfold/DNA-Folding-http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form
- 5. Освежим свою память ДНК, РНК, нуклеотид, нуклеозид Основная парадигма молекулярной биологии
- 6. Терминология: Вектор – самореплицирующая молекула ДНК (например, бактериальная плазмида), используемая в генетической инженерии для переноса генов
- 7. Современная молекулярная биология немыслима без привлечения компьютеров. История 1202 г. — Леонардо Пизанский (Фибоначчи) опубликовал книгу
- 8. 1967 г. — Пер Эдман совместно с Джофри Бэггом создали первый автоматический белковый секвенатор. 1970 г.
- 9. 1986 г. — Первый автоматический ДНК-секвенатор (Applied Biosystems, США). 1990 г. — Разработали программу BLAST. 1990
- 10. Используемые форматы данных FASTA-формат >Rattus_norvegicus | Rattus norvegicus heat shock 20kDa protein (Loc192245), mRNA GCAGGATGGAGATCCGGGTGCCTGTGCAGCCTTCTTGGCTGCGCCGTGCTTCAGCTCCTTTACCGGGTTTTTCCACTCCGGGACGCCTCTTTGACCAGCGTTTCGGCGAAGGGCTGCTTGAGGCAGAGCTGGCTTC >Homo_sapiens
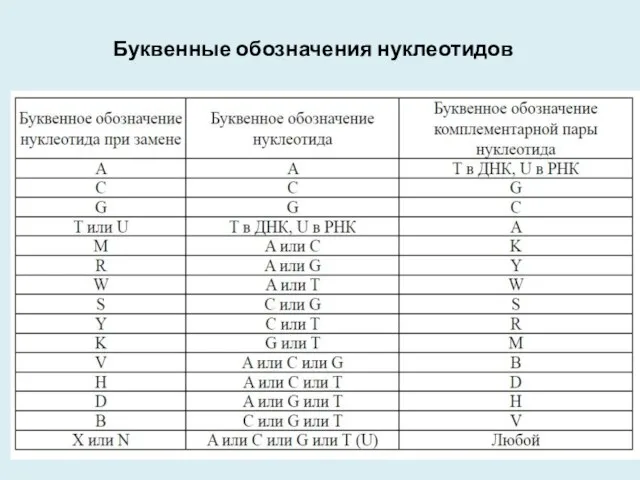
- 11. Буквенные обозначения нуклеотидов
- 12. Nexus файл #NEXUS Begin data; Dimensions ntax=4 nchar=15; Format datatype=dna missing=? gap=-; Matrix Species1 atgctagctagctcg Species2
- 13. Nexus файл Файлы данных Nexus всегда начинаются с символов #nexus, но в остальном организованы в основные
- 14. GB-формат
- 15. GB-формат
- 16. GB-формат
- 18. Скачать презентацию














 Тема: Полимерные трохофорные: Тип Annelida (Кольчатые черви) Тип Pogonophora (Погонофоры)
Тема: Полимерные трохофорные: Тип Annelida (Кольчатые черви) Тип Pogonophora (Погонофоры)  Надежная защита организма
Надежная защита организма Магония падуболистная
Магония падуболистная Круговорот азота в природе
Круговорот азота в природе Спортивная биохимия. (Часть 3)
Спортивная биохимия. (Часть 3) Комнатные растения Диффенбахия Выполнила Шабалина Алина
Комнатные растения Диффенбахия Выполнила Шабалина Алина Чередова Ольга Максимовна Учитель биологии, МКОУ «СОШ №2» п. Сывдарма
Чередова Ольга Максимовна Учитель биологии, МКОУ «СОШ №2» п. Сывдарма Сельва - влажные экваториальные леса в Южной Америке
Сельва - влажные экваториальные леса в Южной Америке Вирусы. Классификация вирусов
Вирусы. Классификация вирусов Основы иммунологии в картинках
Основы иммунологии в картинках Презентация на тему "В гости к министру экологии" - скачать презентации по Биологии
Презентация на тему "В гости к министру экологии" - скачать презентации по Биологии Презентация по биологии ЖИВОТНЫЕ ПРОШЛОГО
Презентация по биологии ЖИВОТНЫЕ ПРОШЛОГО Презентация по экологии планета Земля
Презентация по экологии планета Земля ДНК Підготував: Учень 11-А класу Долинка Ігор
ДНК Підготував: Учень 11-А класу Долинка Ігор  Каменск – Уральский техникум торговли и сервиса
Каменск – Уральский техникум торговли и сервиса Презентация по биологии Вирусные Заболевание животных
Презентация по биологии Вирусные Заболевание животных  The story of British zoos
The story of British zoos Предмет и задачи микробиологии и иммунологии
Предмет и задачи микробиологии и иммунологии Агамы
Агамы Сорокина В.Ю. Онтогенез
Сорокина В.Ю. Онтогенез История изучения клетки. Клеточная теория
История изучения клетки. Клеточная теория Функциональная анатомия ствола головного мозга и конечного мозга
Функциональная анатомия ствола головного мозга и конечного мозга Многообразие цветковых растений
Многообразие цветковых растений Вред и польза сорняков
Вред и польза сорняков Стриопаллидарная система
Стриопаллидарная система Криосохранение крупных биологических объектов
Криосохранение крупных биологических объектов Класс Земноводные (или амфибии)
Класс Земноводные (или амфибии) 6 Б класс МОУ СОШ №18 г. Иркутска Учитель Бабич Т.Р.
6 Б класс МОУ СОШ №18 г. Иркутска Учитель Бабич Т.Р.