Содержание
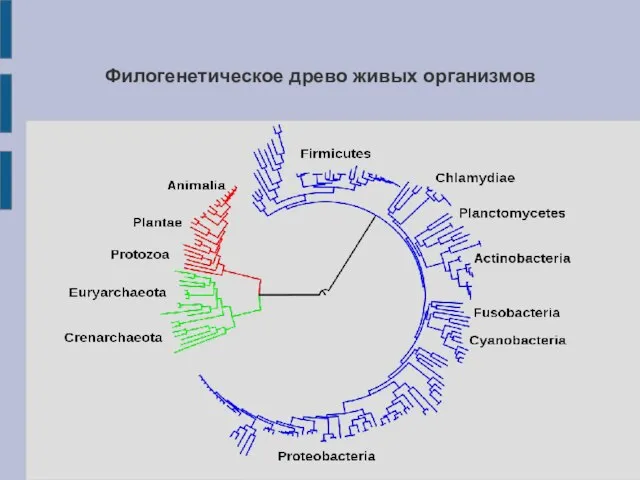
- 2. Филогенетическое древо живых организмов
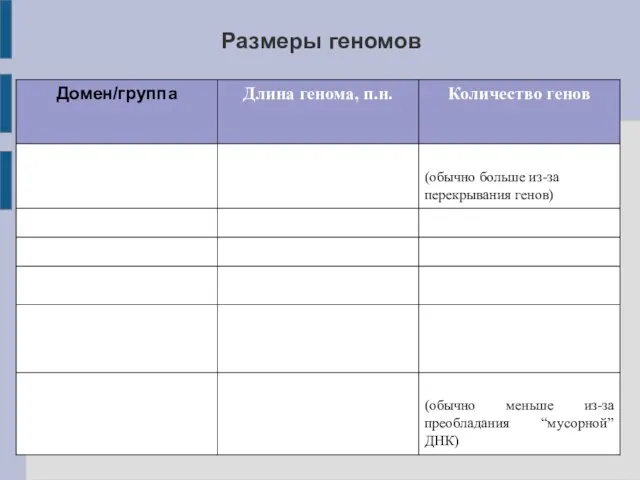
- 3. Размеры геномов
- 4. Открытая рамка считывания (Open Reading Frame, ORF) Потенциально кодирующая последовательность в геноме: Начинается с инициирующего кодона
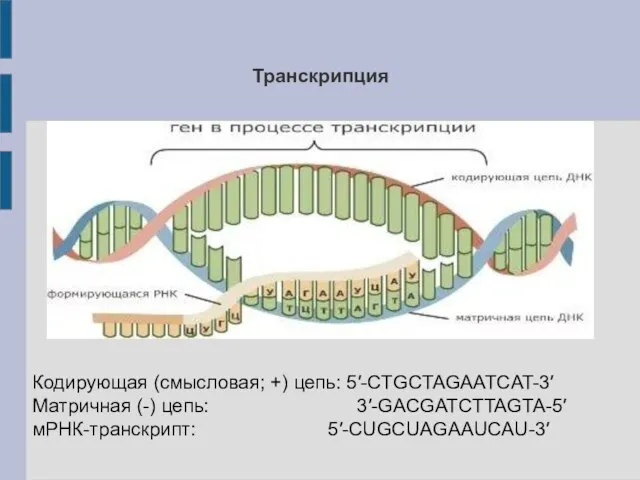
- 5. Транскрипция Кодирующая (смысловая; +) цепь: 5′-CTGCTAGAATCAT-3′ Матричная (-) цепь: 3′-GACGATCTTAGTA-5′ мРНК-транскрипт: 5′-CUGCUAGAAUCAU-3′
- 6. Особенности геномов прокариот (бактерий и архей) Сравнительно малый размер (~105-107п.н.) Гаплоидность Унипартитность/Бипартитность (одна-две разные хромосомы) Распространённость
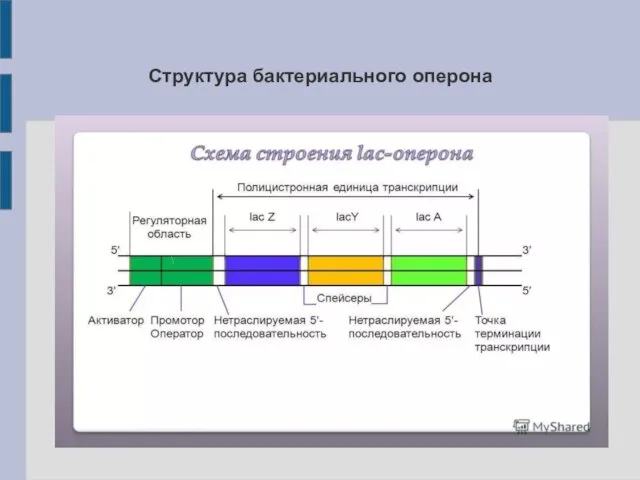
- 7. Структура бактериального оперона
- 8. Особенности геномов эукариот Сравнительно большой размер (~108-109 п.н.) Мультипартитность и мультиплоидность Организация в виде хроматина, имеющего
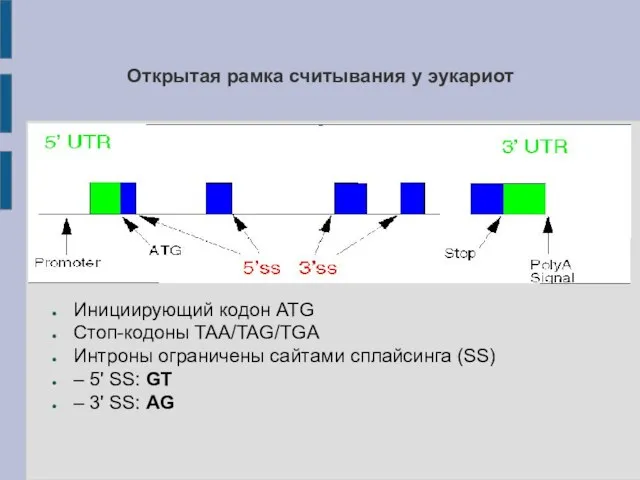
- 9. Открытая рамка считывания у эукариот Инициирующий кодон ATG Стоп-кодоны TAA/TAG/TGA Интроны ограничены сайтами сплайсинга (SS) –
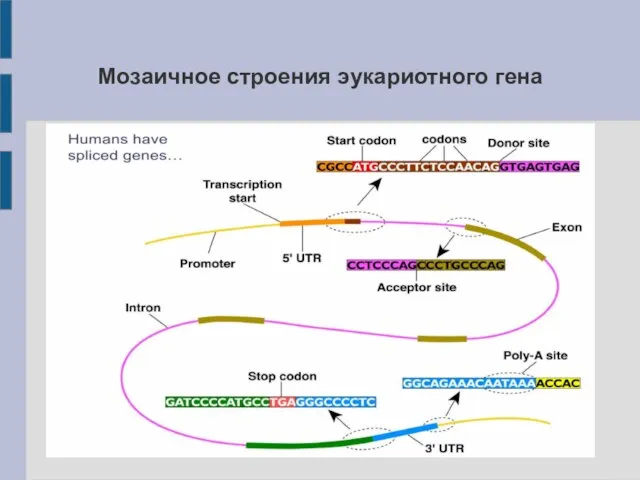
- 10. Мозаичное строения эукариотного гена
- 11. Инфраструктура генома Кодирующие последовательности (гены) – белковые (структурные и регуляторные) – РНК-гены – гены мобильных генетических
- 12. Гены белков Однокопийные/многокопийные Паралогичные Гены-сироты (orphan genes) Криптические (молчащие) Псевдогены Взаимноперекрывающиеся
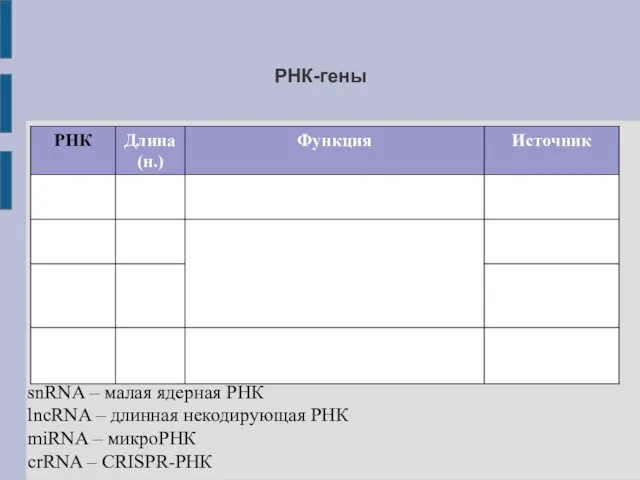
- 13. РНК-гены snRNA – малая ядерная РНК lncRNA – длинная некодирующая РНК miRNA – микроРНК crRNA –
- 14. Повторяющиеся некодирующие последовательности короткие ( 100 п.н.) прямые или инвертированные тандемные или диспергированные
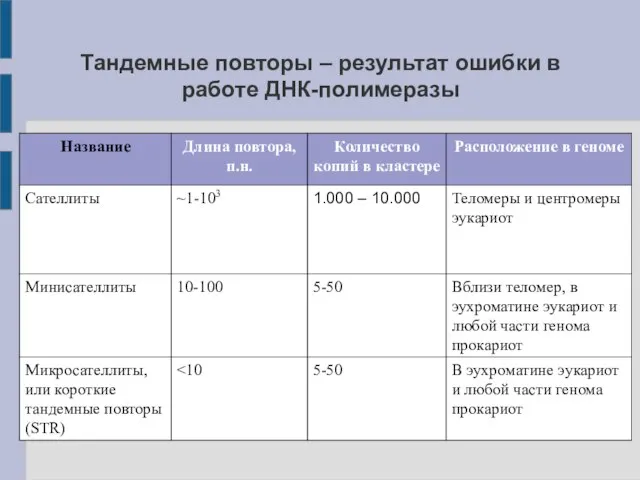
- 15. Тандемные повторы – результат ошибки в работе ДНК-полимеразы
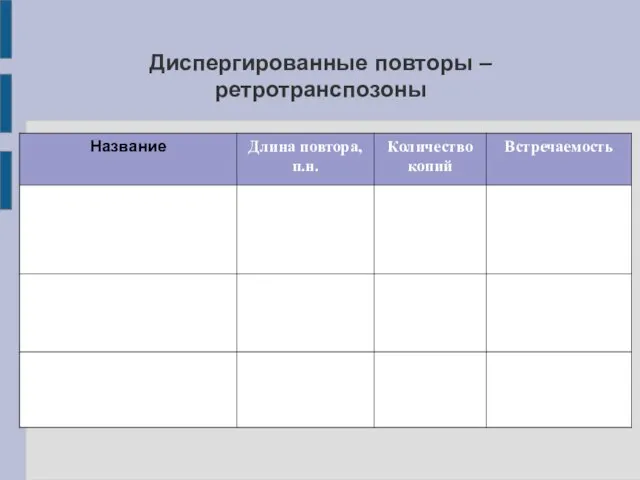
- 16. Диспергированные повторы – ретротранспозоны
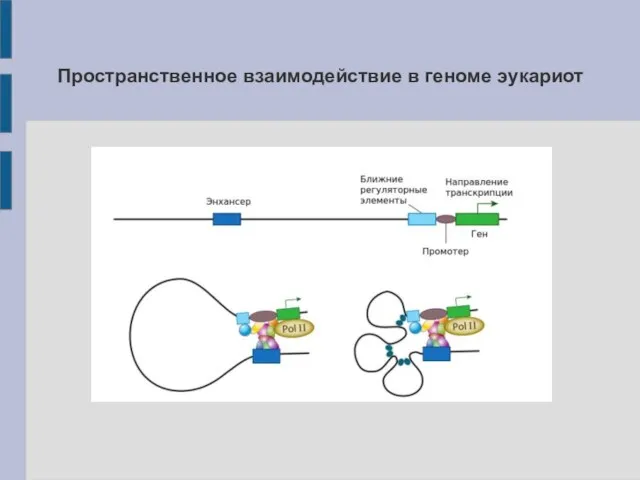
- 17. Пространственное взаимодействие в геноме эукариот
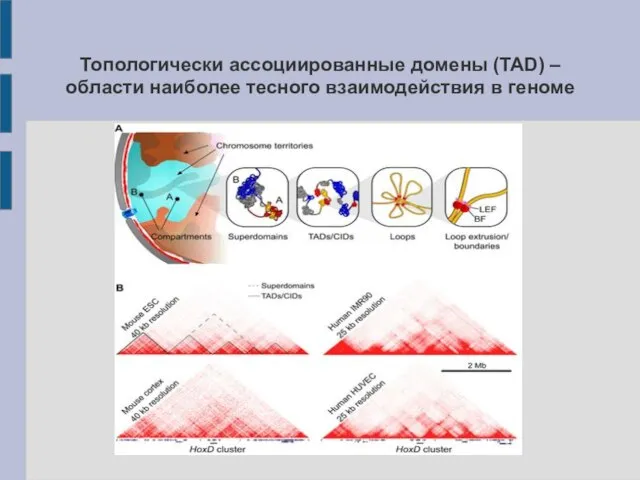
- 18. Топологически ассоциированные домены (TAD) – области наиболее тесного взаимодействия в геноме
- 19. Генетический полиморфизм 1. Явление существования вариантов в нуклеотидной последовательности разных представителей одного вида. 2. Собственно вариант
- 20. Виды генетических полиморфизмов Однонуклеотидный полиморфизм (ОНП; single nucleotide polymorphism, SNP, ‘snip’) Инсерция/делеция (индел) – простая –
- 21. SNPs Самый частый вид полиморфизма Частота: 1 нуклеотид на каждую 1000 п.н. (~3*106 различий между любыми
- 22. Полиморфизм тандемных повторов 1. Полиморфизм числа тандемных повторов – VNTR (полиморфные минисателлиты, отличающиеся числом повторов в
- 23. Виды SNP в зависимости от природы меняющихся азотистых оснований Транзиции (~2/3 всех SNP) – пуриновое основание
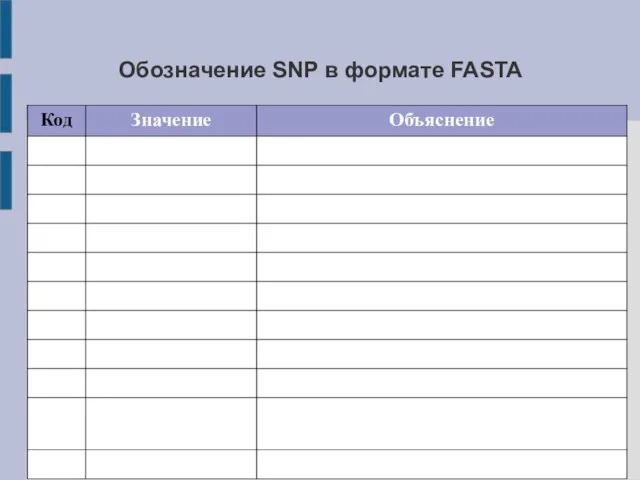
- 24. Обозначение SNP в формате FASTA
- 25. Номенклатура полиморфизмов 1. Указание источника последовательности геномная ДНК (g.) комплементарная ДНК (c.) последовательность белка (p.) мтДНК
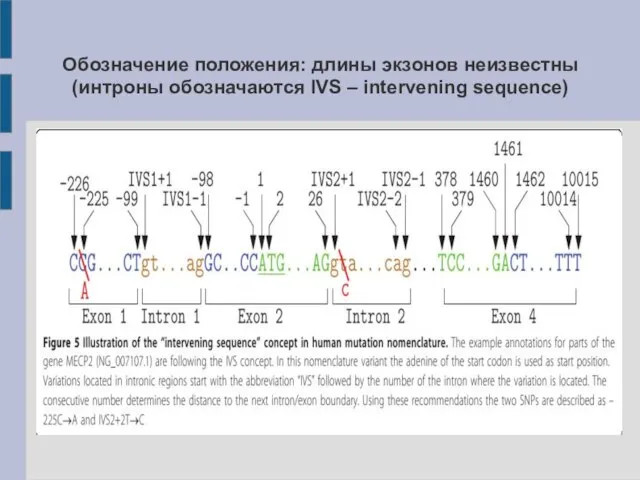
- 26. Обозначение положения: длины экзонов неизвестны (интроны обозначаются IVS – intervening sequence)
- 27. Обозначение положения: длины экзонов известны
- 28. Примеры записи SNPs c.1777A>C замена аденина на цитозин в положении 1777 кДНК IVS33-2A>T замена аденина на
- 29. Использование уникального идентификатора для обозначения SNP База данных dbSNP (>55 организмов, >63 млн. SNP): каждый SNP
- 30. Простая инсерция/делеция Однократная вставка/удаление короткого участка ДНК. Может приводить к сдвигу рамки считывания. Номенклатура: c.441delA делеция
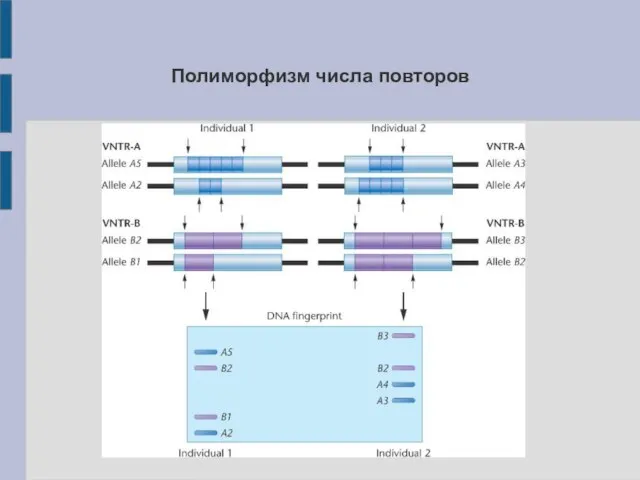
- 31. Полиморфизм числа повторов
- 33. Скачать презентацию
















 Глотка, пищевод и желудок
Глотка, пищевод и желудок Социальный проект
Социальный проект  Эволюция человека
Эволюция человека Составитель: Серебрянская Н.А., учитель биологии МОУ «Образцовская СОШ» Фроловского муниципального района Вольгоградской области
Составитель: Серебрянская Н.А., учитель биологии МОУ «Образцовская СОШ» Фроловского муниципального района Вольгоградской области Водоросли. 5 класс
Водоросли. 5 класс Флора мира Путешествие в мир деревьев
Флора мира Путешествие в мир деревьев Презентация на тему "Грибы-паразиты и меры борьбы с ними" - скачать презентации по Биологии
Презентация на тему "Грибы-паразиты и меры борьбы с ними" - скачать презентации по Биологии Презентация на тему "Надкласс Рыбы. Общая характеристика. Внешнее строение рыб" - скачать презентации по Биологии
Презентация на тему "Надкласс Рыбы. Общая характеристика. Внешнее строение рыб" - скачать презентации по Биологии Вода в теле человека
Вода в теле человека Значение нервной системы
Значение нервной системы Лоси Выполнил Баранов Кирилл
Лоси Выполнил Баранов Кирилл  «Охраняемые растения Апалихинского округа.» Работу выполнили ученицы 10 класса Кузьмичёва А., Салихова А. учитель: Мирон
«Охраняемые растения Апалихинского округа.» Работу выполнили ученицы 10 класса Кузьмичёва А., Салихова А. учитель: Мирон Лекция 7. Активный обмен. Энергобаланс
Лекция 7. Активный обмен. Энергобаланс Клеточное ядро
Клеточное ядро Аттестационная работа. Выращивание лука в домашних условиях 2 класс
Аттестационная работа. Выращивание лука в домашних условиях 2 класс Многообразие водорослей
Многообразие водорослей  75 лет – Дарвинскому государственному природному биосферному заповеднику
75 лет – Дарвинскому государственному природному биосферному заповеднику Способы размножения растений
Способы размножения растений Презентация на тему "Царство прокариоты, подцарство бактерии" - скачать презентации по Биологии
Презентация на тему "Царство прокариоты, подцарство бактерии" - скачать презентации по Биологии Презентация по биологии РЫБЫ
Презентация по биологии РЫБЫ Домашние животные
Домашние животные Презентация на тему " МЫ ЗА ЗДОРОВЫЙ ОБРАЗ ЖИЗНИ!" - скачать презентации по Биологии
Презентация на тему " МЫ ЗА ЗДОРОВЫЙ ОБРАЗ ЖИЗНИ!" - скачать презентации по Биологии Биология лося и северного оленя
Биология лося и северного оленя Презентация на тему "Водоросли: строение и виды" - скачать презентации по Биологии
Презентация на тему "Водоросли: строение и виды" - скачать презентации по Биологии Значение и строение кожи
Значение и строение кожи Проект на тему: ліс – легені нашої планети
Проект на тему: ліс – легені нашої планети Презентация на тему "Травмы глаз" - скачать бесплатно презентации по Биологии
Презентация на тему "Травмы глаз" - скачать бесплатно презентации по Биологии Хімічні елементи живих організмів. Порушення. 10 клас
Хімічні елементи живих організмів. Порушення. 10 клас