Содержание
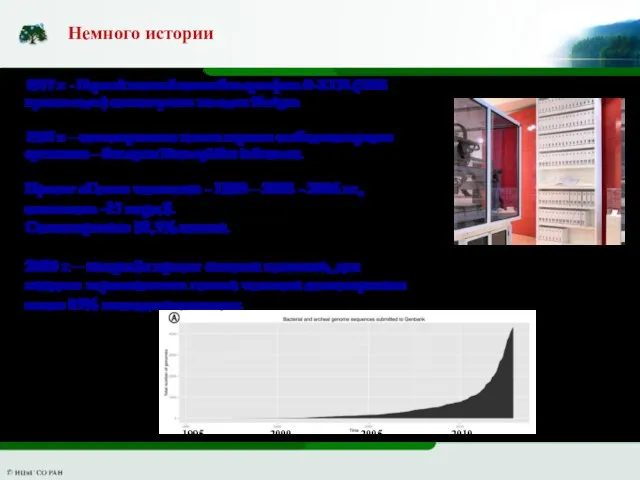
- 2. Немного истории 1977 г. - Первый полный геном бактериофага Φ-X174 (5386 нуклеотидов) секвенирован методом Shotgun 1995
- 3. «Расшифровка» генома Розеттский камень Характерные размеры геномов вирус папиллом человека - 8 т.п.о Escherichia coli -
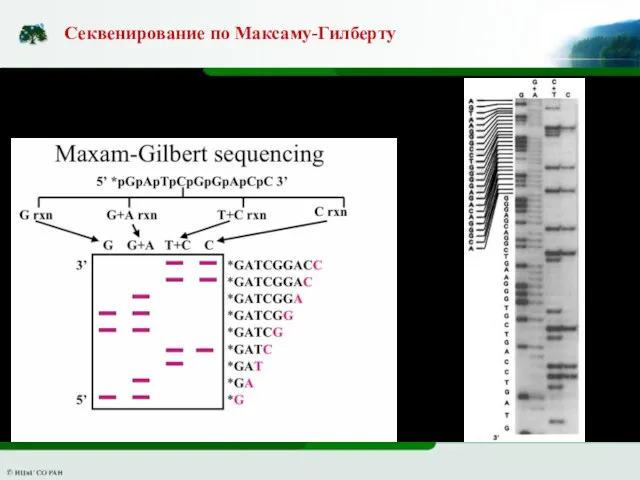
- 4. Секвенирование по Максаму-Гилберту Химическая деградация ДНК
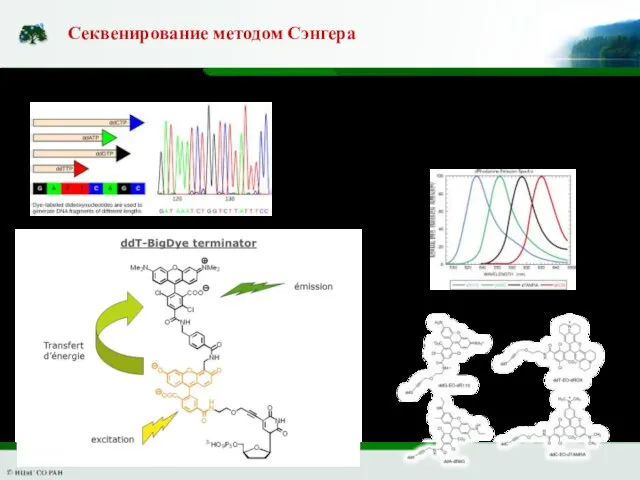
- 5. Реакция Сэнгера – не ПЦР, а многократный синтез с одной матрицы Секвенирование методом Сэнгера
- 6. 96-capillary ABI 3730XL Первое поколение секвенаторов - капиллярные Нанофор 05 – российская модель
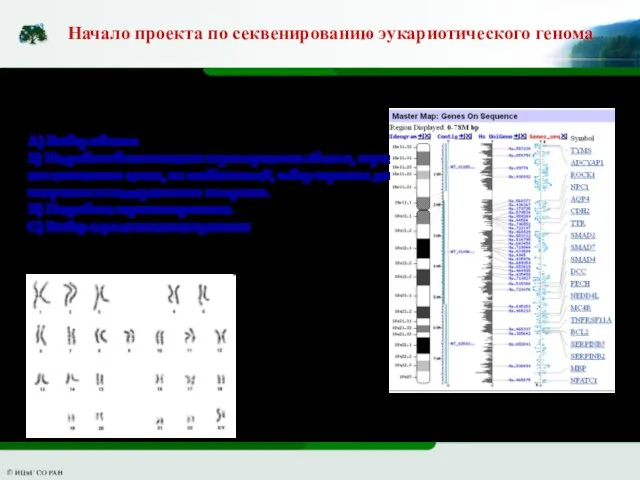
- 7. Начало проекта по секвенированию эукариотического генома А) Выбор объекта Б) Подробная биологическая характеристика объекта, изучение его
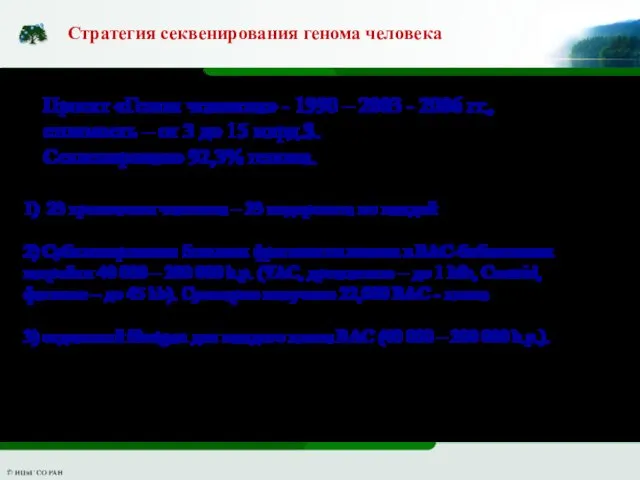
- 8. Стратегия секвенирования генома человека 1) 23 хромосомы человека – 23 подпроекта по каждой 2) Субклонирование больших
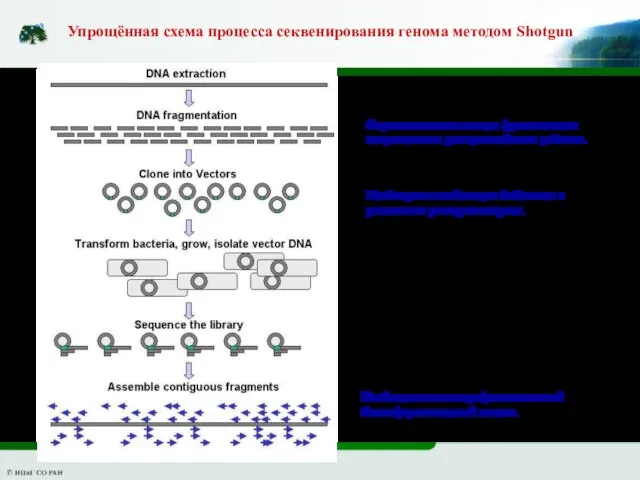
- 9. Упрощённая схема процесса секвенирования генома методом Shotgun Ферментативные методы фрагментации неприменимы для протяжённых районов. Необходима комбинация
- 10. Подробнее Используемые векторы – ранее М13 phagemid Вентер – производная pBR322, Говорун - pCR2.1 (Invitrogen) Лейшмания
- 11. 1998г. – старт проекта секвенирования первого индивидуального генома человека. Крейг Вентер, компания «Celera Genomics» Использование метода
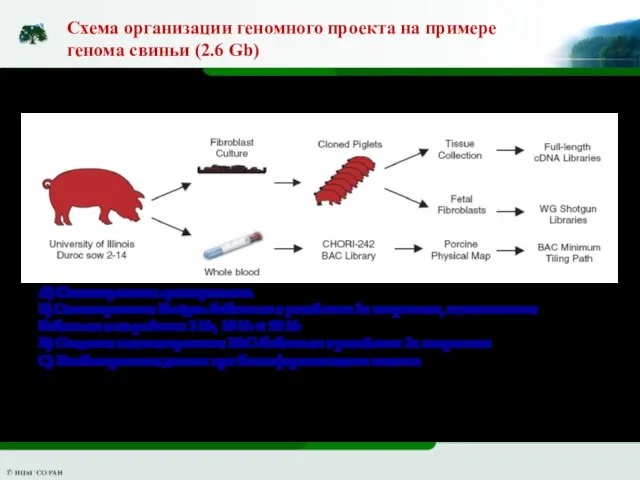
- 12. Schook et al., 2005 Схема организации геномного проекта на примере генома свиньи (2.6 Gb) А) Секвенирование
- 13. Phred 10 = 90% точность, Phred 20 = 99%, Phred 30 = 99,9%, Phred quality score
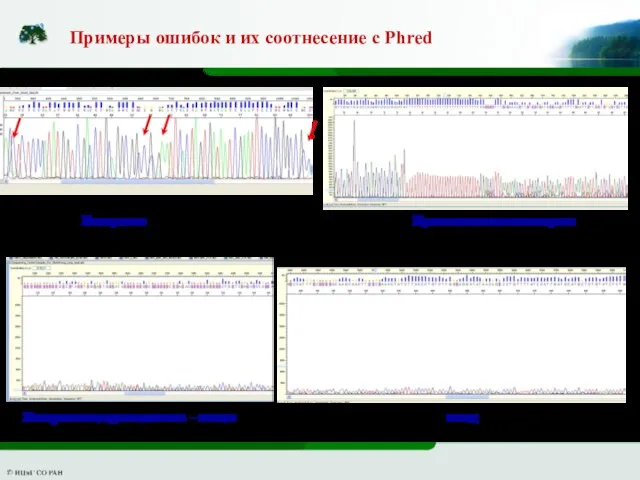
- 14. Примеры ошибок и их соотнесение с Phred Компрессия Проскальзывание полимеразы Химерная последовательность - начало конец
- 15. Основную часть ДНК в эукариотических геномах составляют повторы. Это делает крайне затруднительным правильное ассемблирование одиночных последовательностей
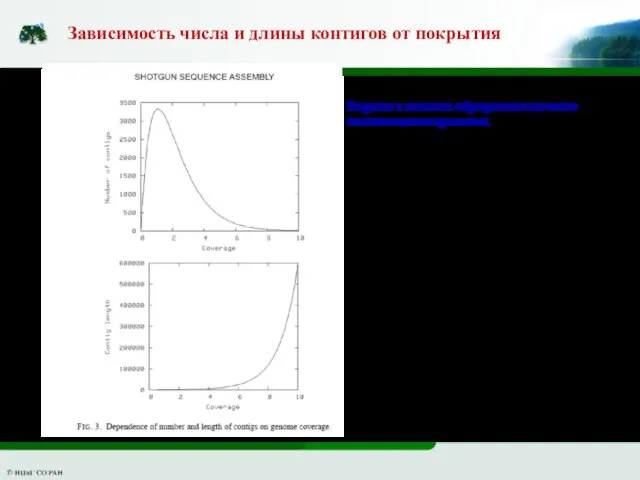
- 16. Зависимость числа и длины контигов от покрытия Разрывы в контигах образуются и по чисто статистическим причинам.
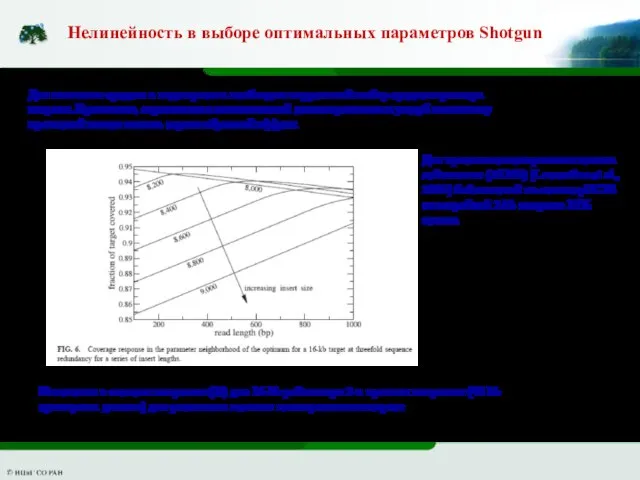
- 17. Нелинейность в выборе оптимальных параметров Shotgun Изменения в степени покрытия (%) для 16-kb района при 3-х
- 18. Этапы биоинформатического анализа Собственно сборка секвенированных последовательностей в геном. Структурная аннотация, включающая : идентификацию различных элементов
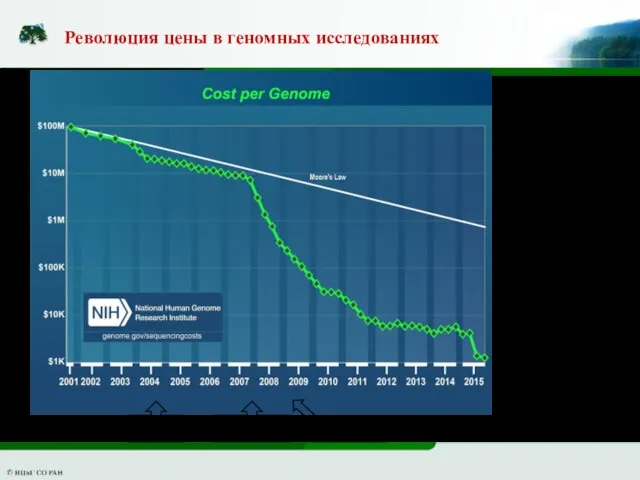
- 19. Революция цены в геномных исследованиях 454 GS20 1000 Геномов SOLiD 3.0 Illumina GA2
- 21. Скачать презентацию






 Как называются эти пары хромосом?
Как называются эти пары хромосом? Kharari_Sapiens
Kharari_Sapiens Австралопитеки
Австралопитеки Презентация на тему Организм и его свойства
Презентация на тему Организм и его свойства  Жизненный цикл клетки
Жизненный цикл клетки Нервная ткань
Нервная ткань Фотосинтез
Фотосинтез Клетка. Учение о тканях. Виды тканей
Клетка. Учение о тканях. Виды тканей Анатомия, физиология и гигиена человека Анатомия - наука о форме и строении живых организмов
Анатомия, физиология и гигиена человека Анатомия - наука о форме и строении живых организмов Тип Кольчатые черви. Класс Малощетинковые
Тип Кольчатые черви. Класс Малощетинковые Питание растений и грибов
Питание растений и грибов Обобщающее повторение
Обобщающее повторение Антропогенез. Гипотезы возникновения человека. Сходство и различия человека и животных. (Часть 3)
Антропогенез. Гипотезы возникновения человека. Сходство и различия человека и животных. (Часть 3) Процессы торможения в ЦНС
Процессы торможения в ЦНС Функциональная анатомия ствола головного мозга
Функциональная анатомия ствола головного мозга Подцарство простейшие. Амебовые, жгутиконосцы
Подцарство простейшие. Амебовые, жгутиконосцы Бионика и биоинженерия
Бионика и биоинженерия Презентация на тему Биология – наука о живой природе
Презентация на тему Биология – наука о живой природе Роль гормонів у фізичному, психічному і статевому розвитку
Роль гормонів у фізичному, психічному і статевому розвитку Гаметогенез, оплодотворение
Гаметогенез, оплодотворение Презентация по биологии Почвенная среда обитания
Презентация по биологии Почвенная среда обитания  Грибы-паразиты. Головня
Грибы-паразиты. Головня Фотосинтез
Фотосинтез Дослідження росту вегетативних органів рослин
Дослідження росту вегетативних органів рослин Міні – проект на тему: Опале листя. Це користь чи шкода?
Міні – проект на тему: Опале листя. Це користь чи шкода? Овогенез. Сперматогенез
Овогенез. Сперматогенез Витамины
Витамины Презентация на тему "Современное представление о механизмах и закономерностях эволюции" - скачать презентации по Биологии
Презентация на тему "Современное представление о механизмах и закономерностях эволюции" - скачать презентации по Биологии