Содержание
- 2. Модели молекулярной эволюции ДНК: 1 5 10 tagcaaaatg
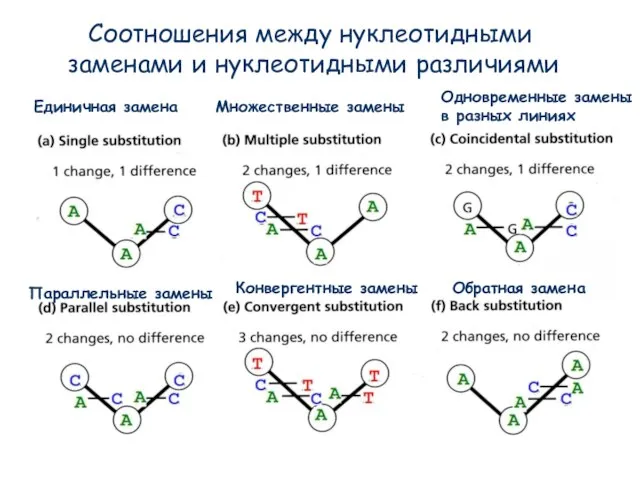
- 3. Соотношения между нуклеотидными заменами и нуклеотидными различиями Единичная замена Множественные замены Параллельные замены Конвергентные замены Обратная
- 4. Единичная замена Множественные замены Параллельные замены Конвергентные замены Обратная замена Одновременные замены в разных линиях Число
- 5. явные и скрытые генетические дистанции 1 10 20 30 Что такое генетическая дистанция? d = p,
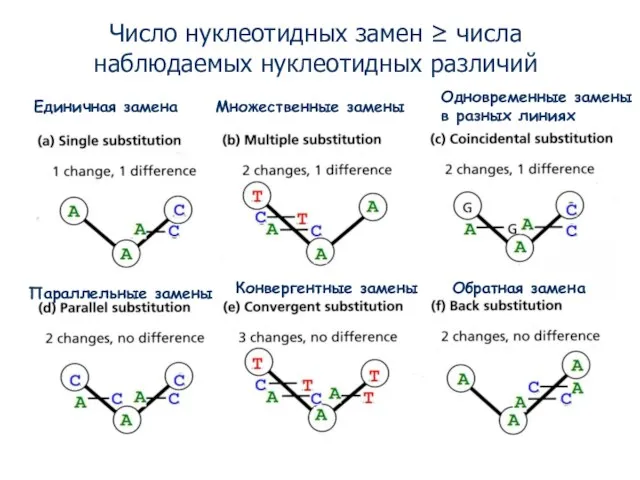
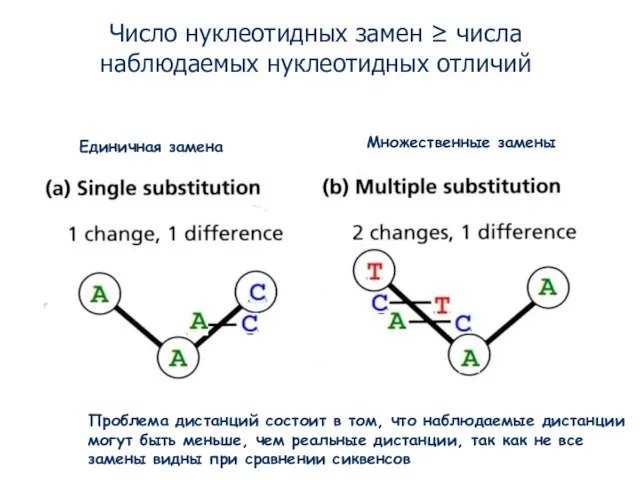
- 6. Единичная замена Множественные замены Число нуклеотидных замен ≥ числа наблюдаемых нуклеотидных отличий Проблема дистанций состоит в
- 7. Наблюдаемые генетические дистанции как правило меньше реальных эволюционных дистанций, так как есть скрытые замены Но как
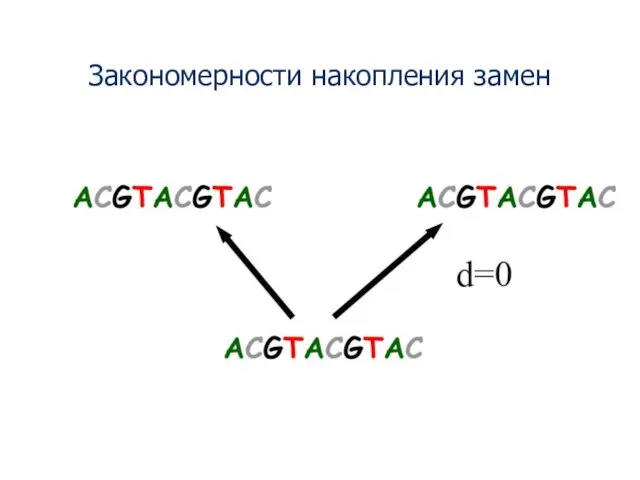
- 8. Закономерности накопления замен
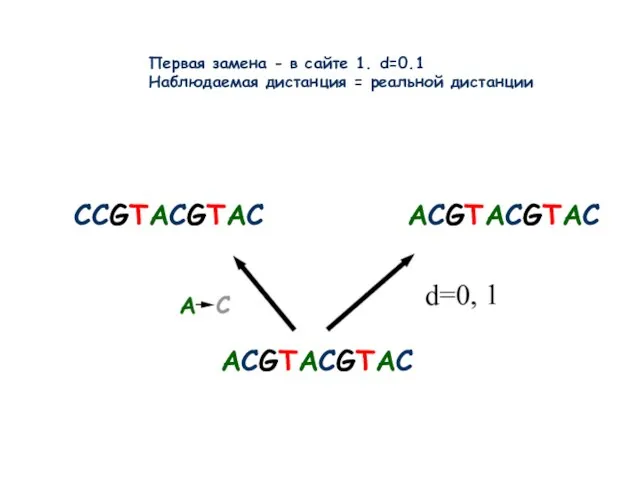
- 9. ACGTACGTAC CCGTACGTAC ACGTACGTAC Первая замена - в сайте 1. d=0.1 Наблюдаемая дистанция = реальной дистанции
- 10. CCGAACGTAC ACGTACGTAC Вторая замена – Имеется вероятность 0.1, что она будет повторной (т.е. тоже в сайте
- 11. CGTACGTACG ACGTACGTAC Третья замена имеет большую вероятность быть повторной, четвертая – еще большую, и. т.д. Т.е.
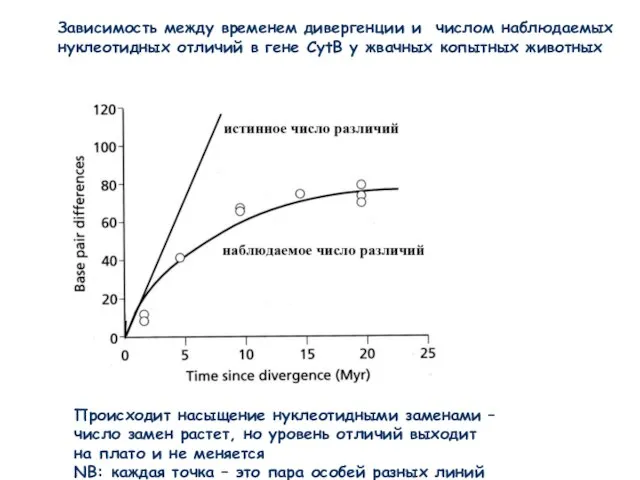
- 12. Зависимость между временем дивергенции и числом наблюдаемых нуклеотидных отличий в гене CytB у жвачных копытных животных
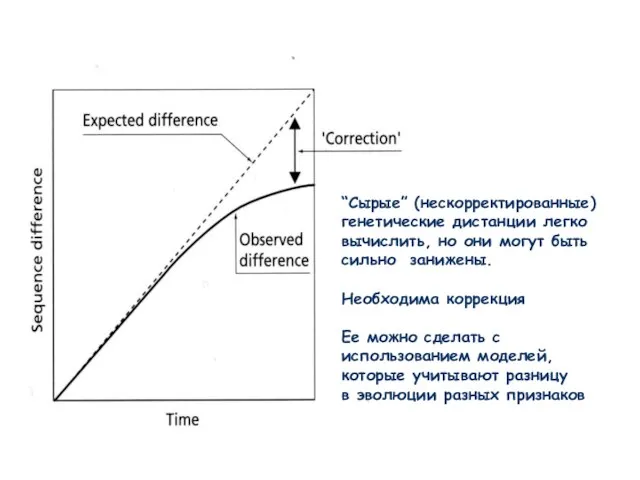
- 13. “Сырые” (нескорректированные) генетические дистанции легко вычислить, но они могут быть сильно занижены. Необходима коррекция Ее можно
- 14. Purines = adenin and guanine Pirimidines = cytosine and thymine
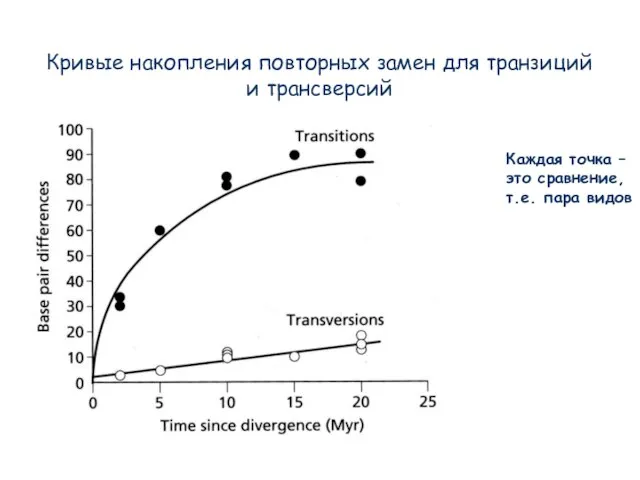
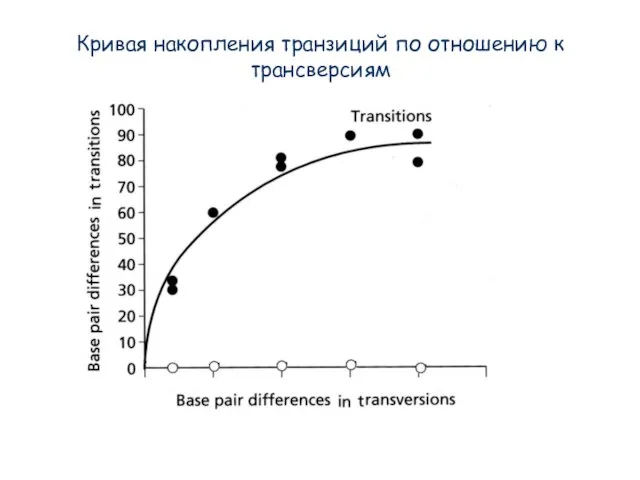
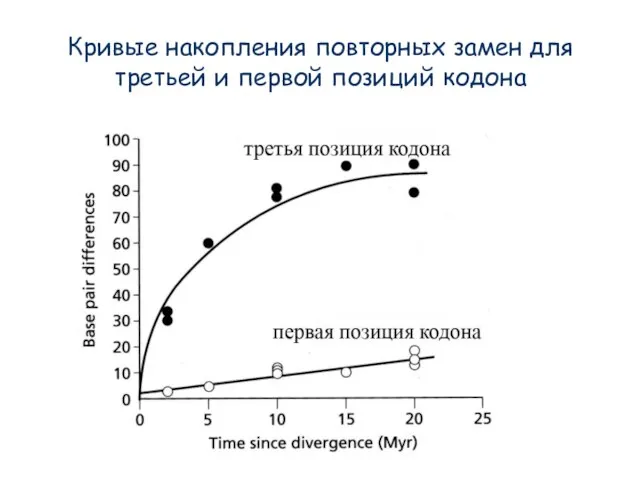
- 15. Кривые накопления повторных замен для транзиций и трансверсий Каждая точка – это сравнение, т.е. пара видов
- 16. Кривая накопления транзиций по отношению к трансверсиям
- 17. Генетический код Замена в первой позиции кодона ведет к замене аминокислоты Замена в третьей позиции кодона
- 18. Кривые накопления повторных замен для третьей и первой позиций кодона
- 19. Какие параметры можно извлечь из нуклеотидного выравнивания? 1 10 20 30
- 20. Какие параметры можно извлечь из нуклеотидного выравнивания? (1) Длина (2) Доля изменчивых сайтов (3) Доля инвариантных
- 21. Какие параметры можно извлечь из нуклеотидного выравнивания? Длина выравнивания Зависит от задач и технических возможностей 1
- 22. Какие параметры можно извлечь из нуклеотидного выравнивания? (2) Доля изменчивых сайтов (3) Доля инвариантных сайтов 1
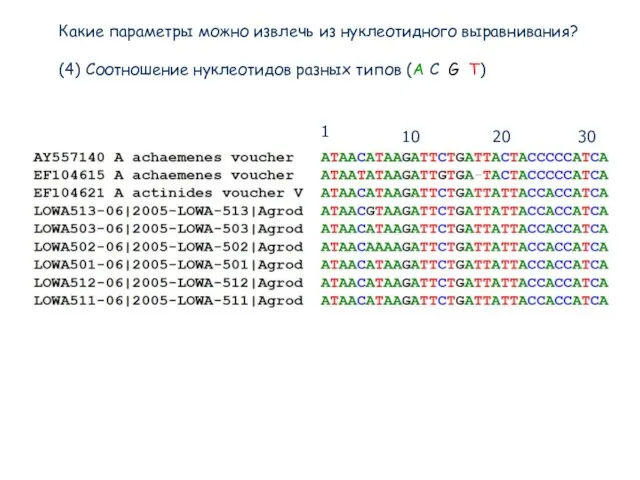
- 23. Какие параметры можно извлечь из нуклеотидного выравнивания? (4) Соотношение нуклеотидов разных типов (A C G T)
- 24. Какие параметры можно извлечь из нуклеотидного выравнивания? (5) Доля транзиций и трансверсий 1 10 20 30
- 25. Какие параметры можно извлечь из нуклеотидного выравнивания? (6) Доля нуклеотидных замен разных типов 1 10 20
- 26. Какие параметры можно извлечь из нуклеотидного выравнивания? (7) Все это (2-6) отдельно для каждой позиции кодона
- 27. Какие параметры можно извлечь из нуклеотидного выравнивания? (8) Доля синонимичных и несинонимичных замен (9) Доля синонимичных
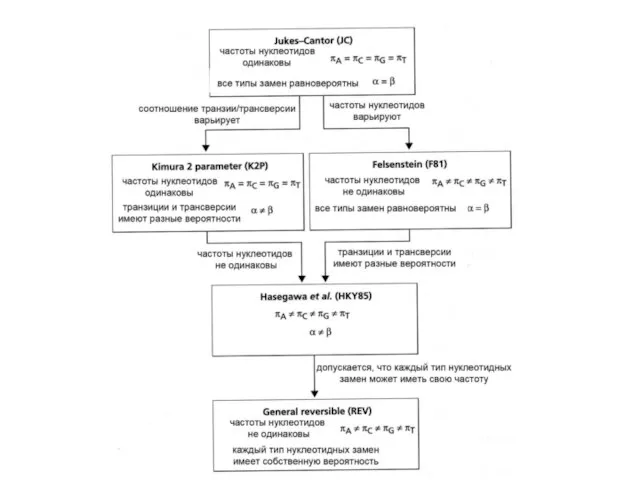
- 28. Предпосылки 1) нуклеотидные замены одного типа равновероятны в разных частях одного гена 2) нуклеотидные замены обратимы
- 29. Если вероятности нуклеотидных замен (p) и частоты нуклеотидов (f) константны во времени, то суммарная эволюционная дистанция
- 30. Если вероятности нуклеотидных замен (p) и частоты нуклеотидов (f) константны во времени, то суммарная эволюционная дистанция
- 31. частоты нуклеотидов и доли замен разного типа берутся непосредственно из выравнивания
- 32. JC Вероятности всех замен одинаковы, частоты нуклеотидов равны D= D = -(3/4)ln(1-4/3 где p – это
- 33. Двухпараметрическая модель Кимуры K2P Вероятности транзиций и трансверсий разные, частоты нуклеотидов равны α – транзиция β
- 34. F81 Вероятности всех замен одинаковы, но частоты нуклеотидов разные
- 35. HKY model Вероятности транзиций и трансверсий разные, частоты нуклеотидов разные
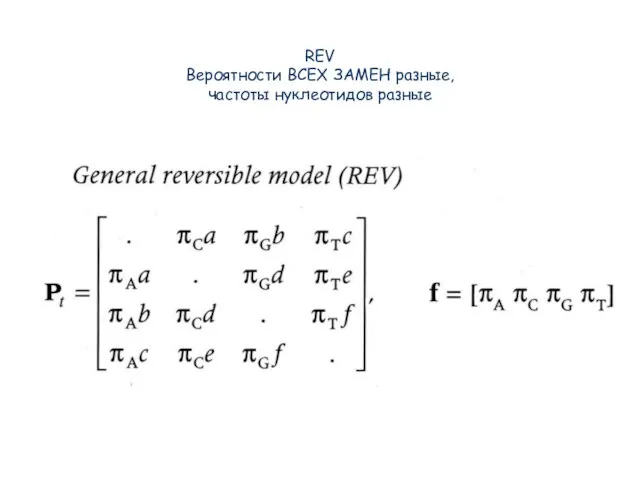
- 36. REV Вероятности ВСЕХ ЗАМЕН разные, частоты нуклеотидов разные
- 38. Чем хороши и чем плохи сложные и простые модели?
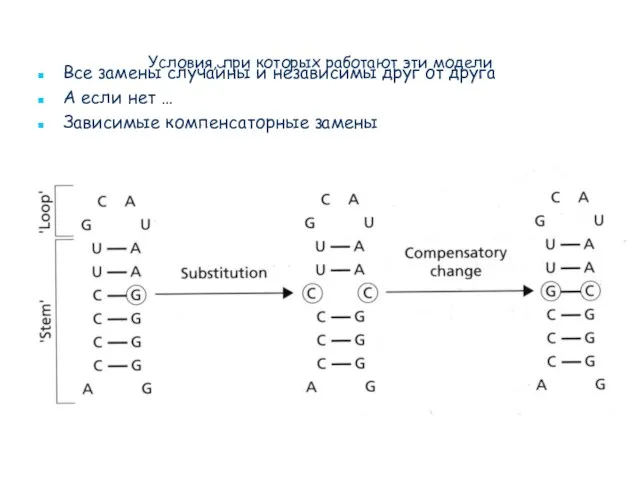
- 39. Условия, при которых работают эти модели Это стохастические модели, которые предполагают, что все замены случайны и
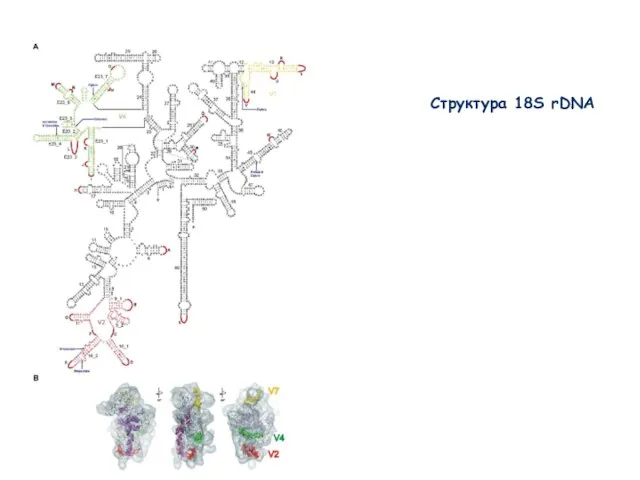
- 40. Структура 18S rDNA
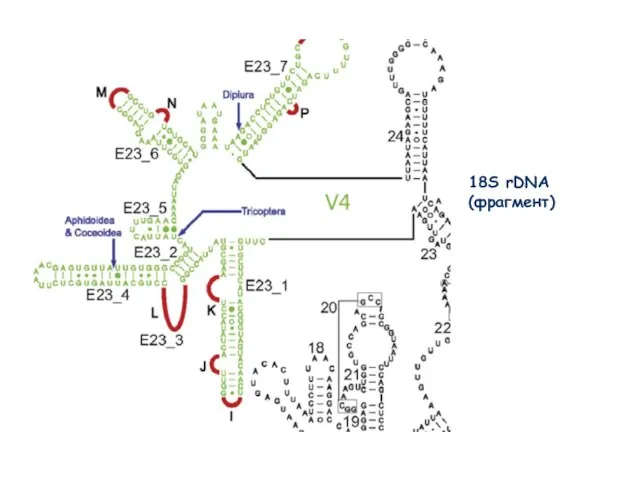
- 41. 18S rDNA (фрагмент) 18S rDNA (фрагмент)
- 42. Условия, при которых работают эти модели Все замены случайны и независимы друг от друга А если
- 43. Общие принципы построения филогений 1) Анализ признаков, 2) выбор оптимальной модели эволюции признака, 3) выбор методов
- 44. Подходы к выявлению филогений традиционный (Геккелевский, эмпирико-интуитивный) традиционная кладистика (Hennig, 1950, 1966) фенетика метод максимальной парсимонии
- 45. Традиционный (эмпирико-интуитивный) метод выведения филогений Строго научный и, как правило, очень качественный анализ признаков сочетается с
- 46. По Геккелю филогенетика – наука о путях, закономерностях и причинах исторического развития организмов Ernst Haeckel (1834-1919)
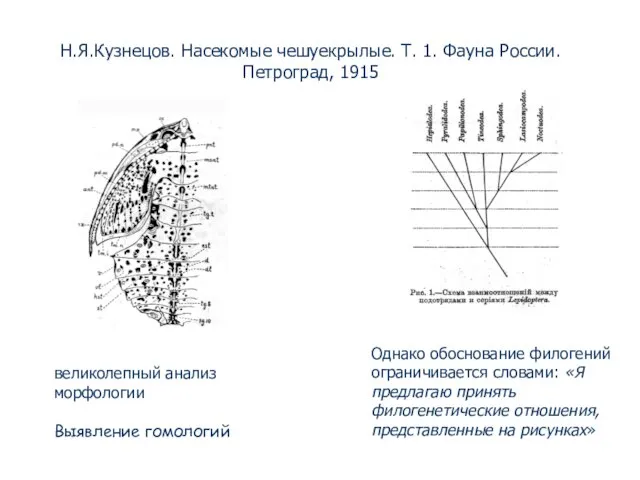
- 47. Н.Я.Кузнецов. Насекомые чешуекрылые. Т. 1. Фауна России. Петроград, 1915 Однако обоснование филогений ограничивается словами: «Я предлагаю
- 48. “Недавно в лабораторию [Моргана]пришла почта с произведениями Северцова с многочисленными филогенетическими древесами, на которые я указал
- 49. Традиционная кладистика (Hennig, 1950, 1966) Хенниг предложил строго научные принципы перехода от анализа признаков к реконструкции
- 50. Признаки Негомологичные (гомоплазии) Гомологичные Плезиоморфии Апоморфии Синапоморфии
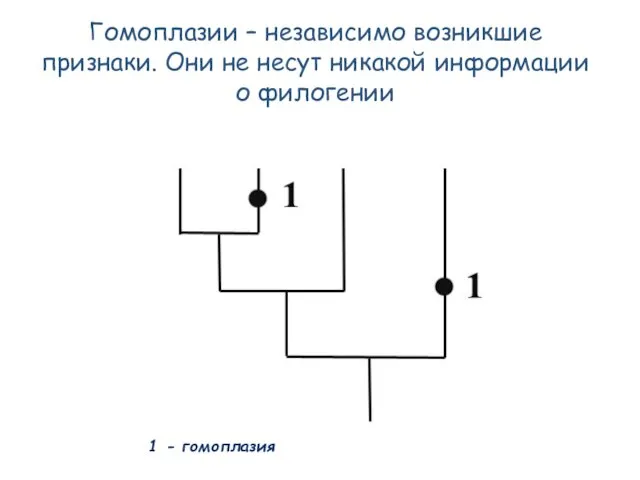
- 51. Гомоплазии – независимо возникшие признаки. Они не несут никакой информации о филогении 1 - гомоплазия
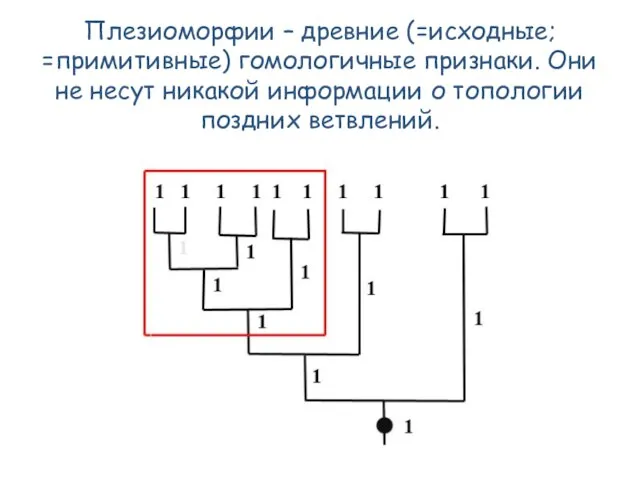
- 52. Плезиоморфии – древние (=исходные; =примитивные) гомологичные признаки. Они не несут никакой информации о топологии поздних ветвлений.
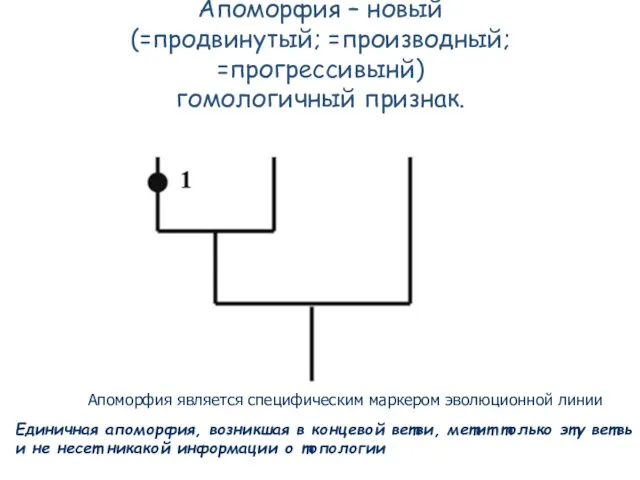
- 53. Апоморфия – новый (=продвинутый; =производный; =прогрессивынй) гомологичный признак. Единичная апоморфия, возникшая в концевой ветви, метит только
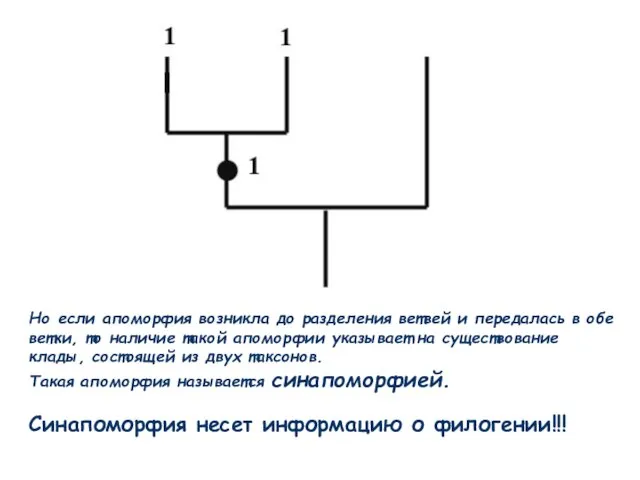
- 54. Но если апоморфия возникла до разделения ветвей и передалась в обе ветки, то наличие такой апоморфии
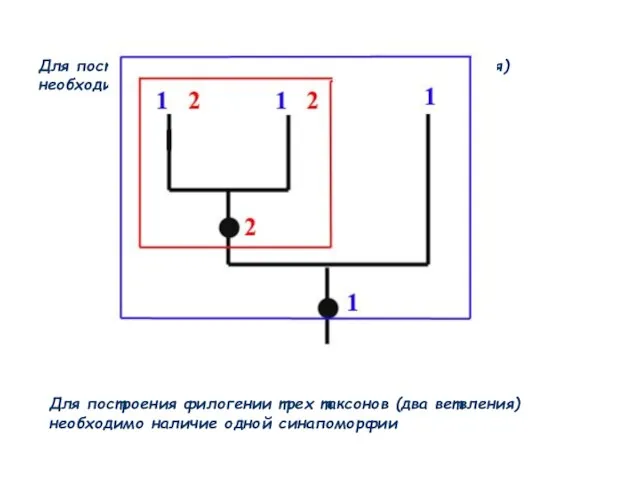
- 55. Для построения филогении трех таксонов (два ветвления) необходимо наличие одной синапоморфии Для построения филогении трех таксонов
- 56. В общем виде для полного разрешения филогении, включающей n ветвлений, необходимо и достаточно n-1 синапоморфий (по
- 57. Филогения строится как система соподчиненных (вложенных одна в другую) клад (монофилетических групп), каждая из которых выявляется
- 58. Модель эволюции в кладистике по Геннигу Топология - строгая дихотомия Процесс – накопление синапоморфий.
- 59. Алгоритм анализа Одна истинная синапоморфия может разрешить узел ветвления филогенетического дерева Выявление филогении – многоступенчатый процесс
- 60. Построение молекулярного дерева с использованием кладистики по Хеннигу 1 AAGT 2 AAGT 3 ACGT
- 61. Построение молекулярного дерева таксонов 1-4 с использованием кладистики по Хеннигу 1 AAGTT 2 AAGTT 3 ACGTT
- 62. Состояние ACGTA плезиоморфно 1 AAGTT 2 AAGTT 3 ACGTT 4 ACGTA 5 ACGTA 6 ACGTA 7
- 63. A во второй позиции – синапоморфия 1 + 2 1 AAGTT 2 AAGTT 3 ACGTT 4
- 64. T в пятой позиции – синапоморфия 1 + 2 +3 1 AAGTT 2 AAGTT 3 ACGTT
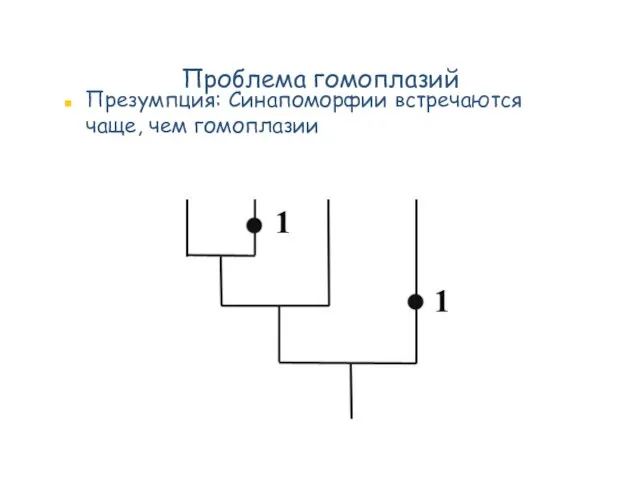
- 65. Проблема гомоплазий Презумпция: Синапоморфии встречаются чаще, чем гомоплазии
- 66. Конфликт между потенциальными синапоморфиями 1 AAGTT 2 AACTT 3 ACCTT 4 ACGTT
- 67. Принципы традиционной кладистики Если возникает конфликт между потенциальными синапоморфиями, то основной путь его решения – переисследование
- 68. Другие проблемы генниговской кладистики: “Надежных” синапоморфий может быть мало, недостаточно для того, что разрешить все узлы
- 69. Проблемы традиционной кладистики “Надежных” синапоморфий может быть мало, недостаточно для того, что разрешить все узлы ветвления
- 70. Проблемы традиционной кладистики “Надежных” синапоморфий может быть мало, недостаточно для того, что разрешить все узлы ветвления
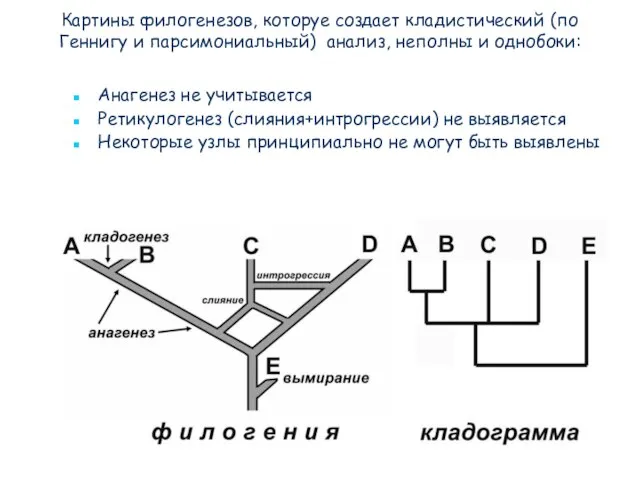
- 71. Картины филогенезов, которуе создает кладистический (по Геннигу и парсимониальный) анализ, неполны и однобоки: Анагенез не учитывается
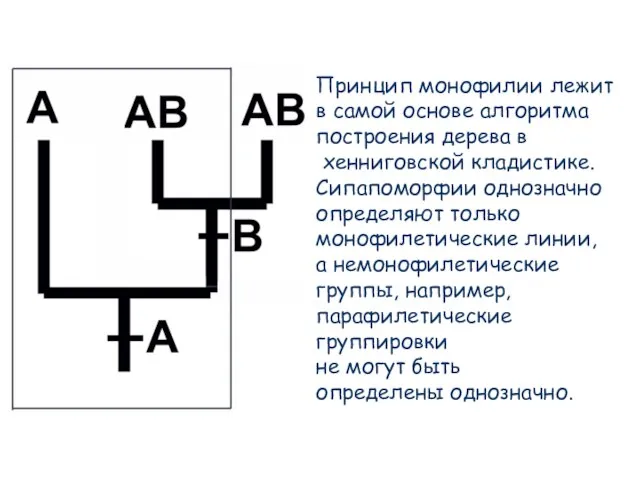
- 72. Принцип монофилии лежит в самой основе алгоритма построения дерева в хенниговской кладистике. Сипапоморфии однозначно определяют только
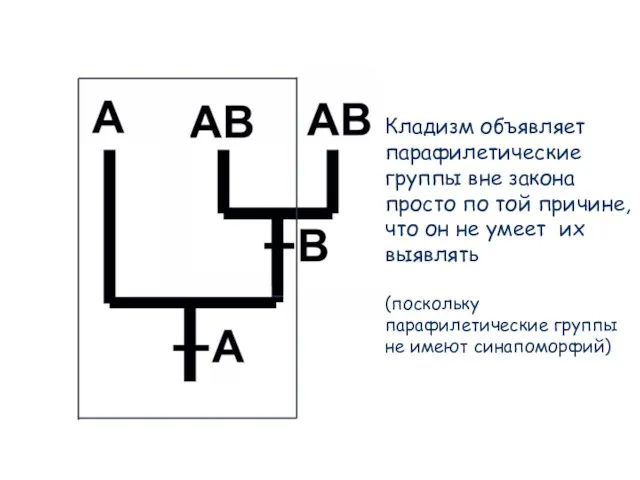
- 73. Кладизм объявляет парафилетические группы вне закона просто по той причине, что он не умеет их выявлять
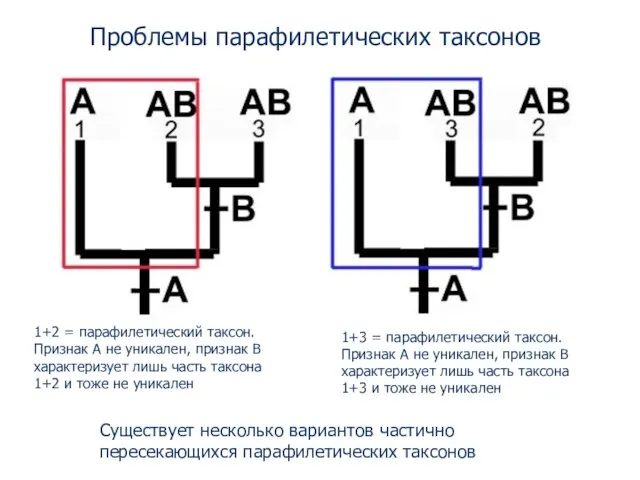
- 74. Проблемы парафилетических таксонов 1+2 = парафилетический таксон. Признак A не уникален, признак B характеризует лишь часть
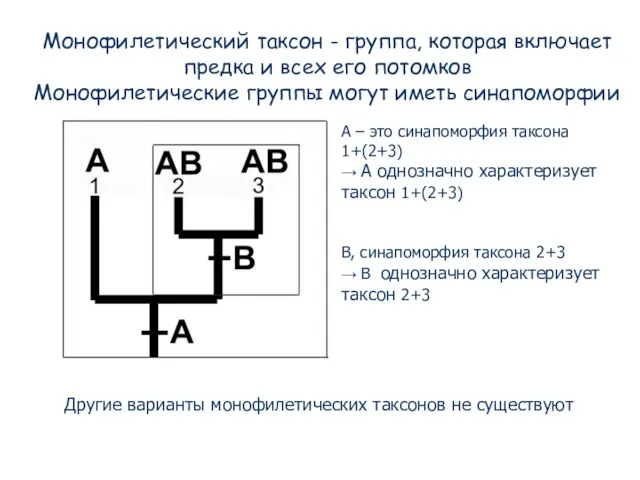
- 75. Монофилетический таксон - группа, которая включает предка и всех его потомков Монофилетические группы могут иметь синапоморфии
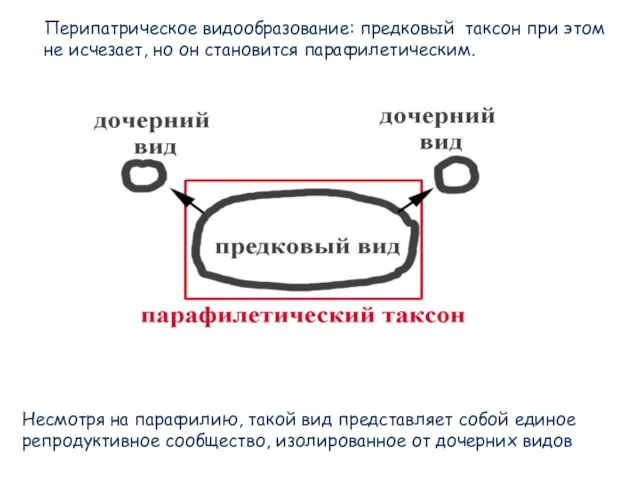
- 76. Перипатрическое видообразование: предковый таксон при этом не исчезает, но он становится парафилетическим. Несмотря на парафилию, такой
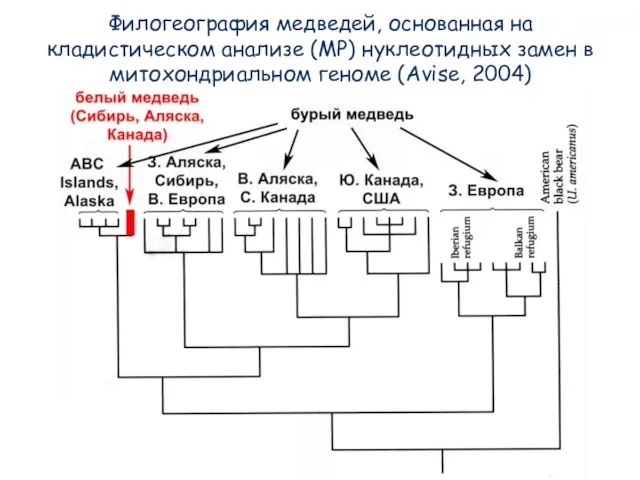
- 77. Филогеография медведей, основанная на кладистическом анализе (MP) нуклеотидных замен в митохондриальном геноме (Avise, 2004)
- 78. Кладистика по Геннигу остается рабочим инструментом филогенетики!
- 79. Фенетика В кладистике процедура выявления гомологичных признаков (дифференциация от гомоплазий) не формализована. Это может быть причиной
- 80. Фенетика Отказ от доминирования принципа гомологии (в фенетике все признаки имеют равный вес) Степень родства =
- 81. Фенетика Кластерный анализ (выявление группировок по степени их сходства). Иерархии таких группировок можно интерпретировать в качестве
- 82. Фенетика Пример научной, но неправильной (неадекватной) методологии Научность – строгое следование принципам научной логики, избегание субъективизма
- 83. Традиционная и нумерическая кладистика Увеличение числа признаков приводит к противоречиям между предполагаемыми синапоморфиями, которые свидетельствуют о
- 84. Если возникает конфликт между потенциальными синапоморфиями, то есть два пути его решения: 1)переисследование материала, поиск и
- 85. Если возникает конфликт между потенциальными синапоморфиями, то есть два пути его решения: 1)переисследование материала, поиск и
- 86. Нумерическая кладистика и метод максимальной парсимонии При наличии противоречий между “синапоморфиями” возможны разные варианты филогении Как
- 87. Метод максимальной парсимонии (наибольшей экономии)
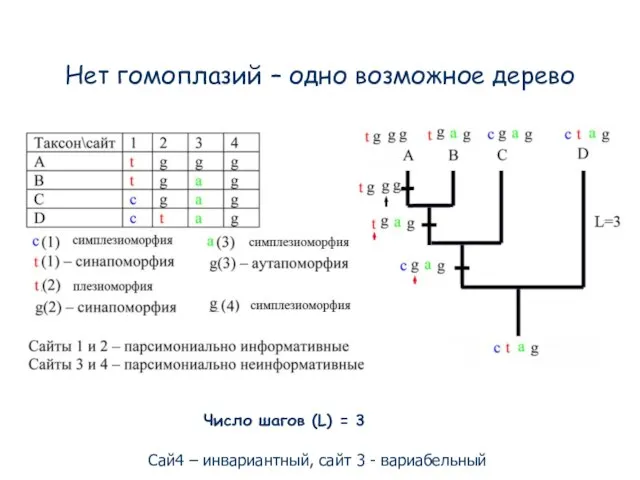
- 88. Нет гомоплазий – одно возможное дерево Число шагов (L) = 3 Сай4 – инвариантный, сайт 3
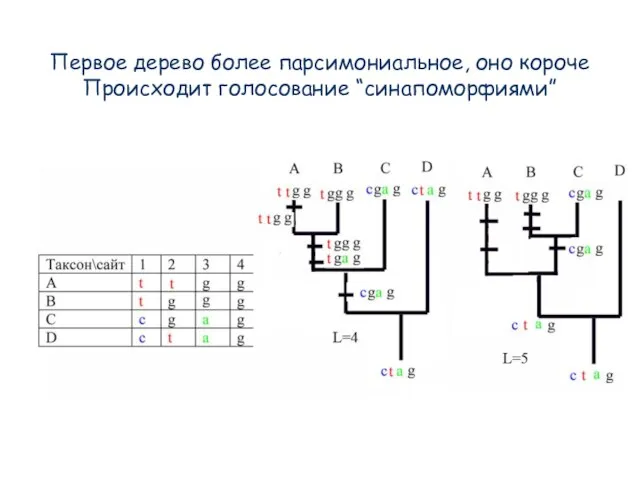
- 89. Первое дерево более парсимониальное, оно короче Происходит голосование “синапоморфиями”
- 90. в реальности у нас исходно нет ни топологии дерева, ни распределения признаков по нему, ни анцестрального
- 91. Шаг 1: выявление признаков и их состояний Признак – цвет глаз Состояния – коричневый, голубой, зеленый
- 92. Признак – цвет глаз Состояния – коричневый (0), голубой (1), зеленый (2) Признак – группа крови
- 93. Шаг 3: Составление матрицы признаков
- 94. Бинарная матрица Матрица множественных состояний
- 95. Нуклеотидное (или аминокислотное) выравнивание – это уже готовая матрица признаков 4 состояния – A C G
- 96. Шаг 4: выбор модели эволюции Модель Камина-Сокола (Camin- Sokal parsimony): анцестральное состояние известно, тогда 0 —›
- 97. Модель Долло (Dollo parsimony) (основана на принципе необратимости эволюции) - допускаются изменения признака в любую сторону,
- 98. Модель Фитча-Вагнера (Fitch-Wagner parsimony) – симметричная модель 0 дерево неукорененное!!!
- 99. Модель Фитча-Вагнера (Fitch-Wagner parsimony) для множественных состояний признака 0 0 0 1 1 2 дерево неукорененное!!!
- 101. Скачать презентацию




























![“Недавно в лабораторию [Моргана]пришла почта с произведениями Северцова с многочисленными филогенетическими](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/518960/slide-47.jpg)




































 Презентация на тему Хрящевые рыбы
Презентация на тему Хрящевые рыбы  Моя собака породы Бишон Фризе, 2 класс
Моя собака породы Бишон Фризе, 2 класс Авторы: Мартюкова Наталья – 5 класс Чухрова Юлия – 4 класс
Авторы: Мартюкова Наталья – 5 класс Чухрова Юлия – 4 класс Презентация на тему "Ухо - это орган слуха" - скачать презентации по Биологии
Презентация на тему "Ухо - это орган слуха" - скачать презентации по Биологии Позвоночные животные
Позвоночные животные Кровь и остальные компоненты внутренней среды организма. Автор учитель биологии МОУ «Гимназия № 23» г. Троицк Че
Кровь и остальные компоненты внутренней среды организма. Автор учитель биологии МОУ «Гимназия № 23» г. Троицк Че Клеточное строение организма
Клеточное строение организма Типы взаимоотношений между организмами. Никитенко О.Д.
Типы взаимоотношений между организмами. Никитенко О.Д. Среды жизни организмов.
Среды жизни организмов. Презентация на тему "Отряды насекомых Таракановые, Прямокрылые, Уховёртки, Подёнки" - скачать бесплатно презентации по Биолог
Презентация на тему "Отряды насекомых Таракановые, Прямокрылые, Уховёртки, Подёнки" - скачать бесплатно презентации по Биолог Среда обитания и экологические факторы. Урок по биологии , 6 класс Учитель биологии: Габдрашитова Гузалия Сагитовна.
Среда обитания и экологические факторы. Урок по биологии , 6 класс Учитель биологии: Габдрашитова Гузалия Сагитовна. самые - самые растения Автор презентации: Данилова Ольга Леонидовна преподаватель специальных дисциплин ГБОУ НПО ПЛ №114 МО г. О
самые - самые растения Автор презентации: Данилова Ольга Леонидовна преподаватель специальных дисциплин ГБОУ НПО ПЛ №114 МО г. О обмін речовин та енергії в організмі людини
обмін речовин та енергії в організмі людини  Гимназия №6 г. Гродно
Гимназия №6 г. Гродно Тема урока: Строение и функции головного мозга.
Тема урока: Строение и функции головного мозга. Закономерности скрещивания. Моногибридное скрещивание
Закономерности скрещивания. Моногибридное скрещивание Постэмбриональный период развития организмов
Постэмбриональный период развития организмов Презентация на тему "состав табачного дыма и его действие на организм" - скачать презентации по Биологии
Презентация на тему "состав табачного дыма и его действие на организм" - скачать презентации по Биологии Внутреннее строение птицы Презентация к уроку биологии Отряскиной Т.А.
Внутреннее строение птицы Презентация к уроку биологии Отряскиной Т.А.  Биотехнология
Биотехнология Эволюция растительного мира на Земле. 6 класс.
Эволюция растительного мира на Земле. 6 класс. Жаворонок
Жаворонок Таежные леса России
Таежные леса России Презентация на тему "Альфред Нобель" - скачать презентации по Биологии
Презентация на тему "Альфред Нобель" - скачать презентации по Биологии Құс жұмыртқасының құрлысы
Құс жұмыртқасының құрлысы Устойчивость растений к стрессорам химической природы. (Лекция 2-3)
Устойчивость растений к стрессорам химической природы. (Лекция 2-3) Тема уроку: Вітаміни. Задачі: сформулювати загальне уявлення про вітаміни, дати поняття про авітаміноз, гіпо- та гіперв
Тема уроку: Вітаміни. Задачі: сформулювати загальне уявлення про вітаміни, дати поняття про авітаміноз, гіпо- та гіперв ТИП ПЛОСКИЕ ЧЕРВИ
ТИП ПЛОСКИЕ ЧЕРВИ