Содержание
- 2. Родини длРНК вірусів Reoviridae – велика родина, мають 10-12 сегменів, інфікує хребетних, безхребетних, рослини і гриби
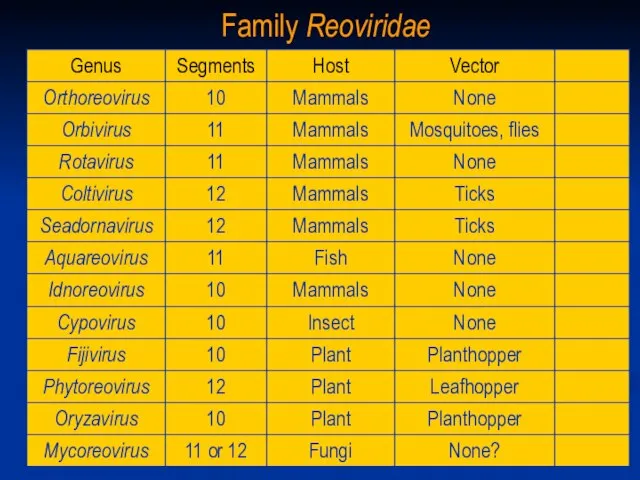
- 3. Family Reoviridae
- 4. Геном Reovirus 10-12 сегментів dsRNA Пакується 1 копія в віріон Загальний розмір геному 22-28 kb (0.8-4.5
- 5. Консервативні кінцеві послідовності сегментів геномів роду Оrbivirus (+ ланцюг) BTV5 5-GUUAAA............................ACUUAC-3 EHDV 5 -GUUAAA..........................A/GCUUAC-3 AHSV 5
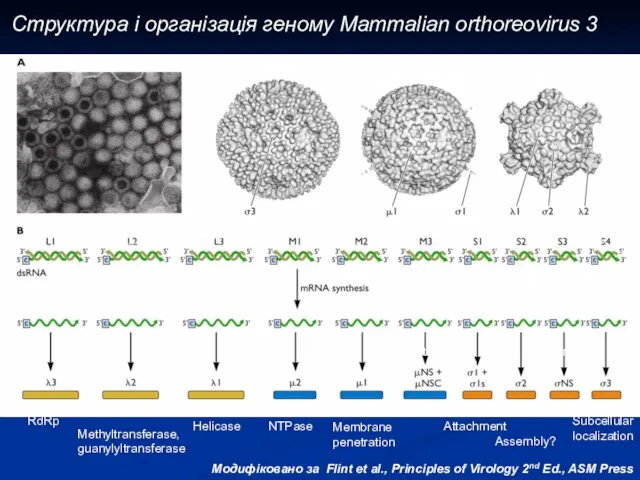
- 6. Модифіковано за Flint et al., Principles of Virology 2nd Ed., ASM Press Структура і організація геному
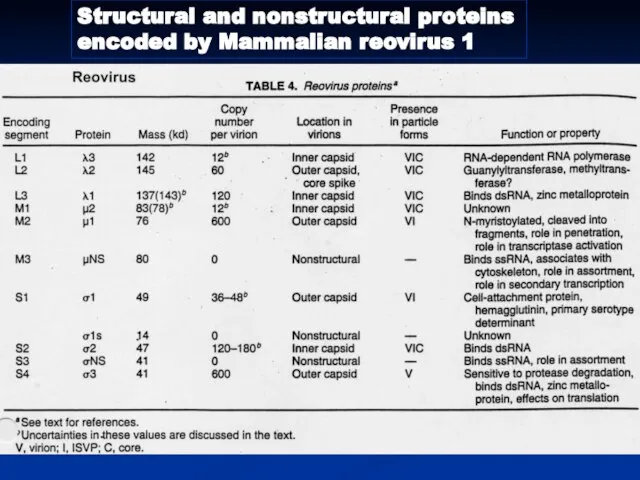
- 7. Structural and nonstructural proteins encoded by Mammalian reovirus 1
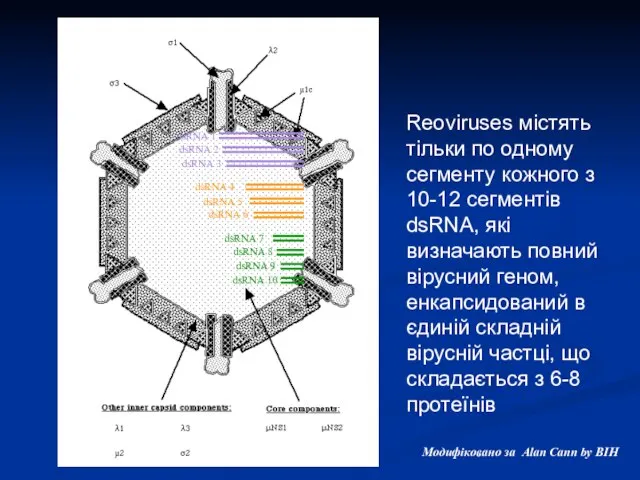
- 8. Модифіковано за Alan Cann by BIH dsRNA 1 dsRNA 2 dsRNA 3 dsRNA 4 dsRNA 5
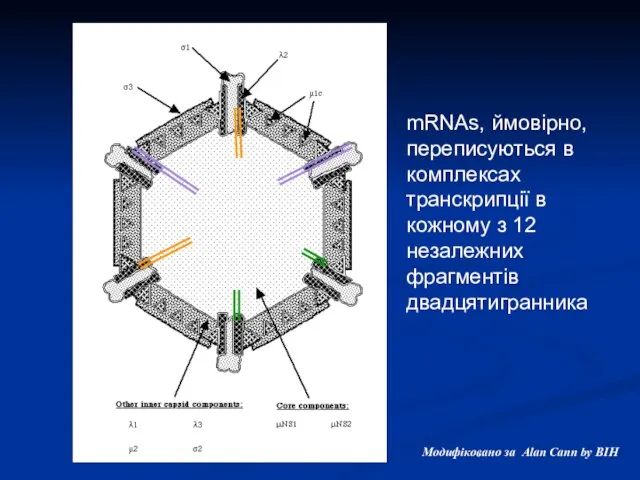
- 9. mRNAs, ймовірно, переписуються в комплексах транскрипції в кожному з 12 незалежних фрагментів двадцятигранника Модифіковано за Alan
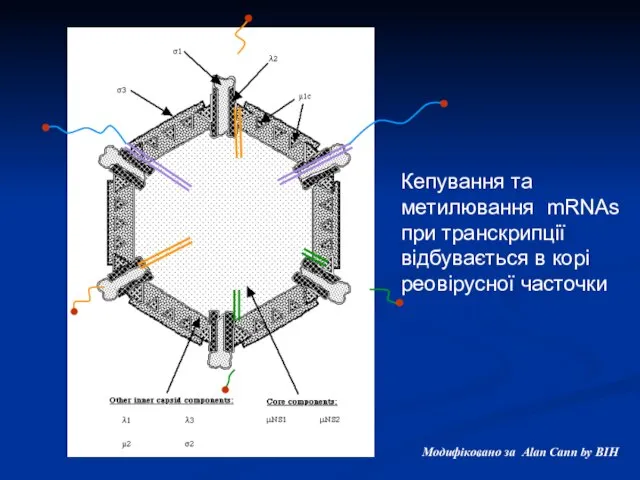
- 10. Кепування та метилювання mRNAs при транскрипції відбувається в корі реовірусної часточки Модифіковано за Alan Cann by
- 11. Транскрипція/Реплікація: RNA транскрибується консервативно: Використовується тільки (‑)смисловий ланцюг; В результаті синтезується (+)смислова mRNAs, Кепувапння відбувається в
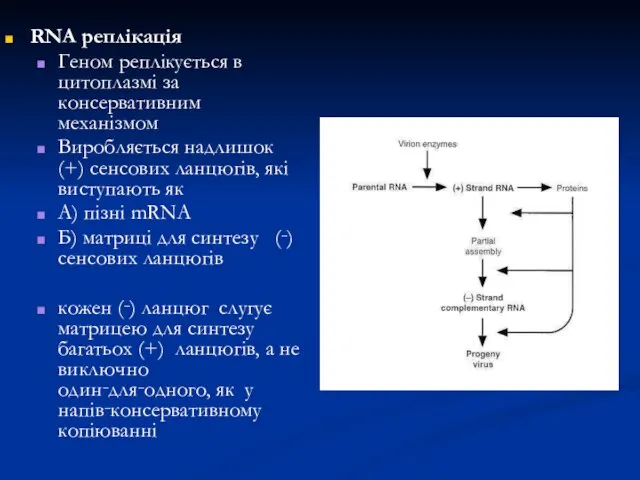
- 12. RNA реплікація Геном реплікується в цитоплазмі за консервативним механізмом Виробляється надлишок (+) сенсових ланцюгів, які виступають
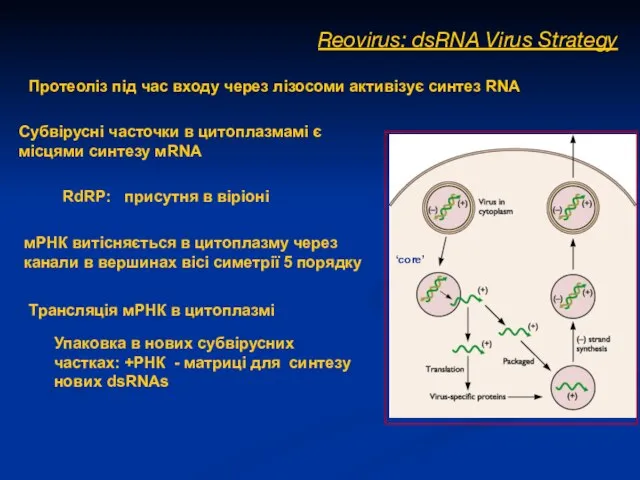
- 13. Reovirus: dsRNA Virus Strategy Субвірусні часточки в цитоплазмамі є місцями синтезу мRNA мРНК витісняється в цитоплазму
- 14. Totiviridae віруси - “killer” фунгі Members of the family Totiviridae Не викликає інфекції в заражених клітинах
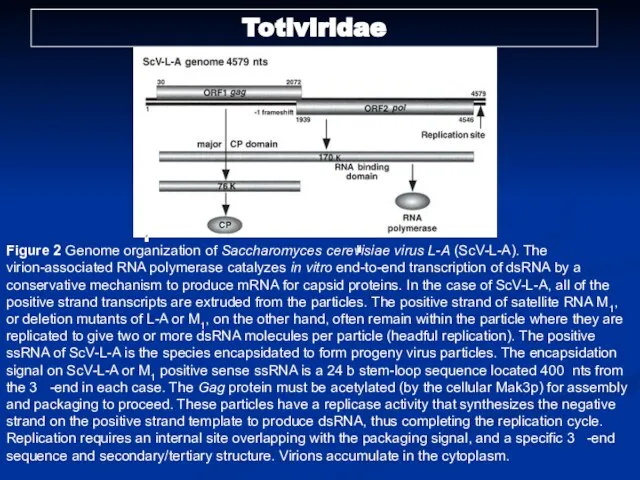
- 15. Figure 2 Genome organization of Saccharomyces cerevisiae virus L-A (ScV-L-A). The virion-associated RNA polymerase catalyzes in
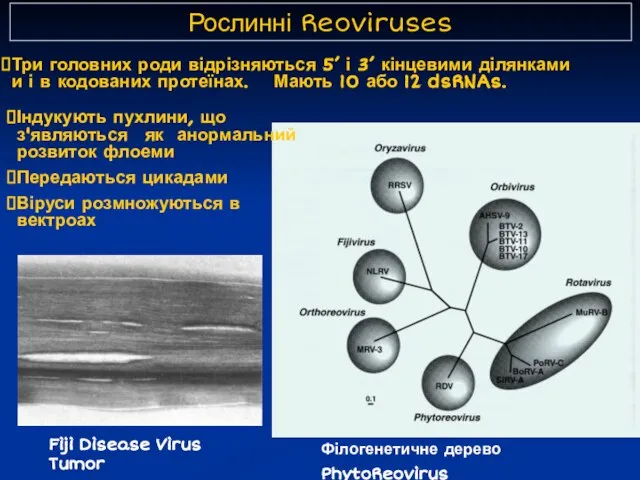
- 16. Рослинні Reoviruses Три головних роди відрізняються 5’ і 3’ кінцевими ділянками и і в кодованих протеїнах.
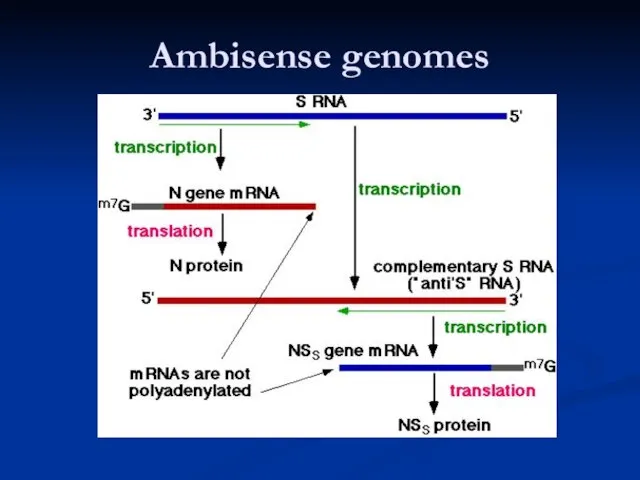
- 17. Ambisense genomes
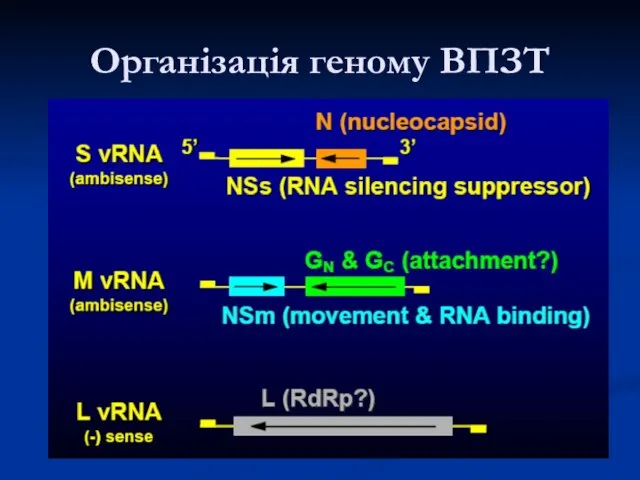
- 18. Організація геному ВПЗТ
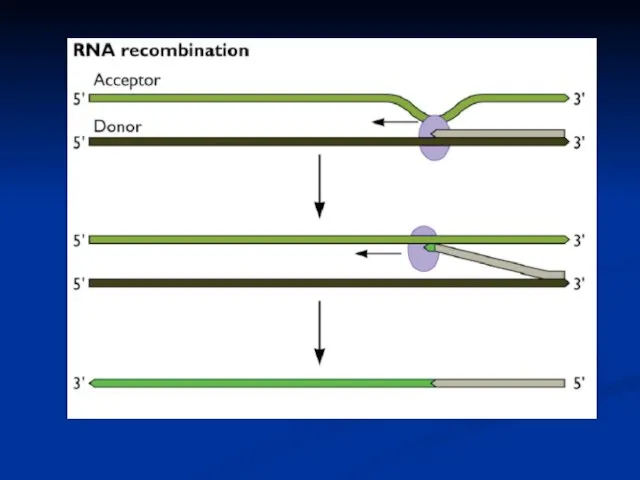
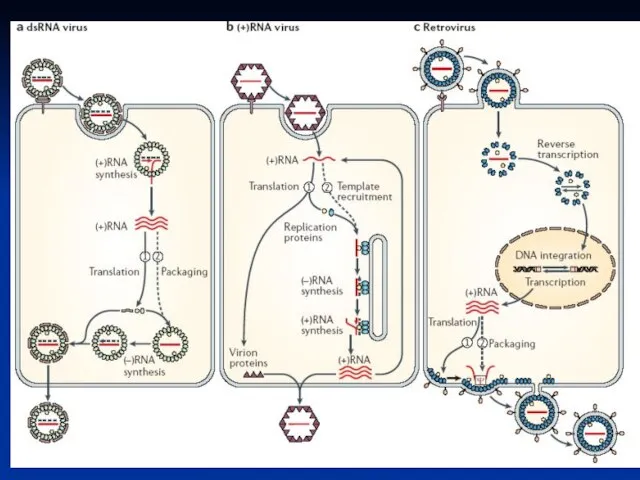
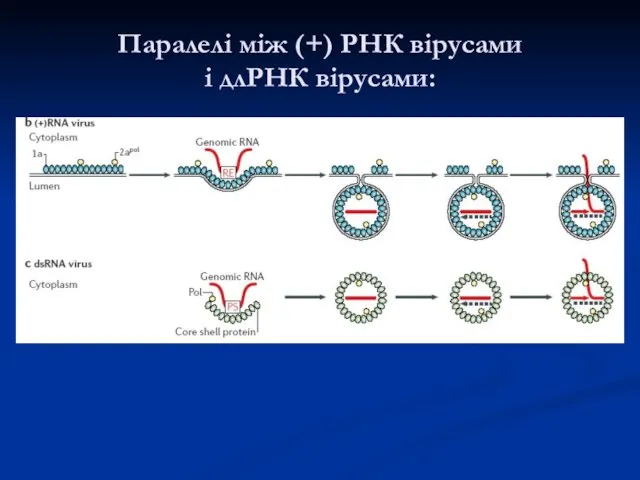
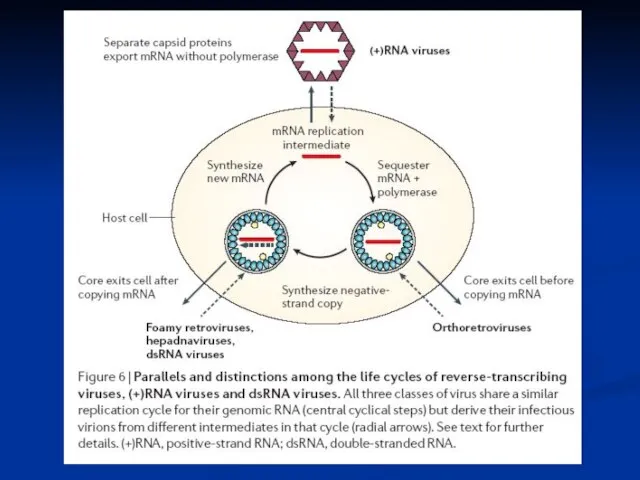
- 20. Подібності реплікативного процесу у (+) РНК вірусів, длРНК вірусів і зворотньо-транскрибуючих вірусів
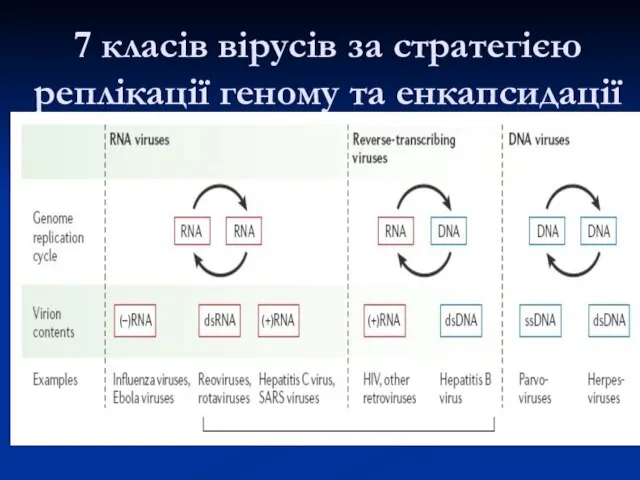
- 21. 7 класів вірусів за стратегією реплікації геному та енкапсидації
- 23. Фундаментальні зв'язки між класами Виявлено паралелі у процесі реплікації геному між: 1) (+) РНК вірусів, 2)
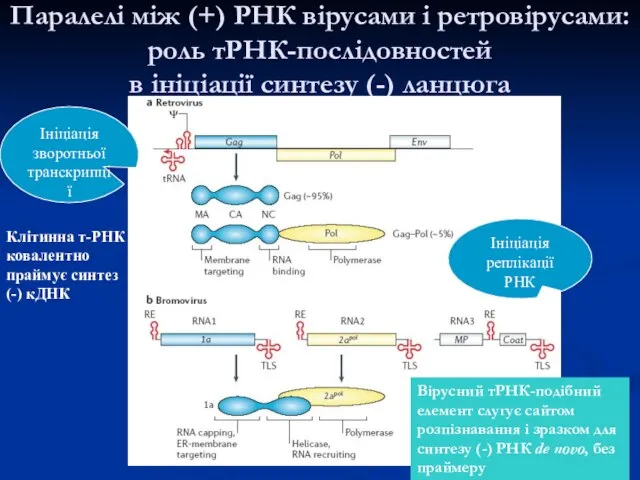
- 24. Паралелі між (+) РНК вірусами і ретровірусами: роль тРНК-послідовностей в ініціації синтезу (-) ланцюга Ініціація реплікації
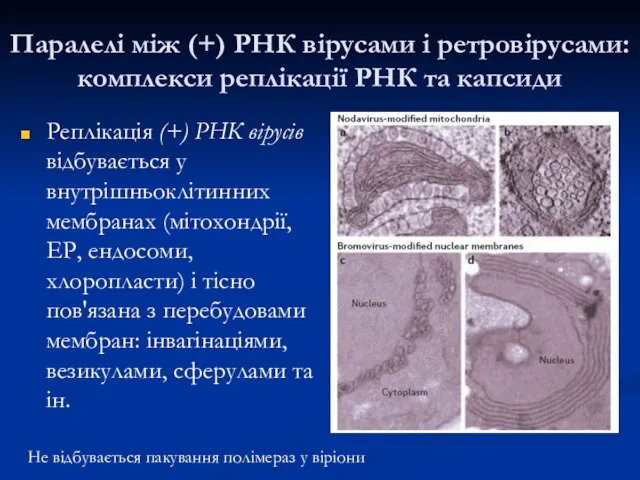
- 25. Паралелі між (+) РНК вірусами і ретровірусами: комплекси реплікації РНК та капсиди Реплікація (+) РНК вірусів
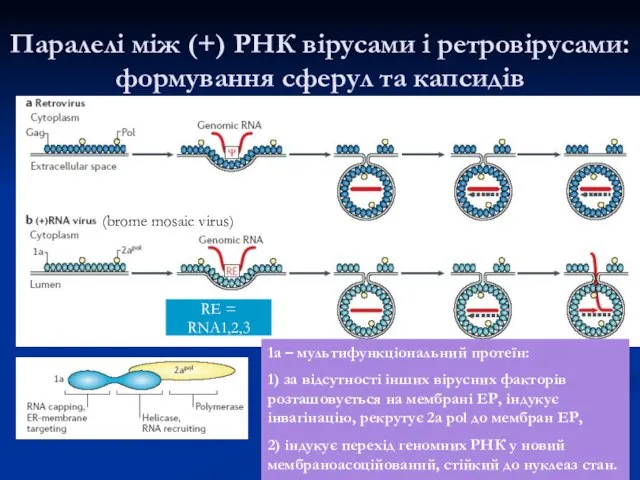
- 26. Паралелі між (+) РНК вірусами і ретровірусами: формування сферул та капсидів (brome mosaic virus) 1а –
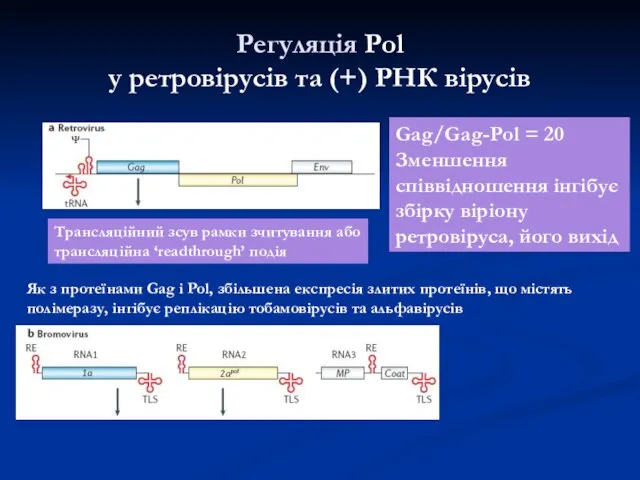
- 27. Регуляція Pol у ретровірусів та (+) РНК вірусів Gag/Gag-Pol = 20 Зменшення співвідношення інгібує збірку віріону
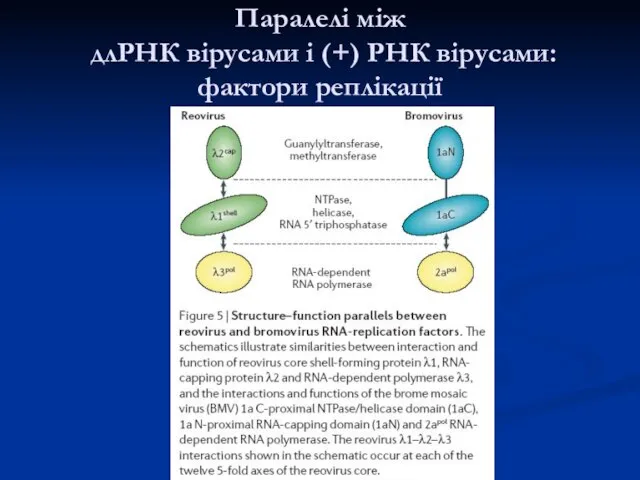
- 28. Паралелі між длРНК вірусами і (+) РНК вірусами: фактори реплікації
- 29. Паралелі між (+) РНК вірусами і длРНК вірусами:
- 31. НЕКАНОНІЧНІ ВІРУСИ
- 32. Сателіти Геном приблизно 500-2000 нуклеотидів з одноланцюгової РНК Геном сателіту не схожий за своїми нуклеотидними послідовностями
- 33. Вірус гепатиту дельта (HDV) Вірус гепатиту дельта – унікальна молекула РНК,, яка схожа на віроїд, однак
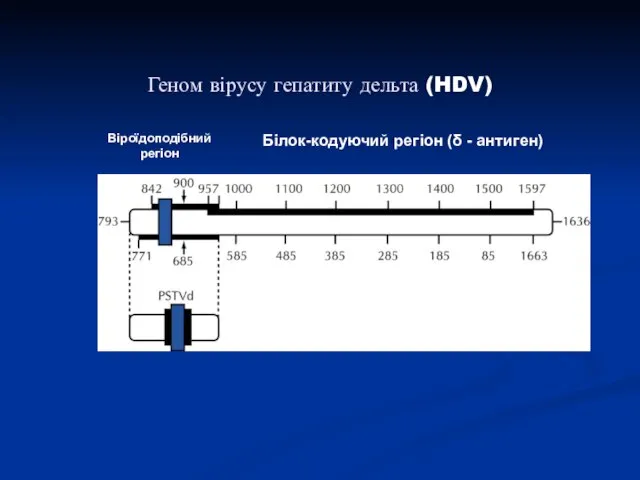
- 34. Геном вірусу гепатиту дельта (HDV) Віроїдоподібний регіон Білок-кодуючий регіон (δ - антиген)
- 35. Віріони HBV та HDV
- 36. Віроїди
- 37. Віроїди Дуже малі, ковалентно замкнені, кільцеві РНК молекули, здатні до автономної реплікації (не потребують віруса-хелпера) та
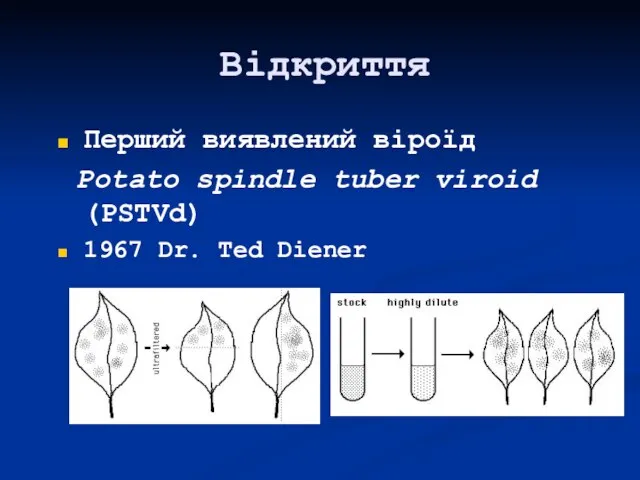
- 38. Відкриття Перший виявлений віроїд Potato spindle tuber viroid (PSTVd) 1967 Dr. Ted Diener
- 39. Potato spindle tuber viroid (PSTVd)
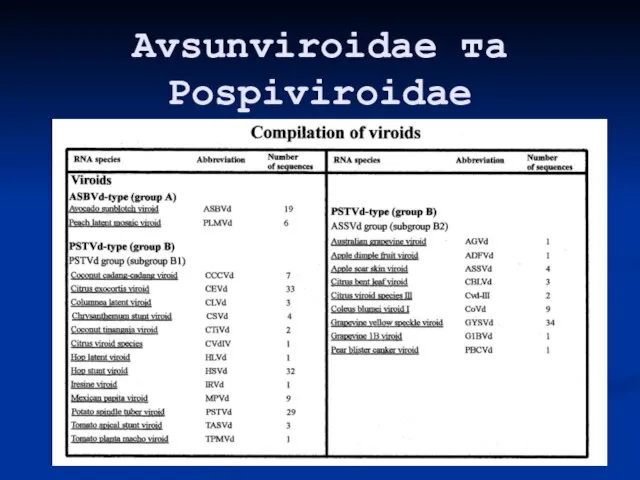
- 40. Avsunviroidae та Pospiviroidae
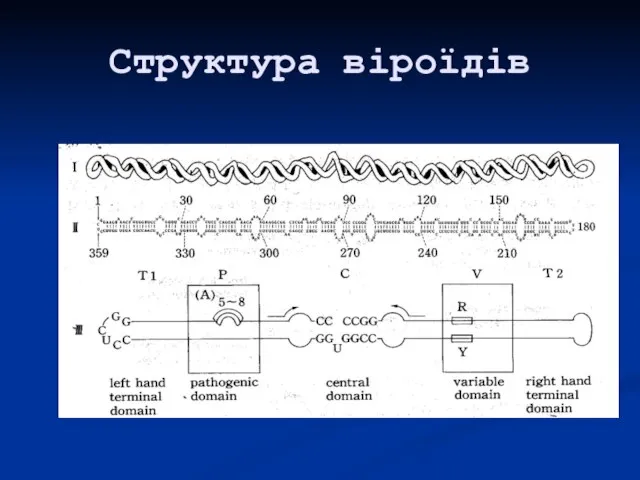
- 41. Структура віроїдів
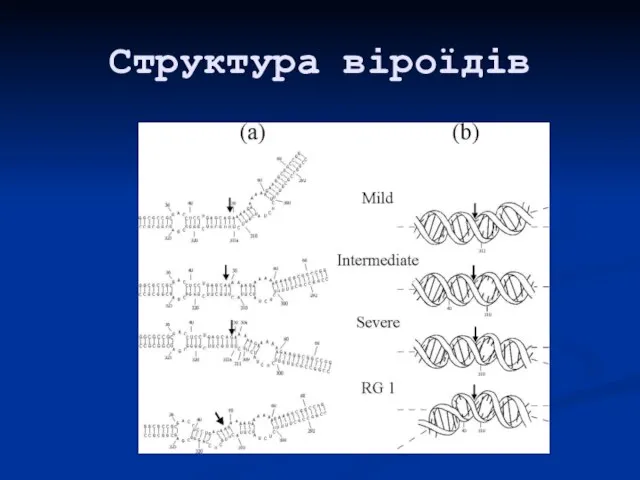
- 42. Структура віроїдів
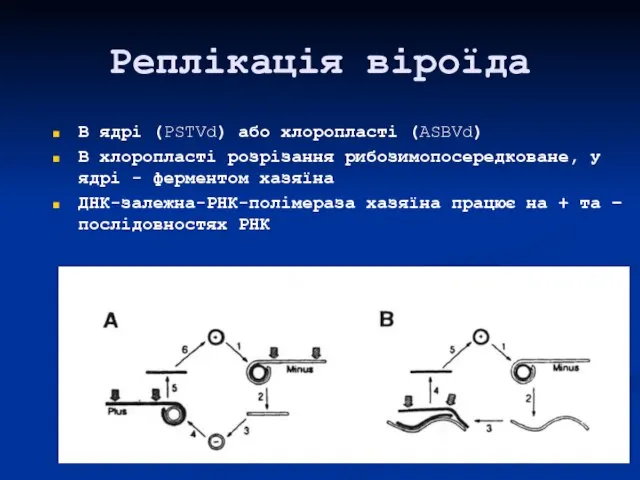
- 43. Реплікація віроїда В ядрі (PSTVd) або хлоропласті (ASBVd) В хлоропласті розрізання рибозимопосередковане, у ядрі - ферментом
- 44. Локалізація +ланцюг віроїдів локалізується і в ядерці, і в нуклеоплазмі -ланцюг – тільки у нуклеоплазмі
- 45. Переміщення віроїда для проникнення в крізь ядерну пору зв’язується з білком VirP1 Через плазмодесми Через флоему
- 46. Основні питання Які молекулярні сигнали примушують РНК-полімеразу хазяїна сприймати віроїд як матрицю для побудови комплементарного ланцюга?
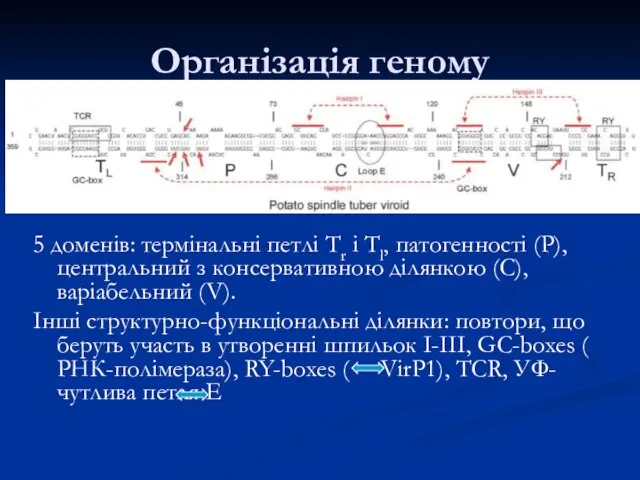
- 47. Організація геному 5 доменів: термінальні петлі Tr і Тl, патогенності (Р), центральний з консервативною ділянкою (С),
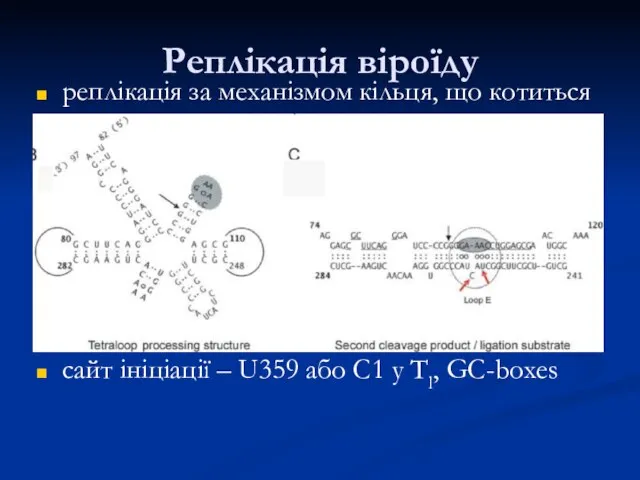
- 48. Реплікація віроїду реплікація за механізмом кільця, що котиться сайт ініціації – U359 або C1 у Тl,
- 49. -РНК локалізуються у нуклеоплазмі, де, очевидно, відбувається транскрипція, у той час як +РНК розподілені між нуклеоплазмою
- 50. відсутність movement-протеїнів мутації у правому Т-домені перешкоджають нормальному міжклітинному транспорту мутації у петлі Е блокують рух
- 51. Взаємодія з компонентами клітини хазяїна щонайменше 3 детермінанти патогенезу у доменах Т, Р і V петля
- 52. Взаємодія з компонентами клітини хазяїна інтерференція РНК господаря (суперечливі результати досліджень) взаємодія з протеїнами: - неспецифічна
- 54. Скачать презентацию





















 Многообразие насекомых Отряды: Чешуекрылые Двукрылые Равнокрылые Блохи
Многообразие насекомых Отряды: Чешуекрылые Двукрылые Равнокрылые Блохи Клонирование. Молекулярное клонирование
Клонирование. Молекулярное клонирование Отряд Вши
Отряд Вши Агрессия собак
Агрессия собак Функциональные блоки мозга
Функциональные блоки мозга Презентация на тему "Половое и бесполое размножение организмов" - скачать презентации по Биологии
Презентация на тему "Половое и бесполое размножение организмов" - скачать презентации по Биологии Отруєння їстівними грибами
Отруєння їстівними грибами  Биология – наука о живой природе
Биология – наука о живой природе Птицы жарких стран
Птицы жарких стран Биохимия крови
Биохимия крови Водный и минеральный обмен
Водный и минеральный обмен Жүректі тыңдау жүрек саздары. Жүректің қақпашаларының нақты орны
Жүректі тыңдау жүрек саздары. Жүректің қақпашаларының нақты орны Презентация на тему "Прорастание" - скачать презентации по Биологии
Презентация на тему "Прорастание" - скачать презентации по Биологии Биотехнология, ее достижения, перспективы развития
Биотехнология, ее достижения, перспективы развития Нервная ткань
Нервная ткань Морж
Морж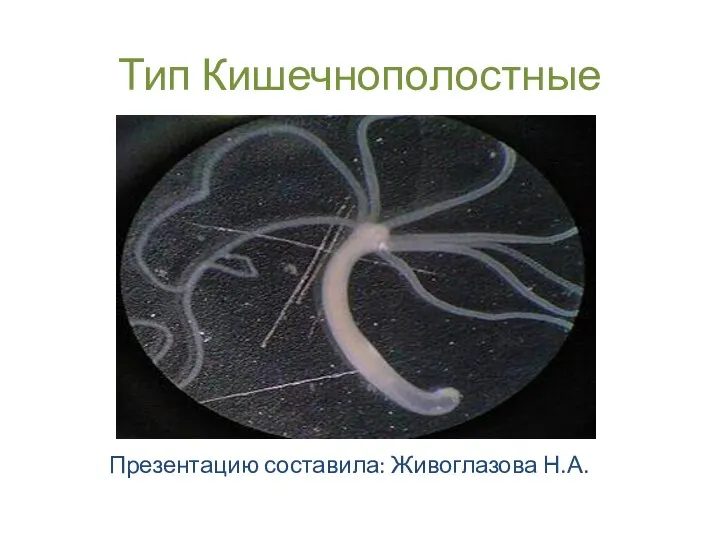 Тип Кишечнополостные Презентацию составила: Живоглазова Н.А.
Тип Кишечнополостные Презентацию составила: Живоглазова Н.А. Программированный опрос Соотнесите каждое из утверждений с правильным ответом, соединив линиями утверждение и ответ. По
Программированный опрос Соотнесите каждое из утверждений с правильным ответом, соединив линиями утверждение и ответ. По Артерии таза и нижней конечности
Артерии таза и нижней конечности Попугаи
Попугаи Ледовских Ильи 11 а класса http://vkontakte.ru/id36052741
Ледовских Ильи 11 а класса http://vkontakte.ru/id36052741 Презентация на тему Биологические катализаторы
Презентация на тему Биологические катализаторы Сравнение Японской и Техасской белой породы перепелов
Сравнение Японской и Техасской белой породы перепелов Интегрированный урок по биологии и ОБЖ на тему «Беспозвоночные животные и сохранение здоровья человека» Презентацию подготовил
Интегрированный урок по биологии и ОБЖ на тему «Беспозвоночные животные и сохранение здоровья человека» Презентацию подготовил Кольчатые черви. Общие сведения
Кольчатые черви. Общие сведения Анатомия и физиология системы пищеварения. План строения пищеварительной системы
Анатомия и физиология системы пищеварения. План строения пищеварительной системы Домашние животные
Домашние животные Одноклеточные животные или Простейшие
Одноклеточные животные или Простейшие