Содержание
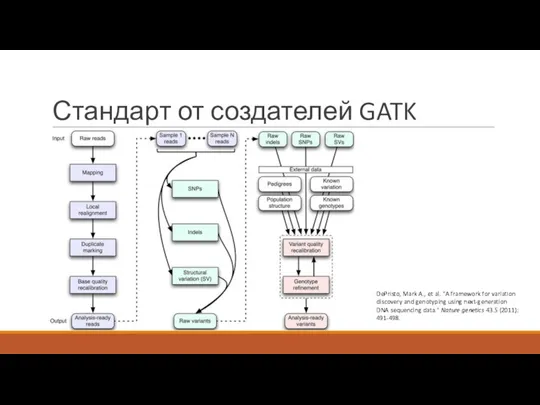
- 6. Стандарт от создателей GATK DePristo, Mark A., et al. "A framework for variation discovery and genotyping
- 7. 1. Получение данных (Fastq)
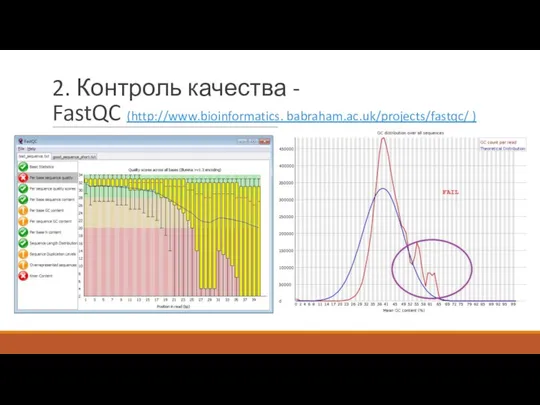
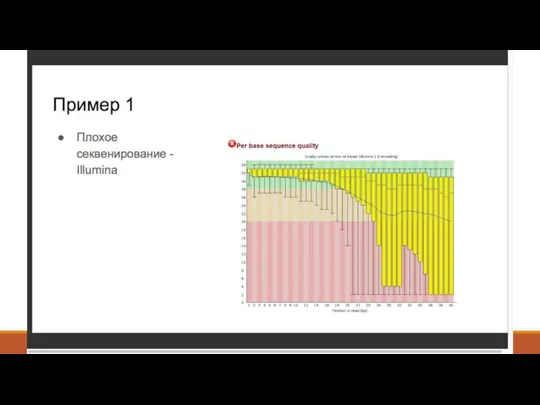
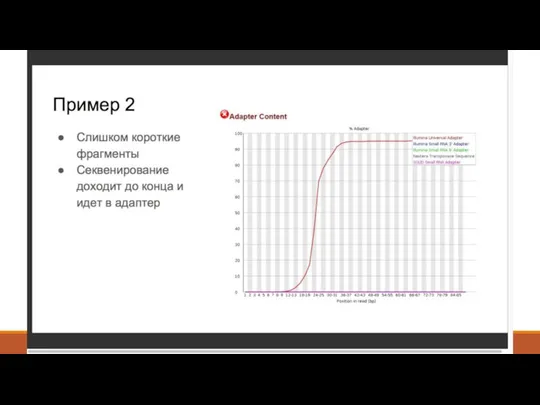
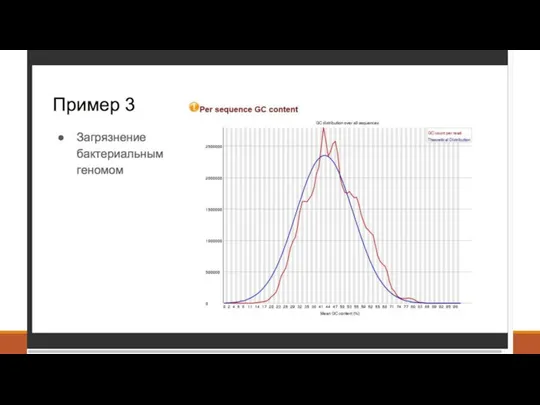
- 8. 2. Контроль качества - FastQC (http://www.bioinformatics. babraham.ac.uk/projects/fastqc/ )
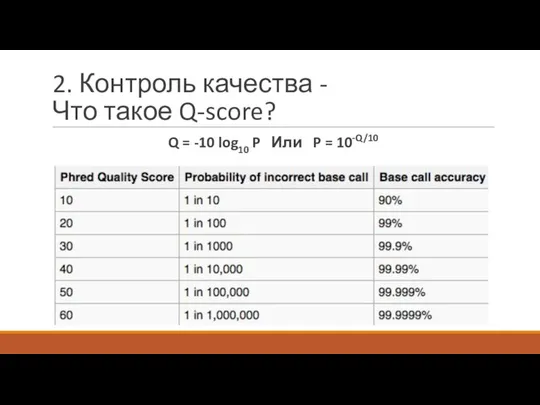
- 9. 2. Контроль качества - Что такое Q-score? Q = -10 log10 P Или P = 10-Q/10
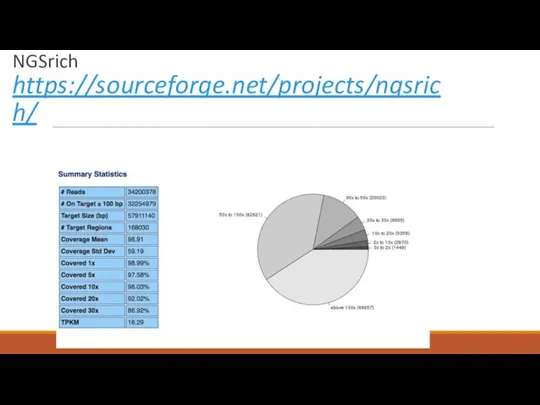
- 18. NGSrich https://sourceforge.net/projects/ngsrich/
- 20. 3. Выравнивание ридов на геном – Как это выглядит Нужно: Риды – FastQ Референс / Индекс
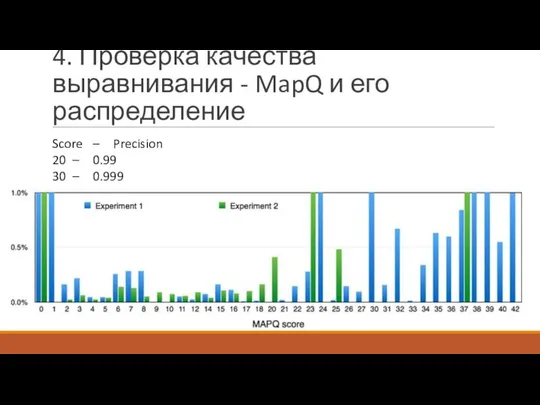
- 22. 4. Проверка качества выравнивания - MapQ и его распределение Score – Precision 20 – 0.99 30
- 23. 5. Поиск вариантов - Как это работает
- 24. VCF – формат данных
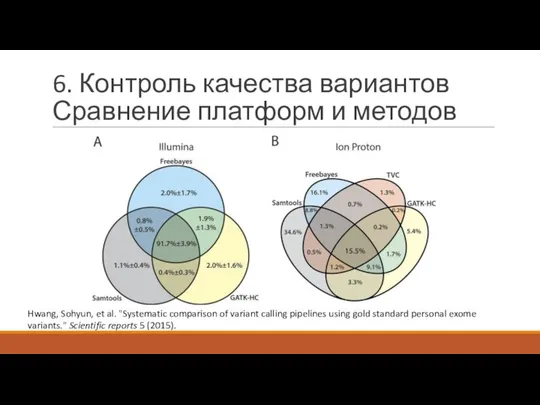
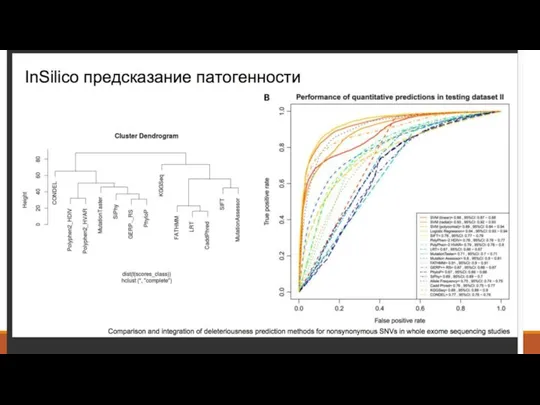
- 25. 6. Контроль качества вариантов Сравнение платформ и методов Hwang, Sohyun, et al. "Systematic comparison of variant
- 26. Alignment and Variant Calling Broken Down 2012 2 VCFs from 23andMe BWA 0.6.1 GATK (early &
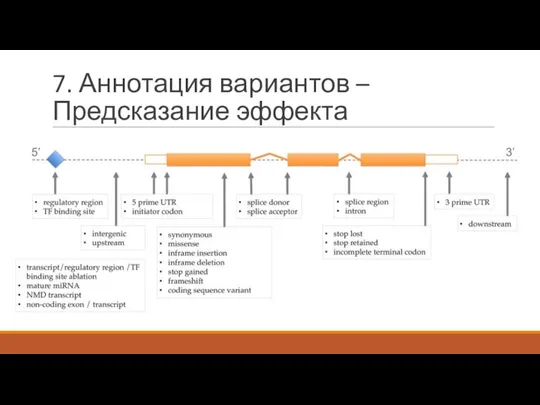
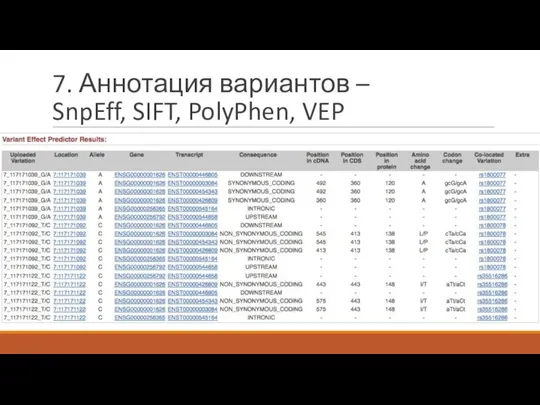
- 27. 7. Аннотация вариантов – Предсказание эффекта
- 28. 7. Аннотация вариантов – SnpEff, SIFT, PolyPhen, VEP
- 29. VCF-Annotate Пример запуска: vcf-annotate -f +/d=8/Q=10/q=10/-a > Ключ Описание [стандартное значение] 1, StrandBias FLOAT Min P-value
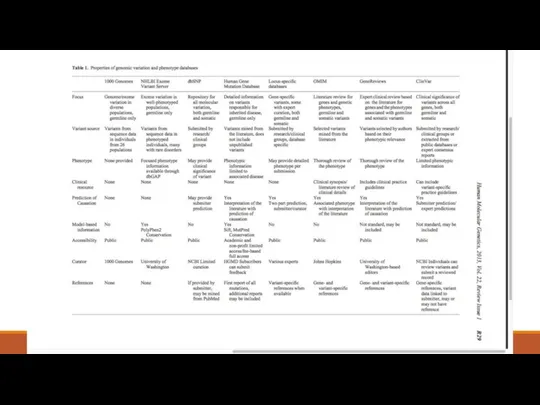
- 30. Аннотация вариантов Chr Start End Ref Alt Func Gene GeneDetail ExonicFunc AAChange 1000G ALL 1000G AFR
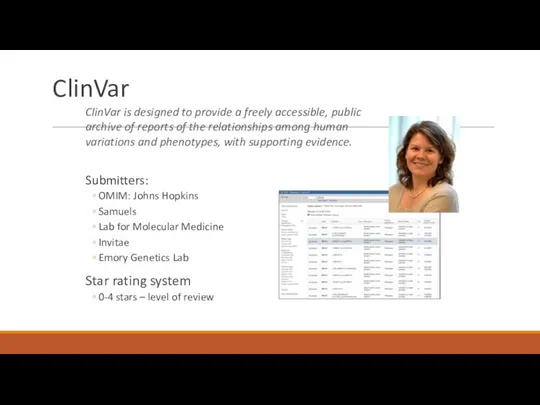
- 32. ClinVar Submitters: OMIM: Johns Hopkins Samuels Lab for Molecular Medicine Invitae Emory Genetics Lab Star rating
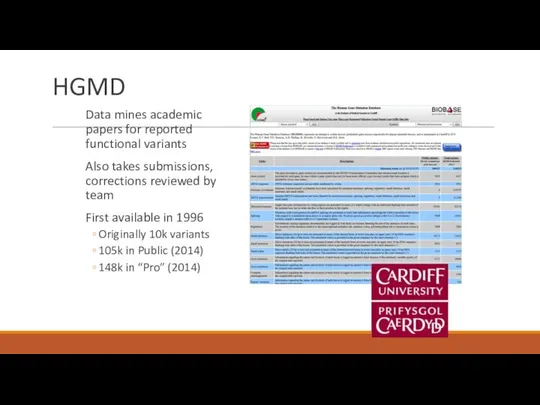
- 33. HGMD Data mines academic papers for reported functional variants Also takes submissions, corrections reviewed by team
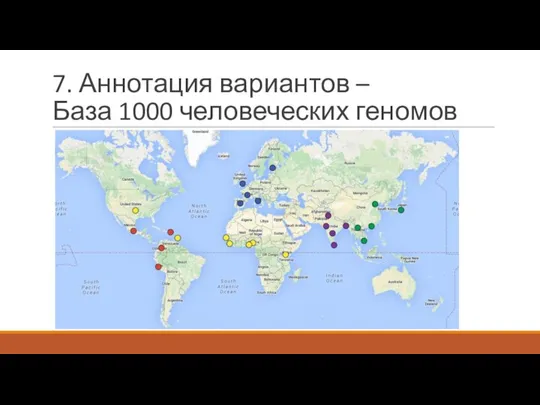
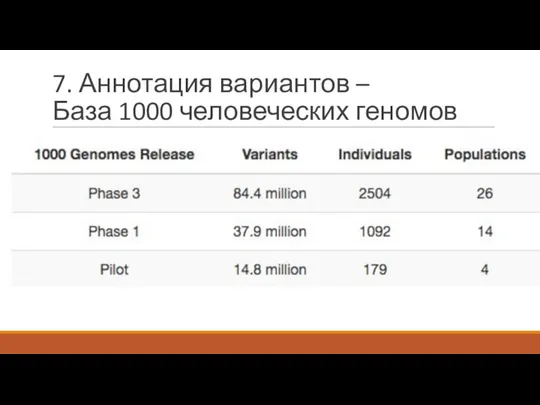
- 34. 7. Аннотация вариантов – База 1000 человеческих геномов
- 35. 7. Аннотация вариантов – База 1000 человеческих геномов
- 36. NHLBI Exome Sequencing Project (ESP) http://evs.gs.washington.edu/EVS/
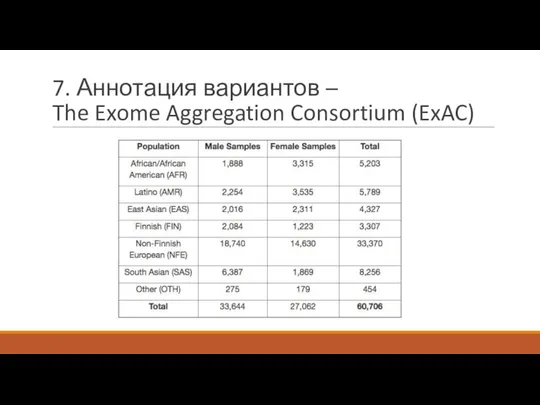
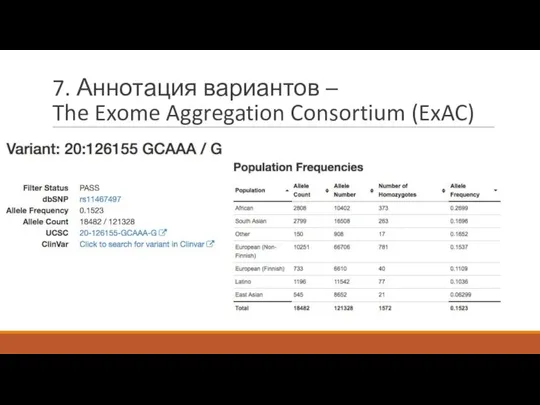
- 37. 7. Аннотация вариантов – The Exome Aggregation Consortium (ExAC)
- 38. 7. Аннотация вариантов – The Exome Aggregation Consortium (ExAC)
- 39. 7. Аннотация вариантов – The Exome Aggregation Consortium (ExAC)
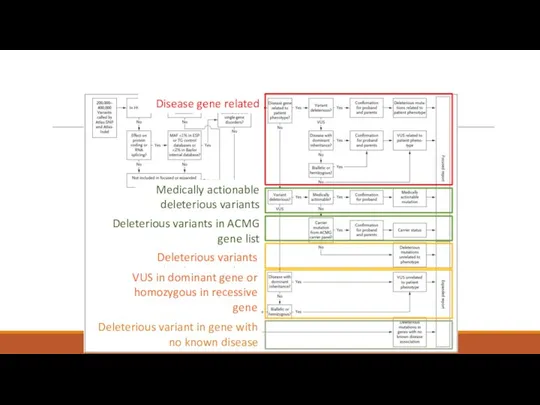
- 41. Disease gene related Medically actionable deleterious variants Deleterious variants in ACMG gene list Deleterious variants VUS
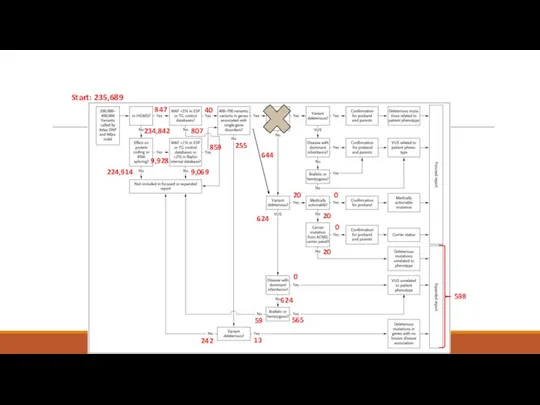
- 42. Start: 235,689 847 234,842 224,914 9,928 9,069 807 859 40 242 13 59 565 0 624
- 43. Резюме по анализу экзомов Детальное изучение клиники и семейной истории для формирования клинико-генетической гипотезы 2 основных
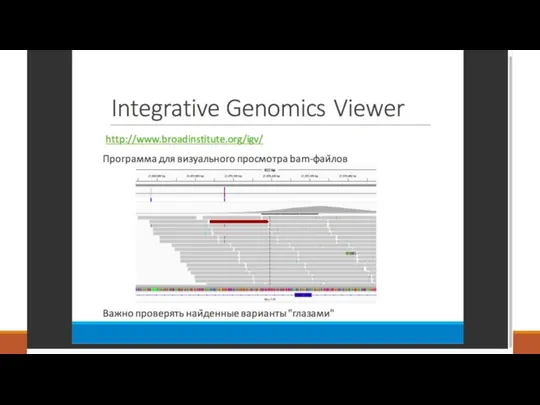
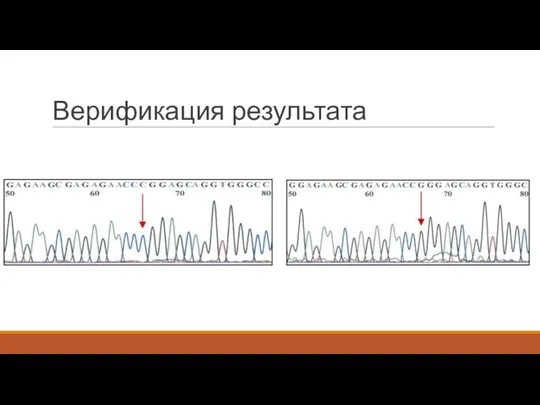
- 46. Верификация результата
- 48. Скачать презентацию










![VCF-Annotate Пример запуска: vcf-annotate -f +/d=8/Q=10/q=10/-a > Ключ Описание [стандартное значение]](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/1364589/slide-28.jpg)





 ККО Прокопьевск-ПК-Угольный. Система видеонаблюдения
ККО Прокопьевск-ПК-Угольный. Система видеонаблюдения Как нарисовать рыбку? - презентация для начальной школы_
Как нарисовать рыбку? - презентация для начальной школы_ Государственное регулирование охраны здоровья граждан, здравоохранения и медицинской деятельности
Государственное регулирование охраны здоровья граждан, здравоохранения и медицинской деятельности Геополитические взаимоотношения России и Китая
Геополитические взаимоотношения России и Китая Проект о создание Туризма В Шегарском районе, области, стране, зарубежье
Проект о создание Туризма В Шегарском районе, области, стране, зарубежье Кондиционирование воздуха
Кондиционирование воздуха Социальные сети - современный инструмент для профессионального общения библиотекарей
Социальные сети - современный инструмент для профессионального общения библиотекарей древнерусская литература 1 ур
древнерусская литература 1 ур ТЕЛЕСКОПИ
ТЕЛЕСКОПИ Влияние алкоголя на организм человека Исполнители: Поротников Антон, Паклин Роман ученики 8 «б» класс
Влияние алкоголя на организм человека Исполнители: Поротников Антон, Паклин Роман ученики 8 «б» класс Люсьен Теньер
Люсьен Теньер Альбрехт Дюрер (1471-1528)
Альбрехт Дюрер (1471-1528) Основные способы плавания
Основные способы плавания Организация крупного фармпроизводства в соответствии со стандартом GMP
Организация крупного фармпроизводства в соответствии со стандартом GMP Травматизм. Первичная и вторичная профилактика. Первая помощь при травмах.
Травматизм. Первичная и вторичная профилактика. Первая помощь при травмах.  грибы - презентация для начальной школы
грибы - презентация для начальной школы Презентация Лизинг
Презентация Лизинг Эпиграфы
Эпиграфы Expression language
Expression language Мотивация деятельности в менеджменте
Мотивация деятельности в менеджменте  Практика привлечения к субсидиарной ответственности
Практика привлечения к субсидиарной ответственности Заглавная буква в именах существительных - презентация для начальной школы_
Заглавная буква в именах существительных - презентация для начальной школы_ Asynchronous Transfer Mode Обзор технологии
Asynchronous Transfer Mode Обзор технологии Класс Земноводные
Класс Земноводные «Путешествие по страницам школьной жизни» Составитель: Иванова Марина Васильевна, учитель начальн
«Путешествие по страницам школьной жизни» Составитель: Иванова Марина Васильевна, учитель начальн Відмінювання числівників
Відмінювання числівників Программирование на языке Си
Программирование на языке Си Презентация Исполнительная власть в механизме государственной власти
Презентация Исполнительная власть в механизме государственной власти