Содержание
- 2. Лекция 6: Трансгенные растения-4 Получение инсерционных мутантов растений на основе агробактериальной трансформации Клонирование генов Изучение с
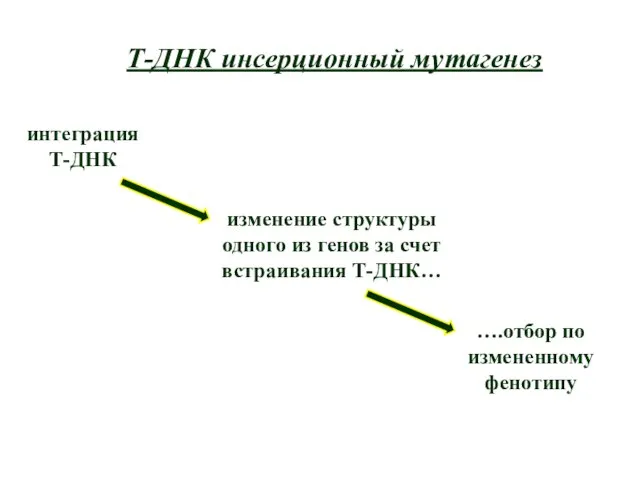
- 3. Т-ДНК инсерционный мутагенез интеграция Т-ДНК изменение структуры одного из генов за счет встраивания Т-ДНК… ….отбор по
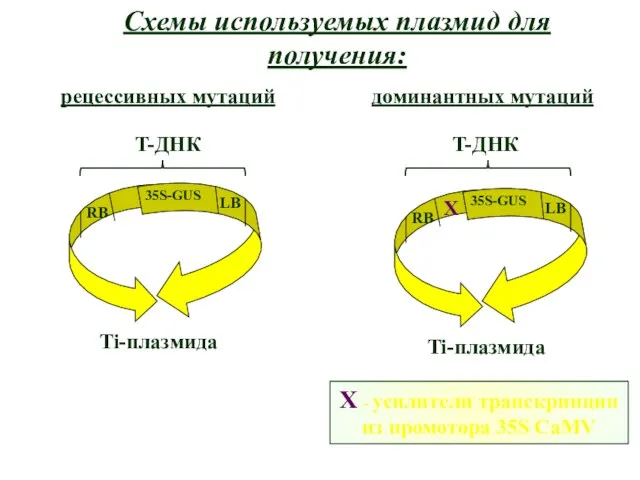
- 4. Схемы используемых плазмид для получения: рецессивных мутаций доминантных мутаций X - усилители транскрипции из промотора 35S
- 5. Клонирование генов
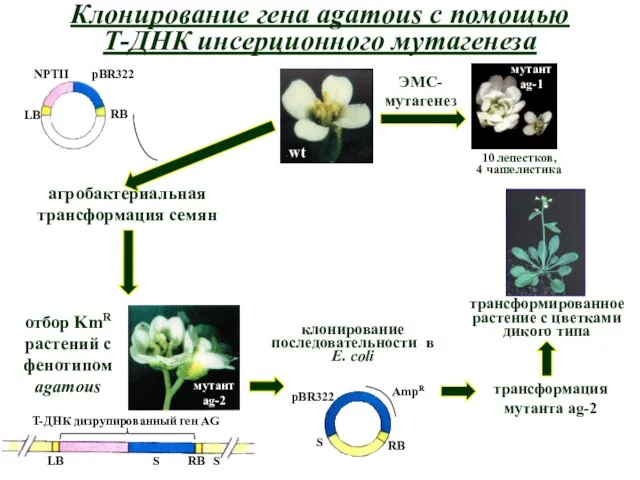
- 6. Клонирование гена agamous с помощью Т-ДНК инсерционного мутагенеза ЭМС-мутагенез 10 лепестков, 4 чашелистика агробактериальная трансформация семян
- 7. Ген, маркированный транспозоном (tagged gene), можно клонировать с помощью «вытягивания за транспозон»
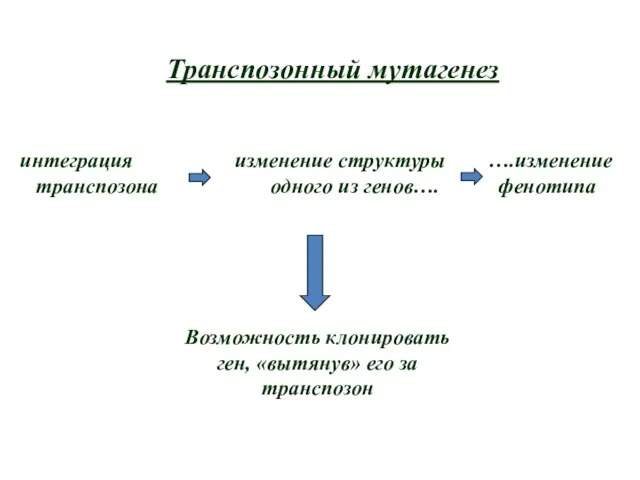
- 8. Транспозонный мутагенез интеграция изменение структуры ….изменение транспозона одного из генов…. фенотипа Возможность клонировать ген, «вытянув» его
- 9. При встраивании транспозона в кодирующую последовательность гена, наблюдается мутантный фенотип. При вырезании фенотип восстанавливается
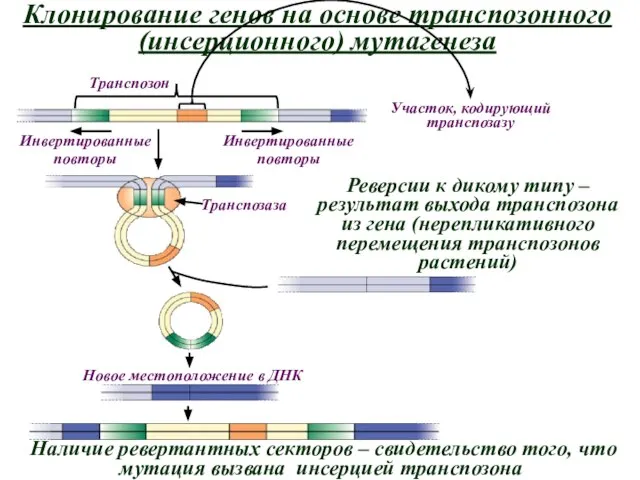
- 10. Клонирование генов на основе транспозонного (инсерционного) мутагенеза
- 11. Клонирование генов на основе транспозонного (инсерционного) мутагенеза
- 12. Получение мутации flo613 и клонирование гена FLORICAULA львиного зева (Coen et al., 1990) Выращивание 26 000
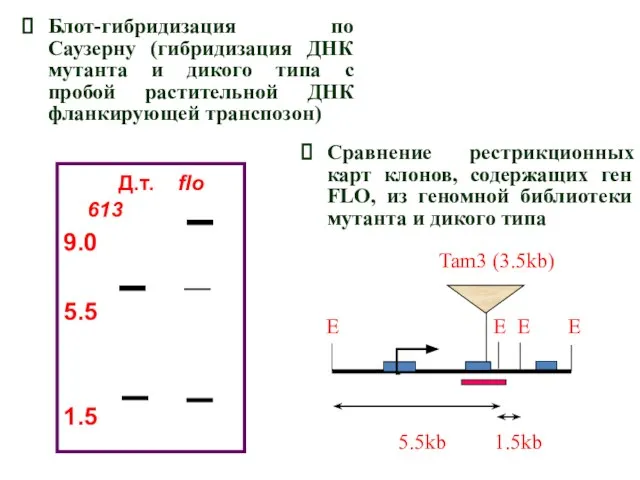
- 13. Рестрикционный анализ ДНК из выделенных клонов, необходимый для выявления участков растительной ДНК, фланкирующих транспозон Субклонирование растительной
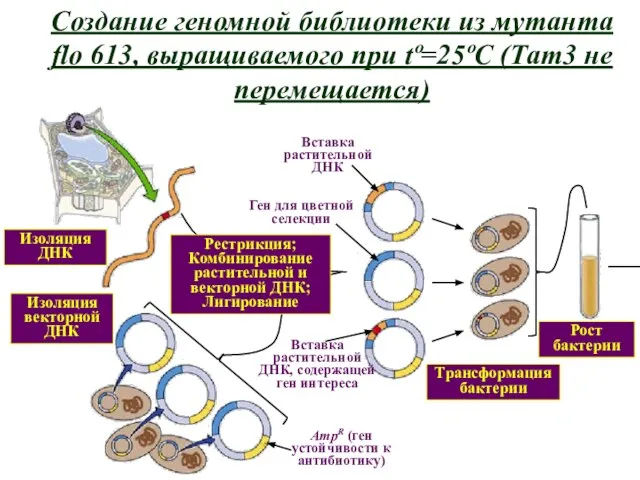
- 14. Создание геномной библиотеки из мутанта flo 613, выращиваемого при tº=25ºC (Тam3 не перемещается)
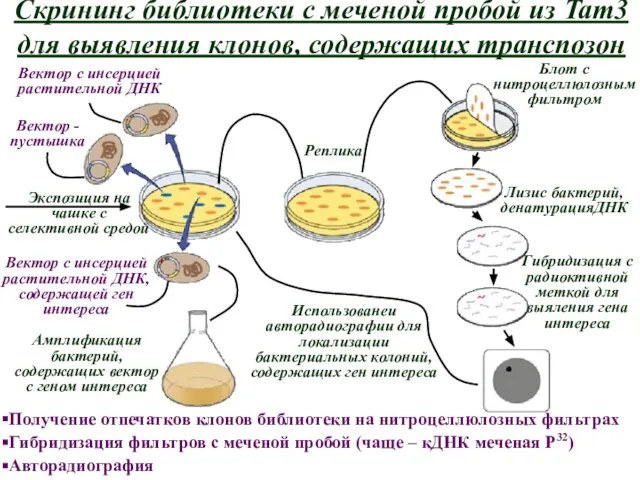
- 15. Скрининг библиотеки с меченой пробой из Tam3 для выявления клонов, содержащих транспозон Получение отпечатков клонов библиотеки
- 16. Д.т. flo 613 9.0 5.5 1.5 Сравнение рестрикционных карт клонов, содержащих ген FLO, из геномной библиотеки
- 17. Ген FLORICAULA – уникальный ген, присутствующий в геноме покрытосеменных в 1 копии. Он кодирует белок, содержащий
- 18. Изучение механизмов регуляции экспрессии генов растений с использованием репортерных (индикаторных) генов Возможности метода: Выявление факторов (внешних
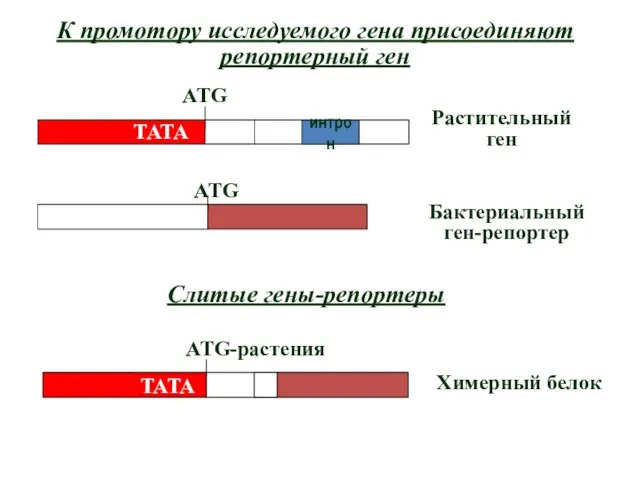
- 19. Слитые гены-репортеры К промотору исследуемого гена присоединяют репортерный ген
- 20. Green fluorescent protein (GFP) Luciferase (LUC) ß-glucuronidase (GUS) Promoter-reporter fusion: Использование гибридных конструкций, содержащих репортерный ген,
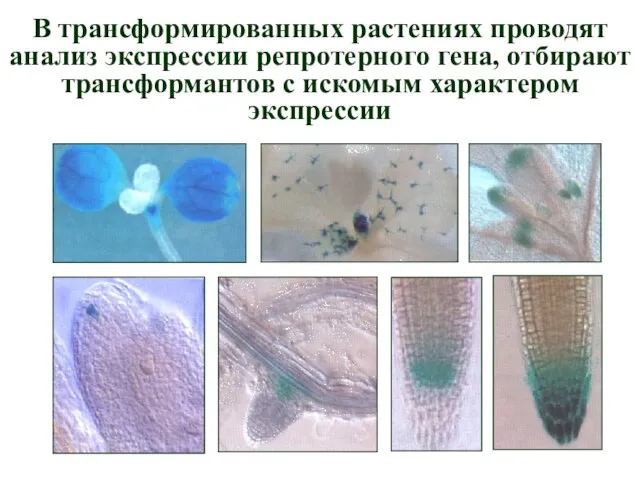
- 21. В трансформированных растениях проводят анализ экспрессии репротерного гена, отбирают трансформантов с искомым характером экспрессии
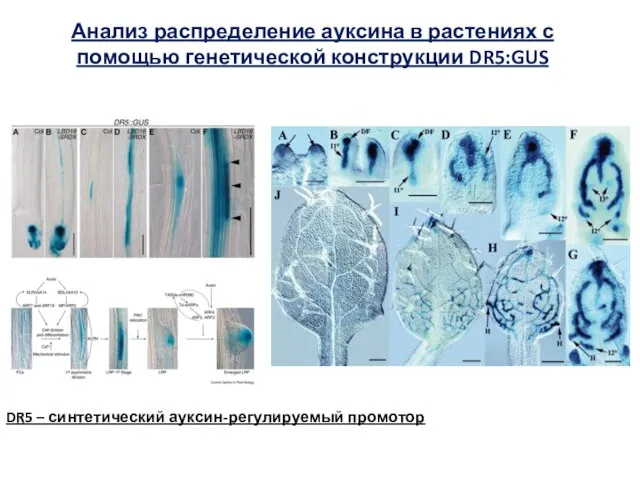
- 22. Анализ распределение ауксина в растениях с помощью генетической конструкции DR5:GUS DR5 – синтетический ауксин-регулируемый промотор
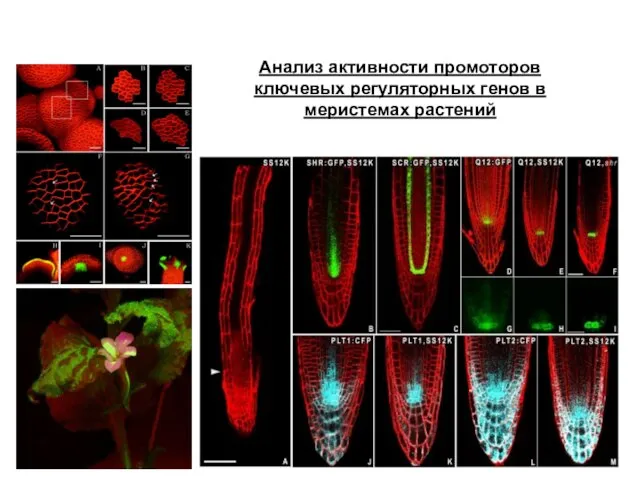
- 23. Анализ активности промоторов ключевых регуляторных генов в меристемах растений
- 24. Получение растений со сверхэкспрессией гена с целью изучения функции гена
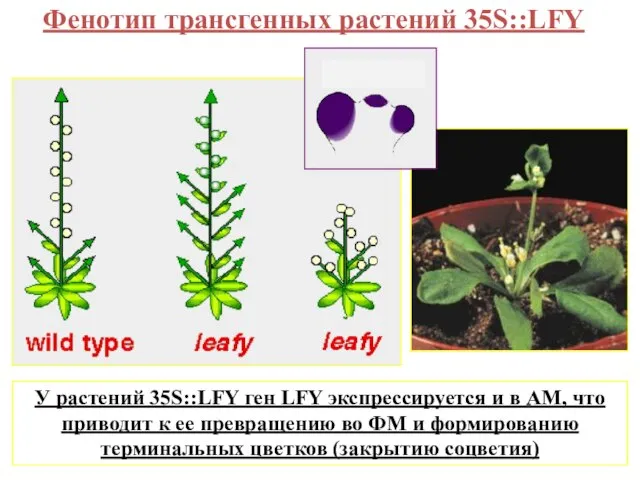
- 25. Фенотип трансгенных растений 35S::LFY У растений 35S::LFY ген LFY экспрессируется и в АМ, что приводит к
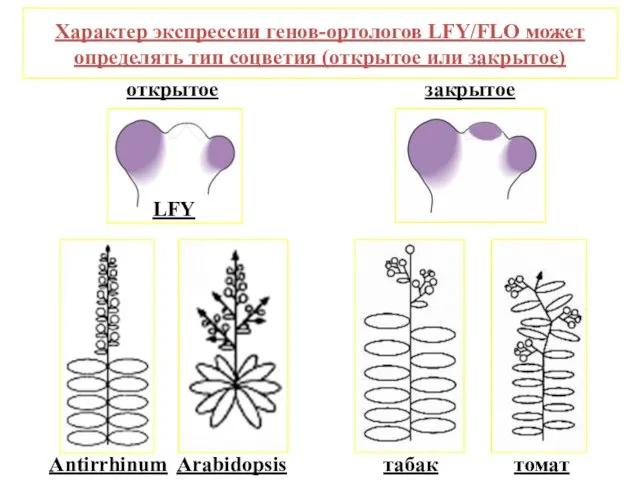
- 26. Характер экспрессии генов-ортологов LFY/FLO может определять тип соцветия (открытое или закрытое)
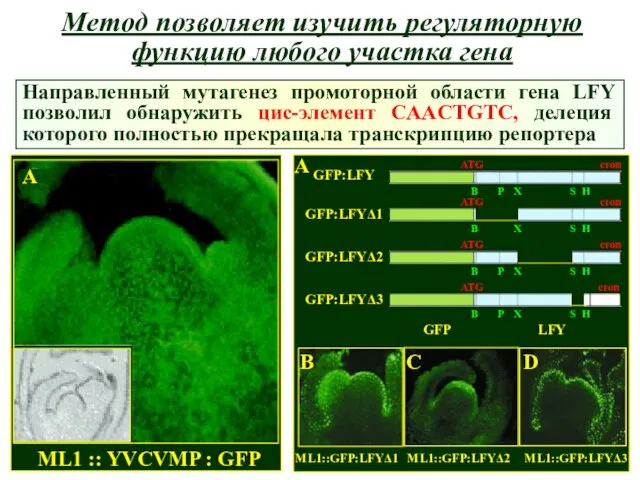
- 27. Метод позволяет изучить регуляторную функцию любого участка гена Направленный мутагенез промоторной области гена LFY позволил обнаружить
- 28. Мутант арабидопсиса по гену LFY
- 29. Основные семейства транскрипционных факторов растений Консервативные для эукариот: Уникальные для растений: ТФ с MADS-доменом ТФ с
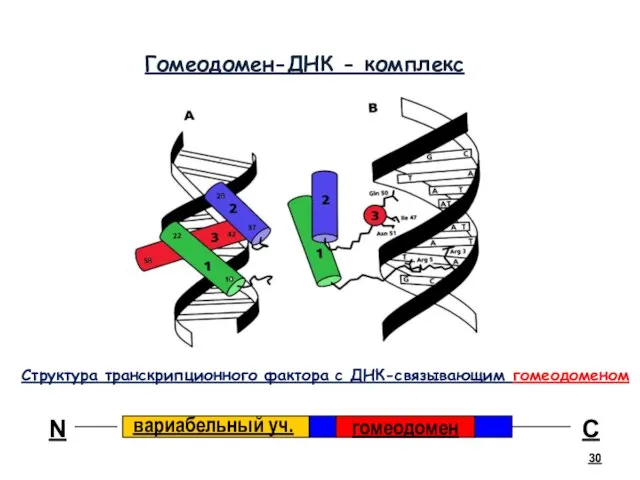
- 30. Гомеодомен-ДНК - комплекс вариабельный уч. гомеодомен N С Структура транскрипционного фактора с ДНК-связывающим гомеодоменом
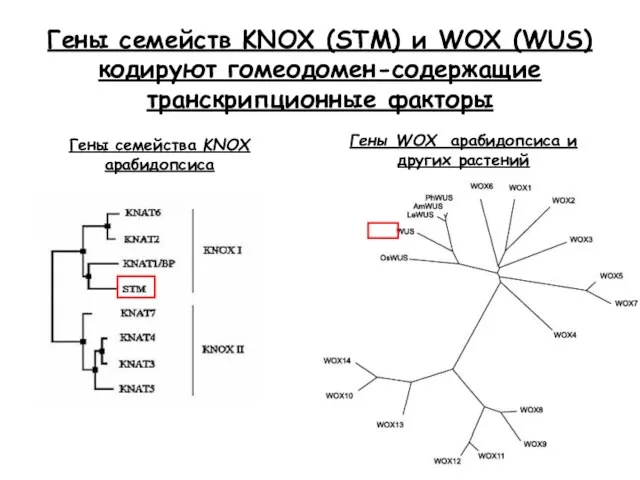
- 31. Гены семейств KNOX (STM) и WOX (WUS) кодируют гомеодомен-содержащие транскрипционные факторы Гены семейства KNOX арабидопсиса Гены
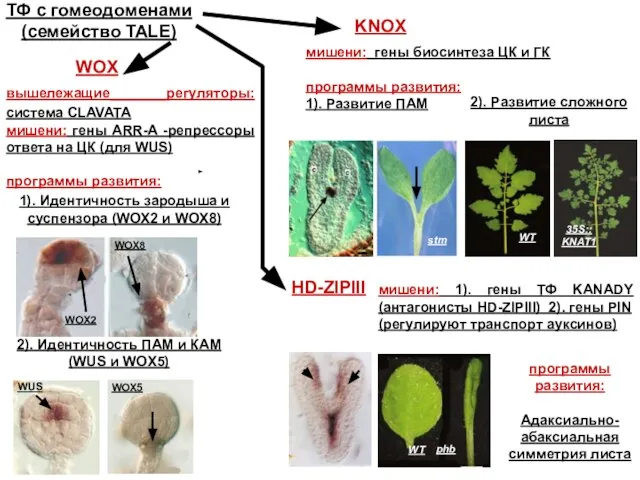
- 32. ТФ с гомеодоменами (семейство TALE) KNOX мишени: гены биосинтеза ЦК и ГК программы развития: 1). Развитие
- 33. Ген KNOTTED1 был идентифи-цирован на основе доминантных мутаций kn1, вызывающих отсутствие лигулы и появление узелков (knots)
- 34. Зная о важной роли гомеобоксных генов в развитии животных, приступили к клонированию и изучению KNOTTED1-подобных генов
- 35. Сверхэкспрессия гена KNAT1 у арабидопсиса На листьях возникали дополнительные меристематические очаги, из которых могли возникать почки
- 36. Сверхэкспрессия гена KNAT в листьях томатов дикий тип 35S::KNAT
- 37. KNOTTED-подобные гены – возможные участники морфологической эволюции растений
- 38. Обнаружение связи между уровнем экспрессии KNOX-генов и усложнением структуры листа позволило предположить, что эти гены могли
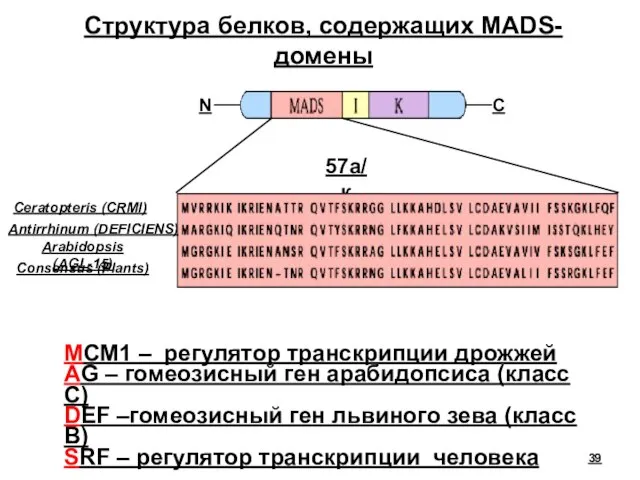
- 39. MCM1 – регулятор транскрипции дрожжей AG – гомеозисный ген арабидопсиса (класс С) DEF –гомеозисный ген львиного
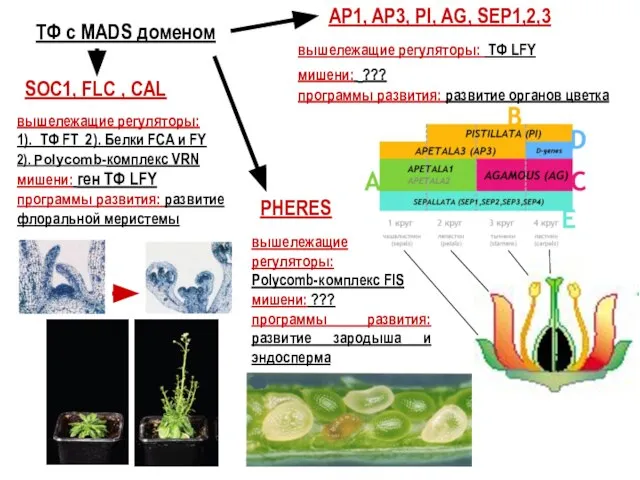
- 40. ТФ с MADS доменом вышележащие регуляторы: ТФ LFY мишени: ??? программы развития: развитие органов цветка AP1,
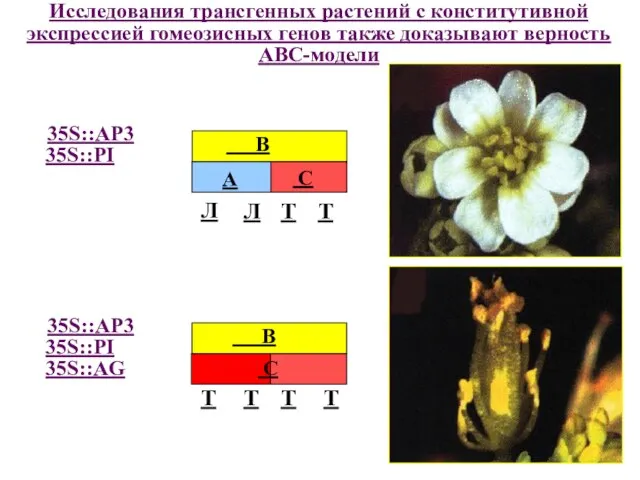
- 41. Исследования трансгенных растений с конститутивной экспрессией гомеозисных генов также доказывают верность АВС-модели 35S::AP3 35S::PI 35S::AP3 35S::PI
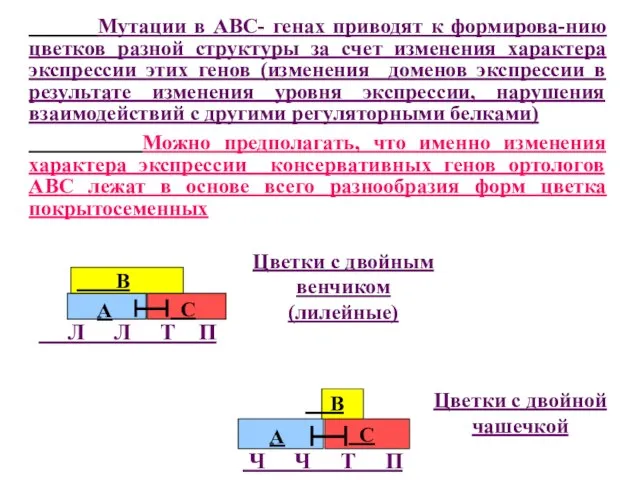
- 42. Цветки с двойным венчиком (лилейные) Цветки с двойной чашечкой Мутации в АВС- генах приводят к формирова-нию
- 43. Использование методов «генетической хирургии» для изучения взаимодействия клеток и тканей в процессах развития
- 44. Day, Galgoci, Irish, 1995. Генетическое удаление лепестков и тычинок для выяснения роли клеточных взаимодействий в развитии
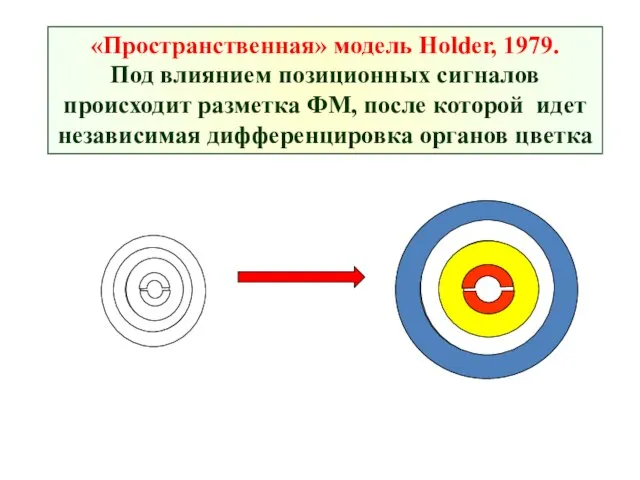
- 45. «Пространственная» модель Holder, 1979. Под влиянием позиционных сигналов происходит разметка ФМ, после которой идет независимая дифференцировка
- 46. В основе «генетической хирургии» лежит метод трансформации растений химерными генами следующей конструкции: Кодирующая часть гена –
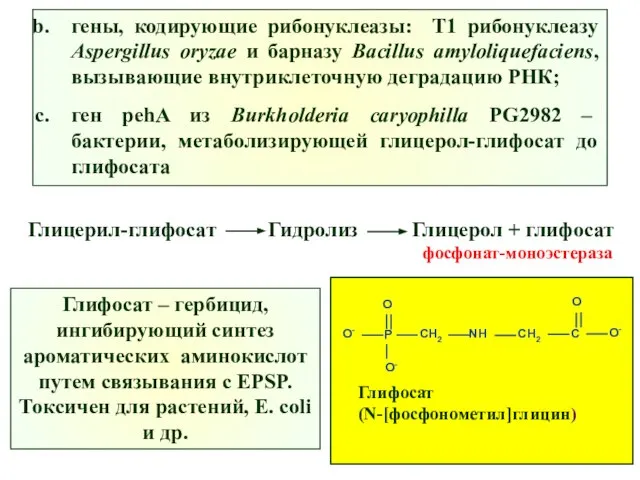
- 47. гены, кодирующие рибонуклеазы: Т1 рибонуклеазу Aspergillus oryzae и барназу Bacillus amyloliquefaciens, вызывающие внутриклеточную деградацию РНК; ген
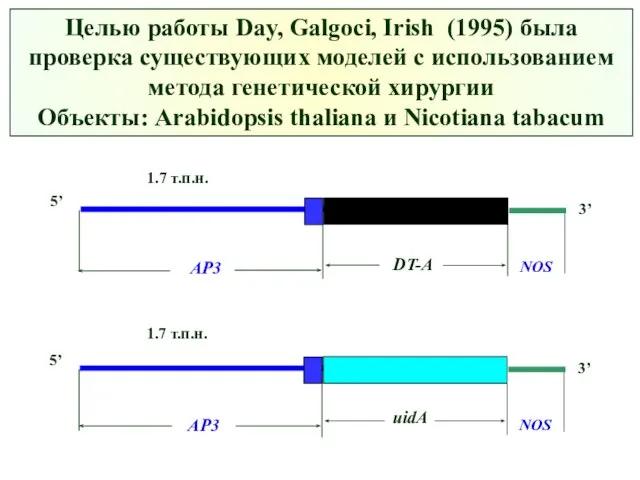
- 48. Целью работы Day, Galgoci, Irish (1995) была проверка существующих моделей с использованием метода генетической хирургии Объекты:
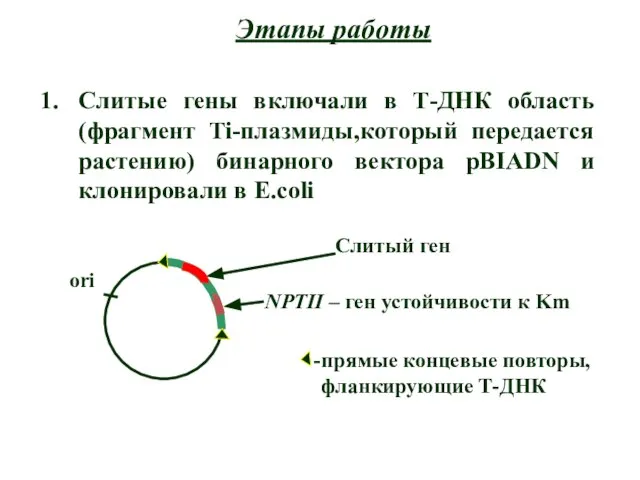
- 49. Этапы работы Слитые гены включали в Т-ДНК область (фрагмент Ti-плазмиды,который передается растению) бинарного вектора pBIADN и
- 50. У резушки получено 5 трансгенных растений, в геноме которых присутствовали от 1 до 5 копий трансгенов
- 51. Использование трансформации для изучения роли гормонов в развитиии растений
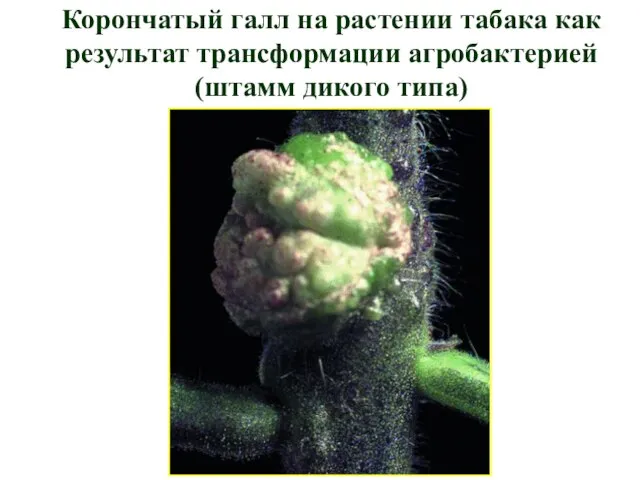
- 53. Корончатый галл на растении табака как результат трансформации агробактерией (штамм дикого типа)
- 54. Трансгенные рстения с геном tmr
- 55. Ген tmr в трансгенных растениях, экспрессирующийся в плодах (промотор 2А11)
- 56. Опухолеобразование у редиса – генетический признак
- 57. Фенокопии опухолеобразования у трансгенных растений с геном ipt
- 58. Прямая генетика: фенотип ген Обратная генетика: ген фенотип Прямая и обратная генетика в изучении функции гена
- 59. - гомологичная рекомбинация/замещение гена -замещение гена: альтернативные подходы (Zn-fingers nucleases, TALEN) - РНК-интерференция/ генный сайленсинг -
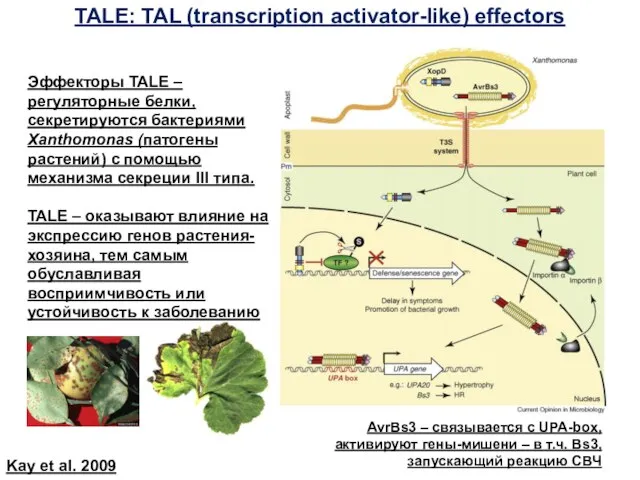
- 60. TALE: TAL (transcription activator-like) effectors Эффекторы TALE – регуляторные белки, секретируются бактериями Xanthomonas (патогены растений) с
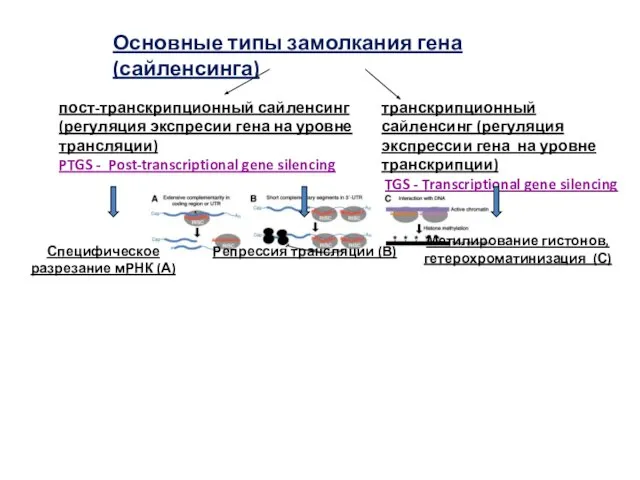
- 61. Основные типы замолкания гена (сайленсинга) пост-транскрипционный сайленсинг (регуляция экспресии гена на уровне трансляции) PTGS - Post-transcriptional
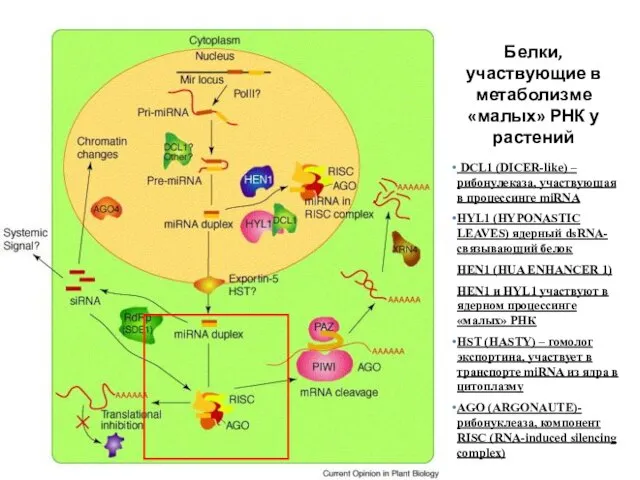
- 62. Белки, участвующие в метаболизме «малых» РНК у растений DCL1 (DICER-like) – рибонулеказа, участвующая в процессинге miRNA
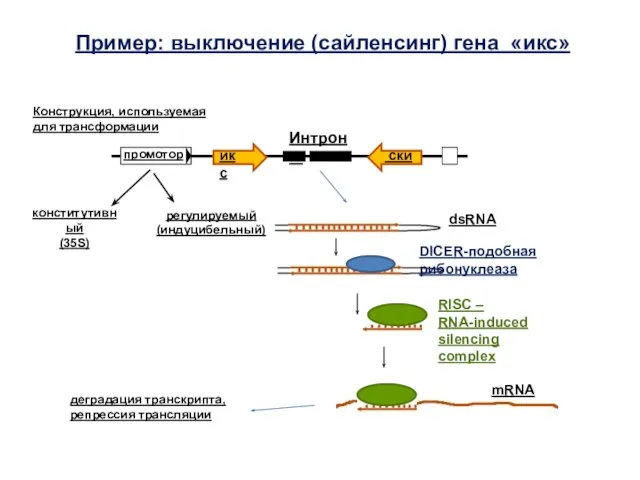
- 63. Пример: выключение (сайленсинг) гена «икс» конститутивный (35S) регулируемый (индуцибельный) Интроны промотор икс ски dsRNA RISC –
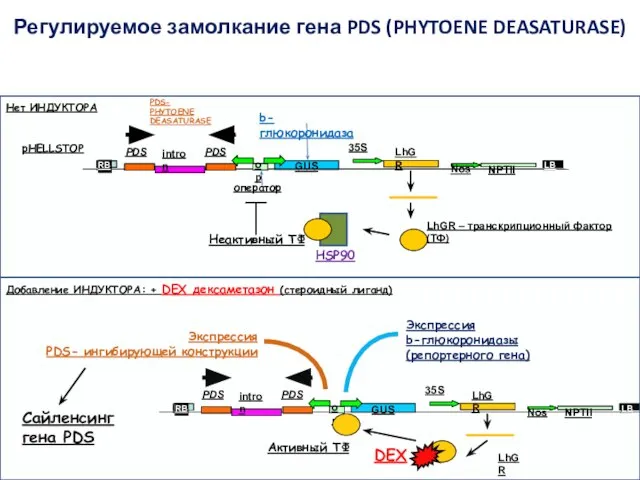
- 64. Регулируемое замолкание гена PDS (PHYTOENE DEASATURASE) LB RB RB LB 35S LhGR GUS op PDS PDS
- 65. Регулируемое замолкание (индуцибельный сайленсинг) гена PDS (PHYTOENE DEASATURASE) No Dex Dex
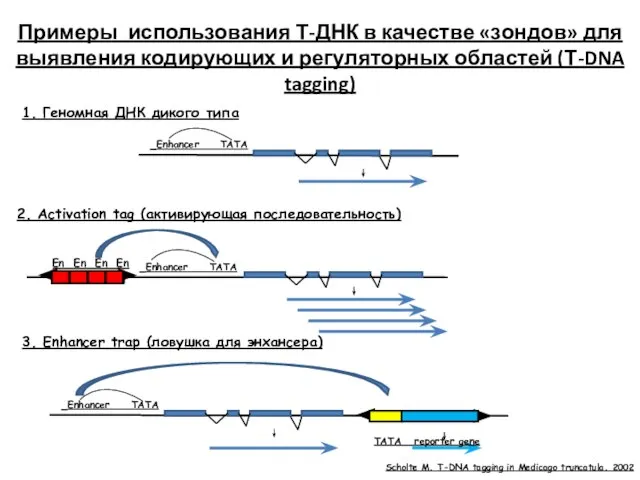
- 66. Enhancer TATA En En En En Enhancer TATA Enhancer TATA 3. Enhancer trap (ловушка для энхансера)
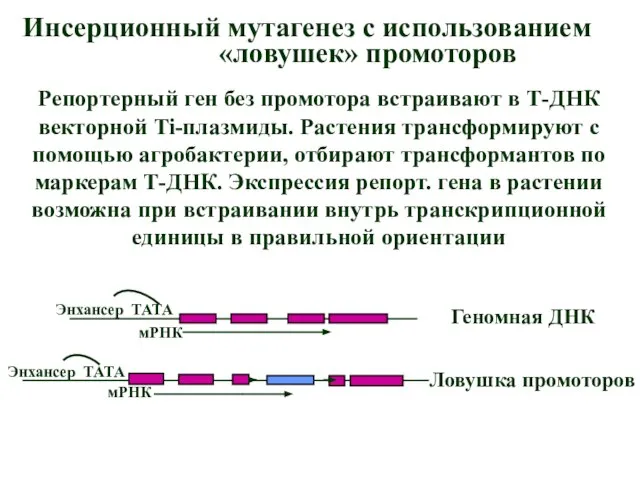
- 67. Инсерционный мутагенез с использованием «ловушек» промоторов Репортерный ген без промотора встраивают в Т-ДНК векторной Ti-плазмиды. Растения
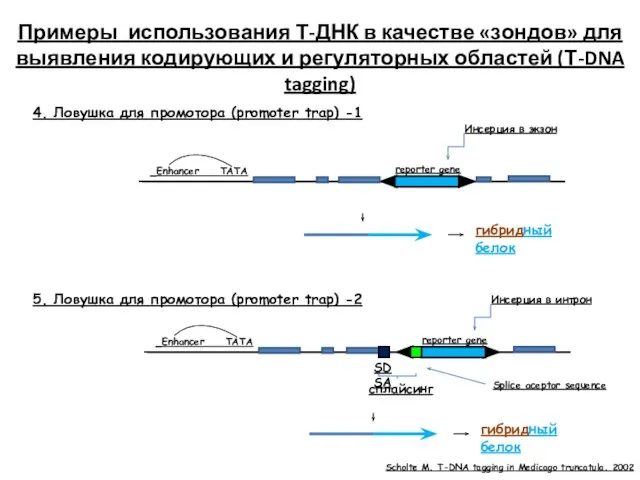
- 68. Enhancer TATA 4. Ловушка для промотора (promoter trap) -1 reporter gene гибридный белок Инсерция в экзон
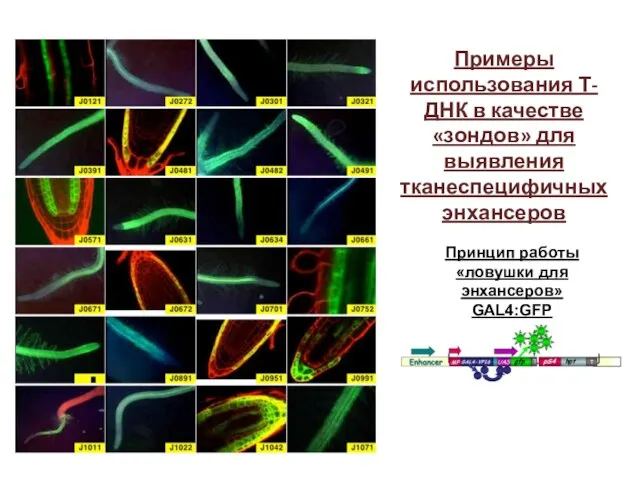
- 69. Принцип работы «ловушки для энхансеров» GAL4:GFP Примеры использования Т-ДНК в качестве «зондов» для выявления тканеспецифичных энхансеров
- 70. Методы редактирования генома растений
- 71. Модификации генома: разновидности Изменение случайного места в геноме Редактирование генома
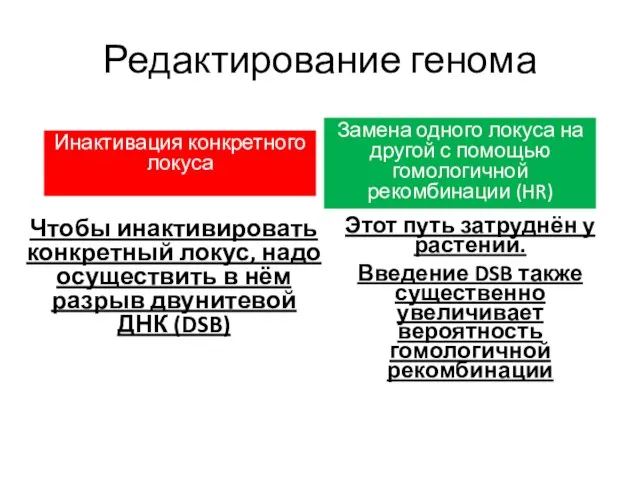
- 72. Редактирование генома Инактивация конкретного локуса Замена одного локуса на другой с помощью гомологичной рекомбинации (HR) Этот
- 73. Программируемые нуклеазы ZFN (Zink-Finger Nucleases) TALENs (Transcription Activator-Like Effector Nucleases) CRISPR-Cas система (Clustered regularly Interspaced Palindromic
- 74. Нуклеазы с цинковыми пальцами В качестве ДНК-связывающего домена используется универсальный домен типа «Цинковые пальцы»
- 76. Как программируют ZFN? Wolfe et al., 1999 Каждый цинковый палец распознаёт 3 (4) нуклеотида. Обычно используют
- 77. TALEN (Transcription Activator-Like Effectors Nucleases)
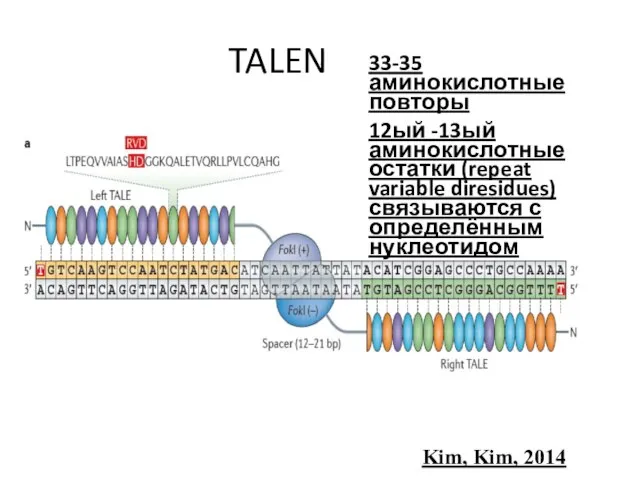
- 78. TALEN Kim, Kim, 2014 33-35 аминокислотные повторы 12ый -13ый аминокислотные остатки (repeat variable diresidues) связываются с
- 79. Применение TALEN Потеря функции гена LOX3 (кодирует липоксигеназу, создающую перекись водорода) увеличивает сроки хранения семян Этот
- 80. Система CRISPR/Cas9 (Clustered regularly Interspaced Palindromic Repeats- CRISPR associated protein)
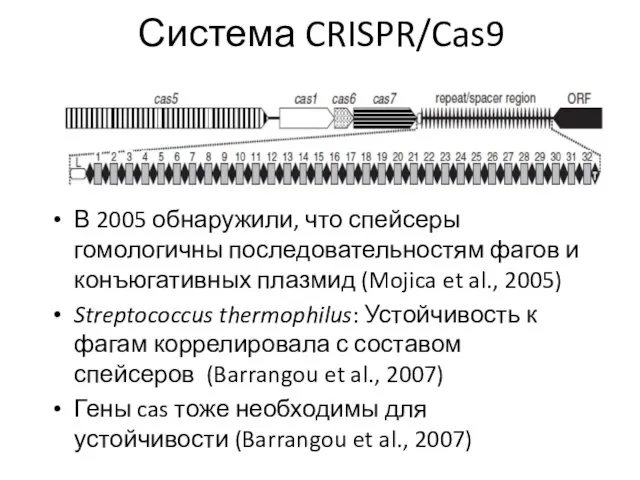
- 81. Система CRISPR/Cas9 В 2005 обнаружили, что спейсеры гомологичны последовательностям фагов и конъюгативных плазмид (Mojica et al.,
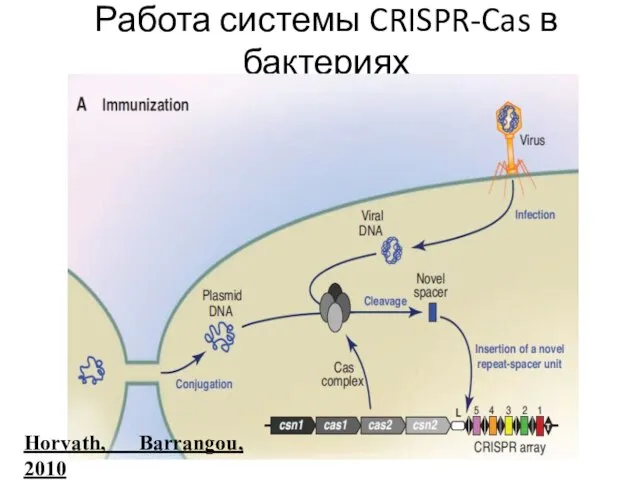
- 82. Работа системы CRISPR-Cas в бактериях Horvath, Barrangou, 2010
- 83. Čermák et al., 2015
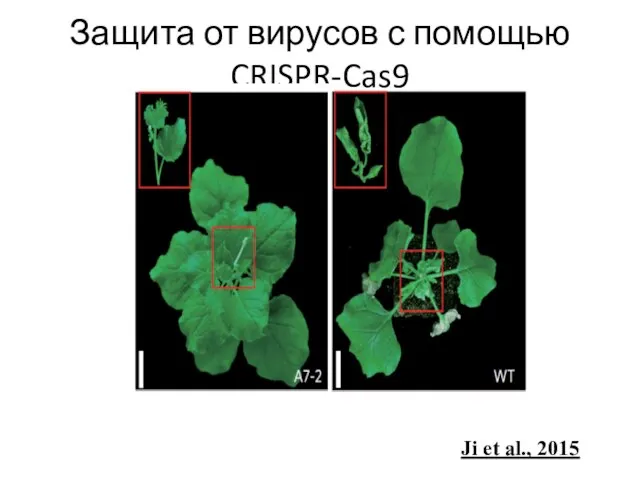
- 84. Защита от вирусов с помощью CRISPR-Cas9 Ji et al., 2015
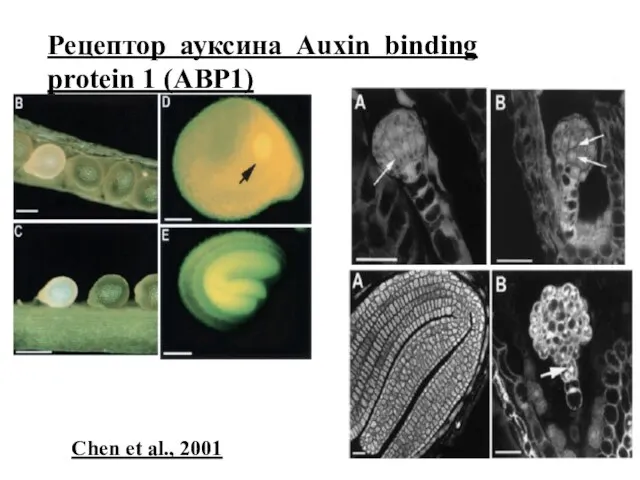
- 85. Рецептор ауксина Auxin binding protein 1 (ABP1) Chen et al., 2001
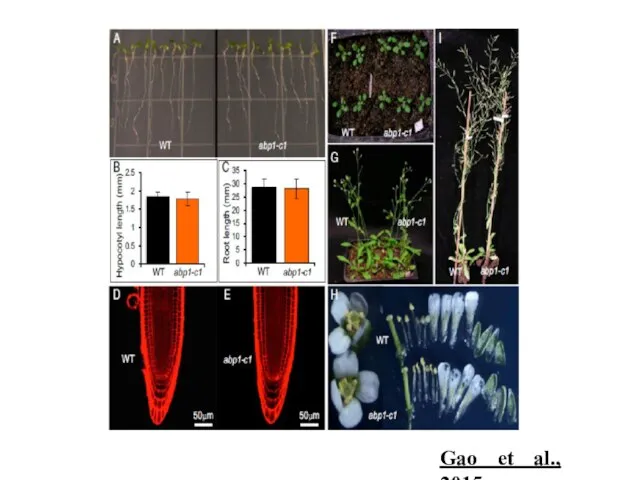
- 86. Gao et al., 2015
- 88. Скачать презентацию






































 LAMARK (1)
LAMARK (1) Основные виды питательных веществ и их значение в питании человека
Основные виды питательных веществ и их значение в питании человека Презентация на тему Отряд Пингвины
Презентация на тему Отряд Пингвины  Семейство Крестоцветные
Семейство Крестоцветные Презентация на тему Класс Паукообразные
Презентация на тему Класс Паукообразные  Старение и инсулиновый сигналинг у человека
Старение и инсулиновый сигналинг у человека Биофизика мышечного сокращения
Биофизика мышечного сокращения Устойчивость микроорганизмов к воздействию тепла и штатные процессы тепловой консервации
Устойчивость микроорганизмов к воздействию тепла и штатные процессы тепловой консервации Изучение эффекта синергизма между вирусным и бактериальным агентами при борьбе с непарным шелкопрядом
Изучение эффекта синергизма между вирусным и бактериальным агентами при борьбе с непарным шелкопрядом Взгляды и гипотезы о происхождении жизни на Земле Урок биологии в 9классе Учитель биологии МОУСОШ №2
Взгляды и гипотезы о происхождении жизни на Земле Урок биологии в 9классе Учитель биологии МОУСОШ №2  Свойства и функции белков
Свойства и функции белков Минеральное (корневое) питание растений
Минеральное (корневое) питание растений Выполнил: Мустафин Тимур МОУ СОШ №156 2а класс Екатеринбург, 2011 г
Выполнил: Мустафин Тимур МОУ СОШ №156 2а класс Екатеринбург, 2011 г  Сны и сновидения. Выполнили: Ученицы 8 «В» класса Лицея №1 им. Г.С.Титова Барткив София и Сысоева Елизавета
Сны и сновидения. Выполнили: Ученицы 8 «В» класса Лицея №1 им. Г.С.Титова Барткив София и Сысоева Елизавета Многообразие стеблей и видоизменения побегов.
Многообразие стеблей и видоизменения побегов. Витамины - укрепляют организм
Витамины - укрепляют организм Юные исследователи
Юные исследователи Презентация на тему Синдром Шерешевского-Тернера
Презентация на тему Синдром Шерешевского-Тернера  Физиологические основы голода и насщыения. Регуляция аппетита
Физиологические основы голода и насщыения. Регуляция аппетита Биосферный центральный заповедник
Биосферный центральный заповедник Леса Азии
Леса Азии Переломы Работа учащихся Даньковской основной школы
Переломы Работа учащихся Даньковской основной школы Пищеварительная система
Пищеварительная система Этапы урока. Организация начала занятия. Проверка выполнения домашнего задания. Подготовка к усвоению новых знаний Изучение
Этапы урока. Организация начала занятия. Проверка выполнения домашнего задания. Подготовка к усвоению новых знаний Изучение  Земноводные. Амфибии
Земноводные. Амфибии Что такое онтогенез?
Что такое онтогенез? Презентация Загрязнение атмосферы
Презентация Загрязнение атмосферы Список препаратов набор 1
Список препаратов набор 1