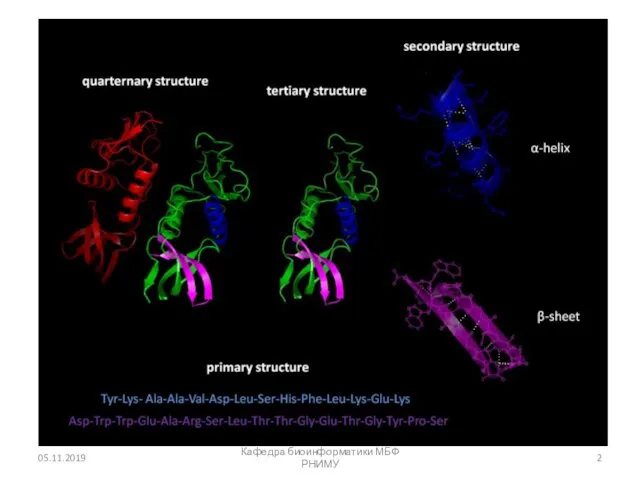
Содержание
- 2. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
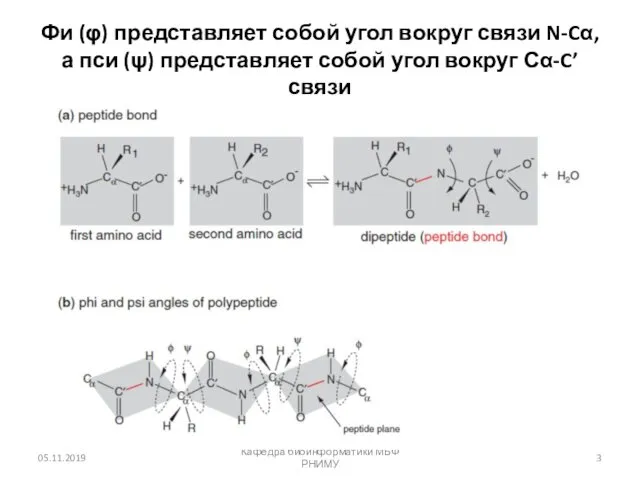
- 3. Фи (φ) представляет собой угол вокруг связи N-Cα, а пси (ψ) представляет собой угол вокруг Сα-C’
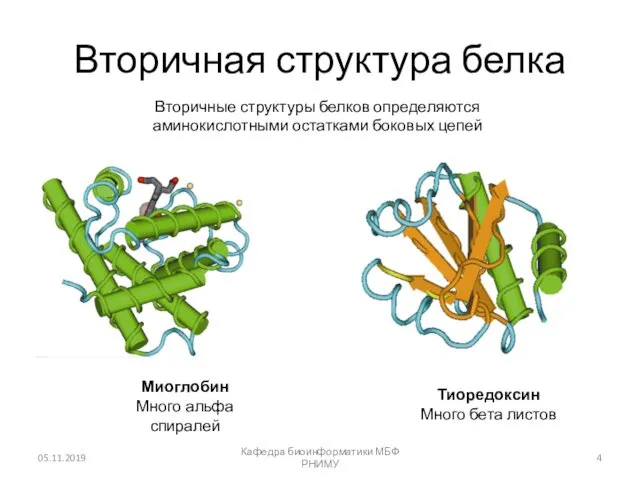
- 4. Вторичная структура белка 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Миоглобин Много альфа спиралей Тиоредоксин Много бета листов
- 5. Альфа спираль (α-helix) Элемент вторичной структуры белков, который имеет форму правозакрученой винтовой линии и в котором
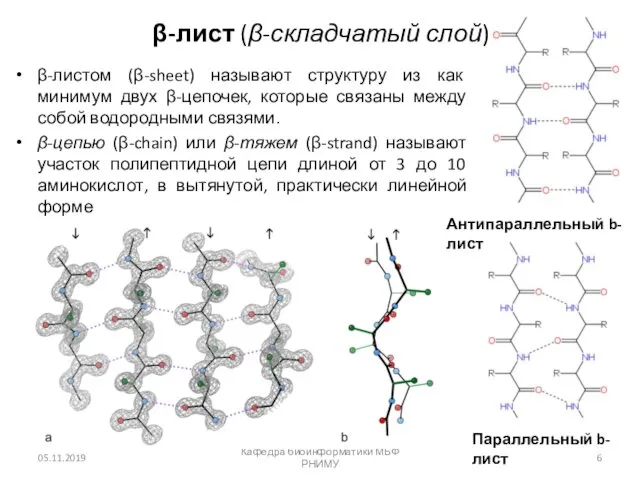
- 6. β-лист (β-складчатый слой) β-листом (β-sheet) называют структуру из как минимум двух β-цепочек, которые связаны между собой
- 7. Диаграмма Рамачандрана отображает пси и фи углы для каждой аминокислоты белка (за исключением пролина, а в
- 8. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
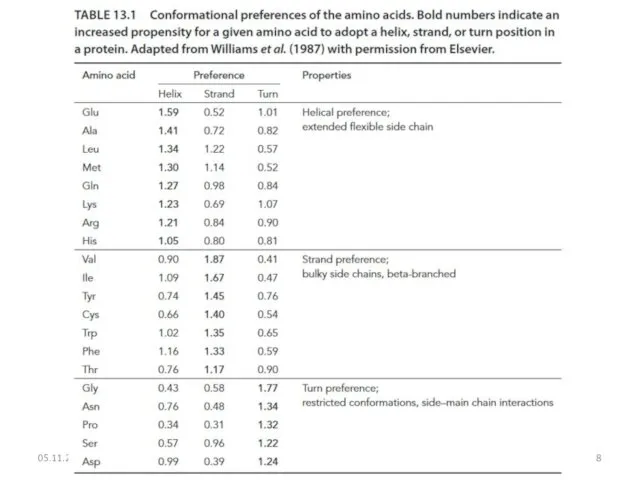
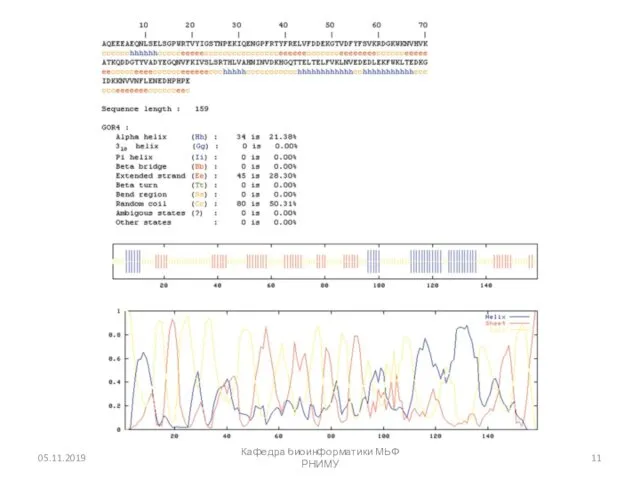
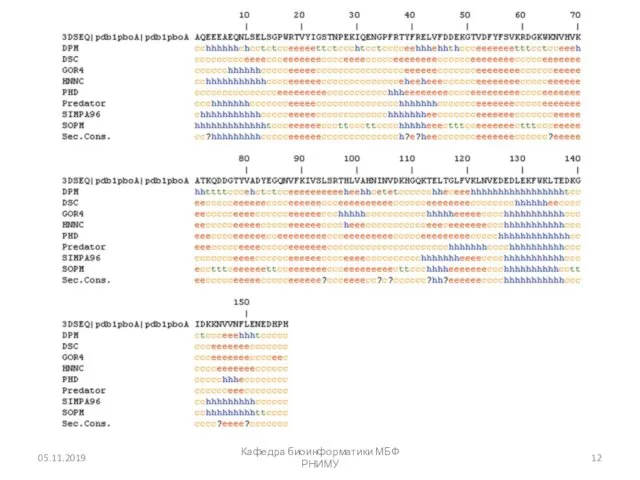
- 9. Предсказание вторичной структуры белка Chou и Fasman (1974) разработали алгоритм, основанный на частотах аминокислот, обнаруженных в
- 10. On-line сервисы по прогнозу вторичной структуры доступные в интернете 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 11. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 12. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 13. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
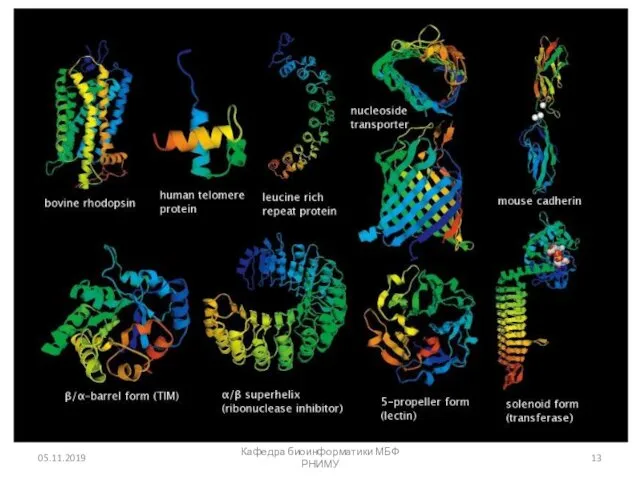
- 14. Третичная структура белка: сворачивание белка Основные подходы: [1] Экспериментальное определение (Рентгеновская кристаллография, ЯМР) [2] Прогнозирование сравнительное
- 15. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
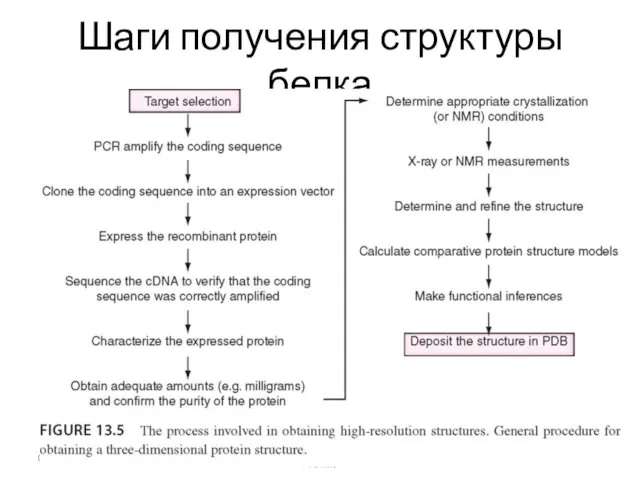
- 16. Шаги получения структуры белка 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
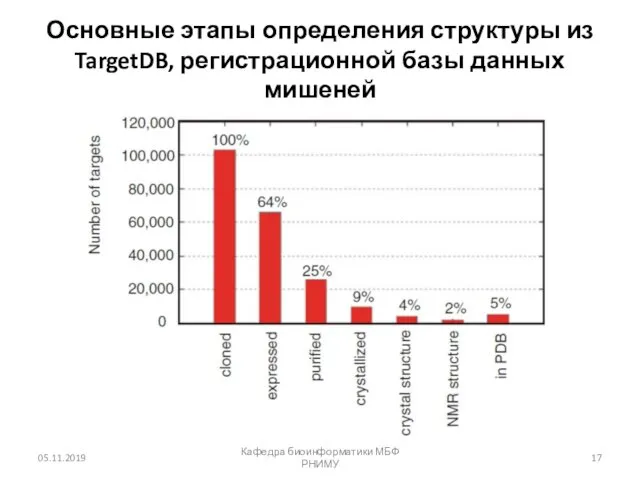
- 17. Основные этапы определения структуры из TargetDB, регистрационной базы данных мишеней 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 18. Приоритеты для выбора мишени для определения ее структуры Исторические - небольшие, растворимые, распространенные белки (гемоглобин, цитохромы
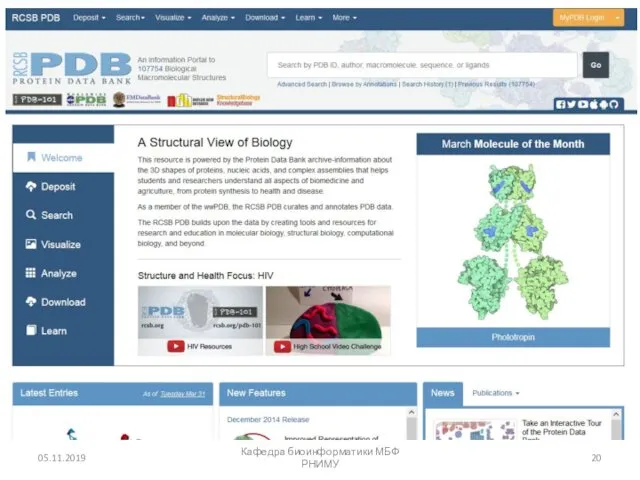
- 19. The Protein Data Bank (PDB) PDB является основным хранилищем для белковых структур Основан в 1971 году
- 20. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
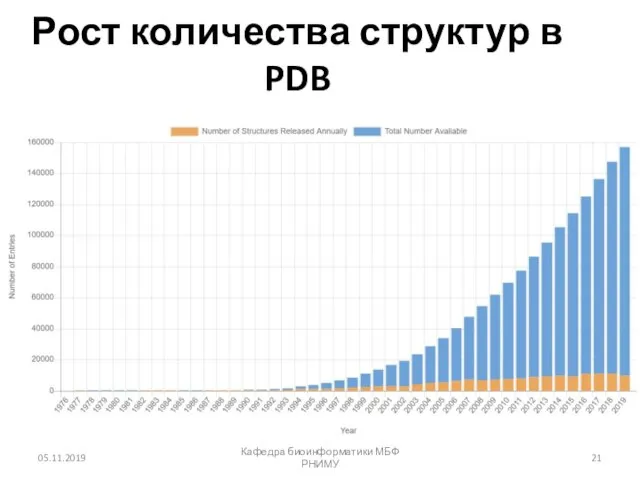
- 21. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Рост количества структур в PDB
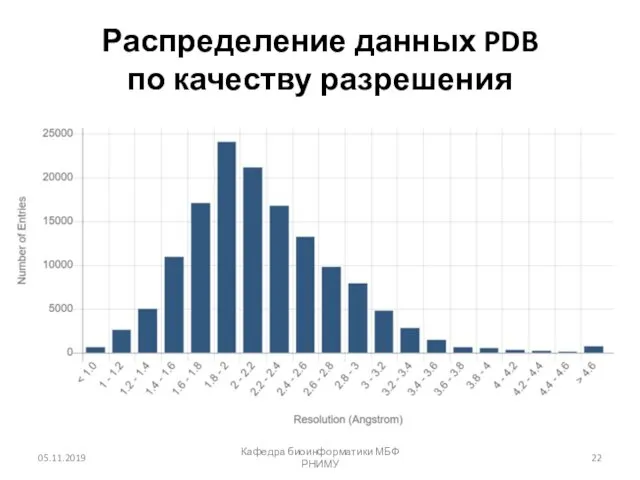
- 22. Распределение данных PDB по качеству разрешения 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
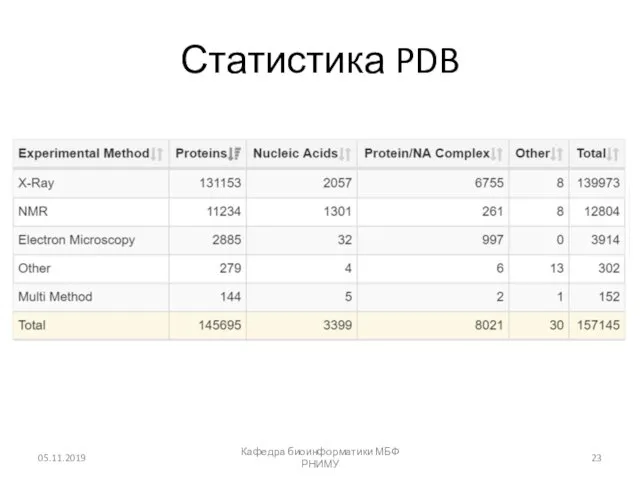
- 23. Статистика PDB 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
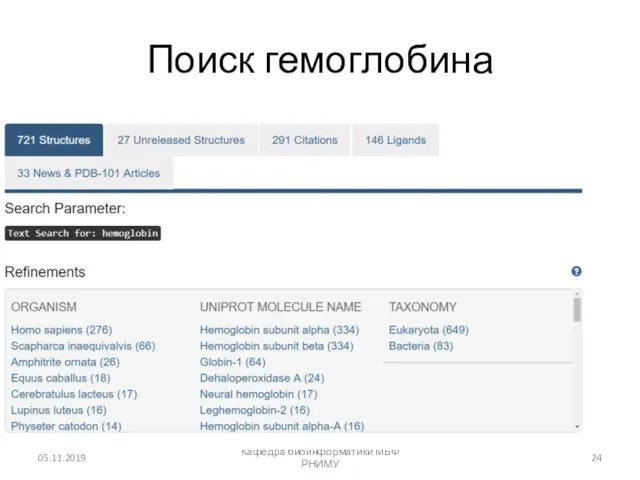
- 24. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Поиск гемоглобина
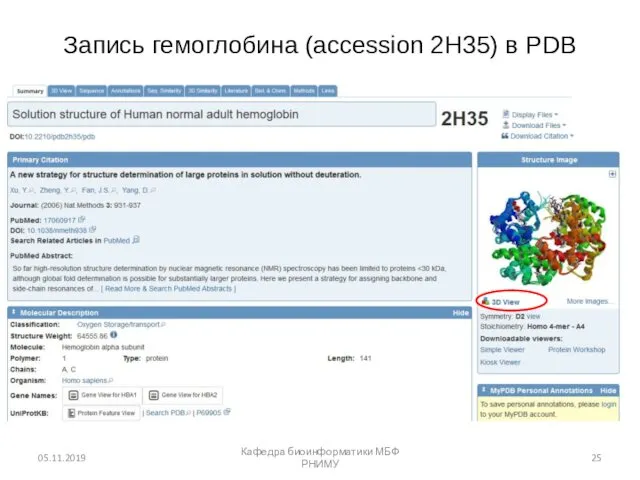
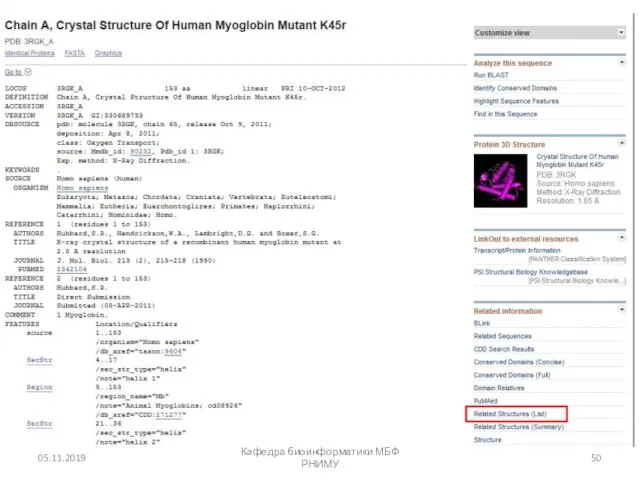
- 25. Запись гемоглобина (accession 2H35) в PDB 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
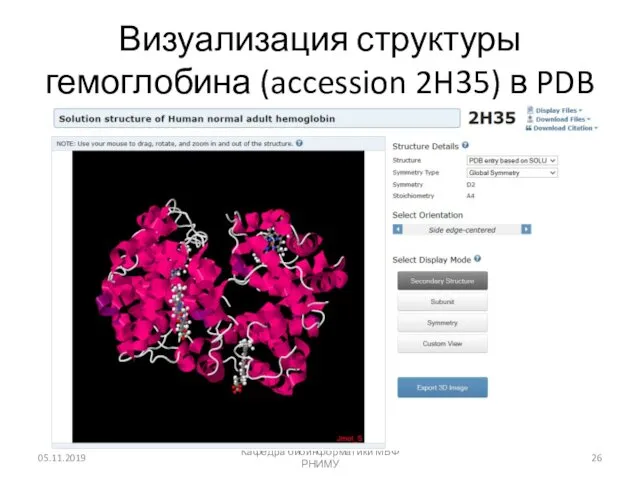
- 26. Визуализация структуры гемоглобина (accession 2H35) в PDB 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 27. Инструменты для интерактивной визуализация белковых структур 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
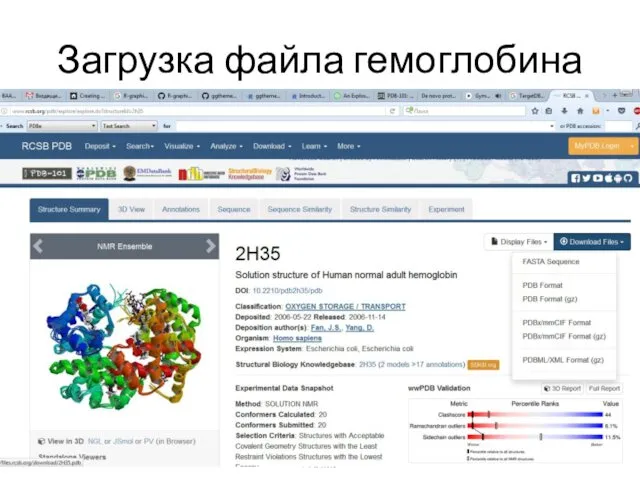
- 28. Загрузка файла гемоглобина 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 29. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Структура PDB файла «TITLE» – в этом разделе указывается название модели
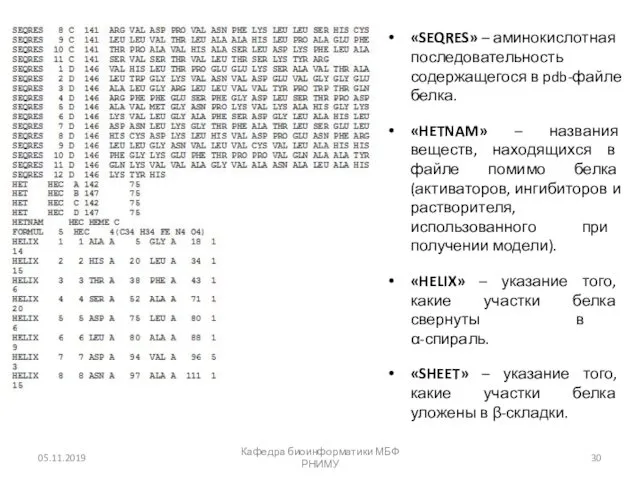
- 30. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ «SEQRES» – аминокислотная последовательность содержащегося в pdb-файле белка. «HETNAM» – названия
- 31. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ http://www.wwpdb.org/documentation/file-format-content/format33/v3.3.html «SITE» - описывает остатки, содержащие каталитические, взаимодействующие с кофактором, анти-кодона,
- 32. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Protein Data Bank Swiss-Prot, NCBI, EMBL CATH, Dali, SCOP, FSSP Пути
- 33. CATH: http://www.cathdb.info/ 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
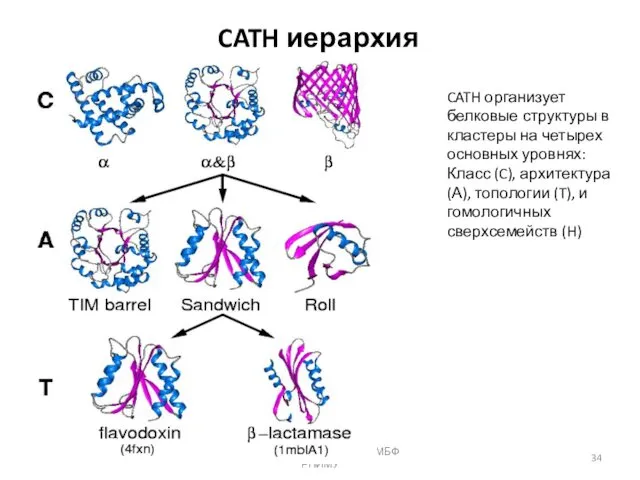
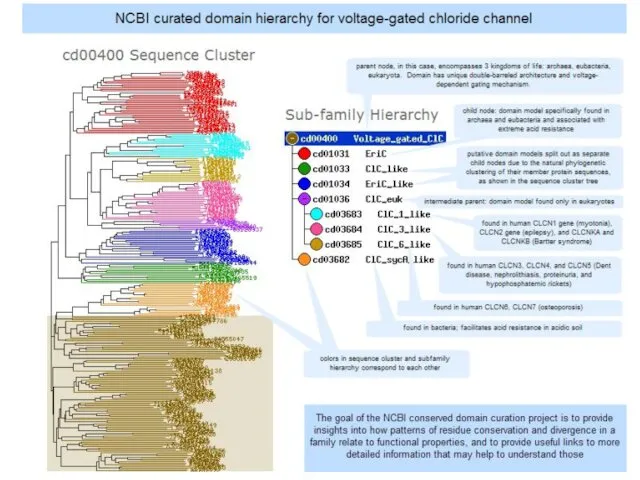
- 34. CATH иерархия 05.11.2019 Кафедра биоинформатики МБФ РНИМУ CATH организует белковые структуры в кластеры на четырех основных
- 35. Dali Обеспечивает классификацию всех структур из PDB и описание семейств белковых последовательностей, связанных с представительными белков
- 36. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 37. Доступ к PDB через NCBI Перейти на сайт БД Structure, на домашней странице NCBI Через Entrez
- 38. БД Structure, на домашней странице NCBI 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 39. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 40. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 41. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
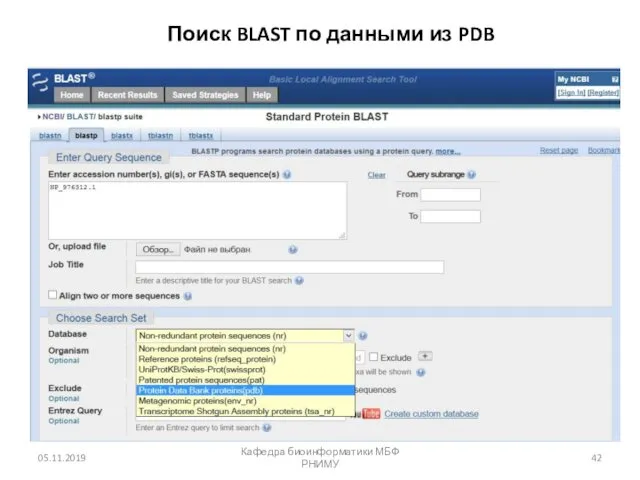
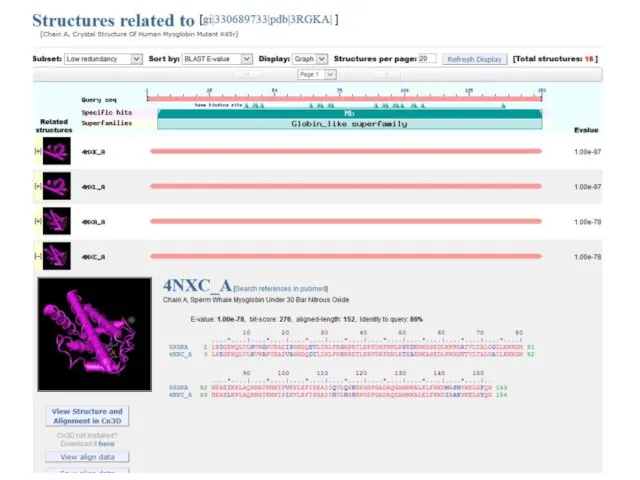
- 42. Поиск BLAST по данными из PDB 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 43. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Поиск BLAST по данными из PDB
- 44. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
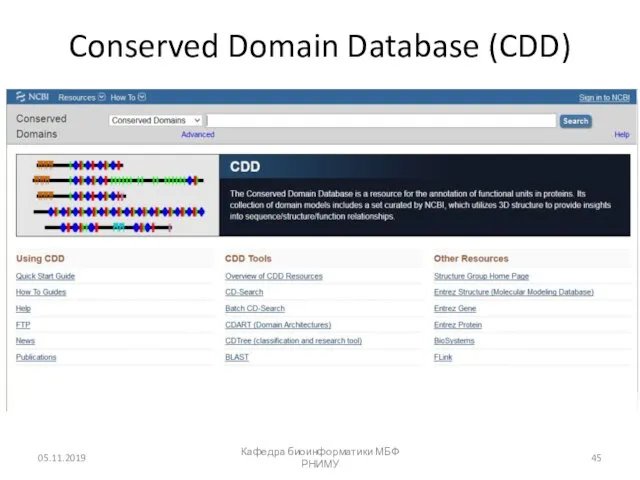
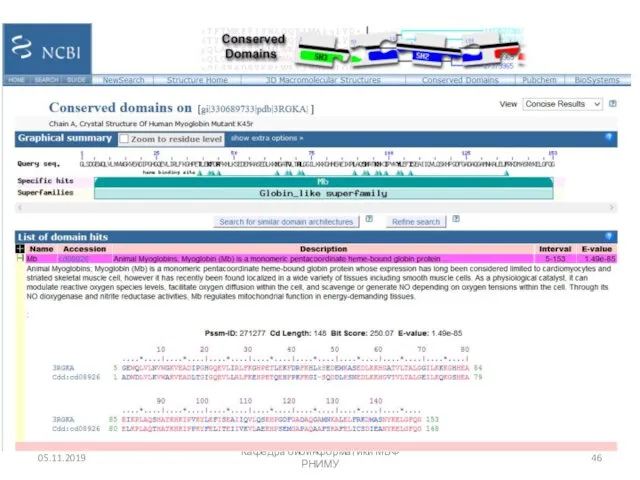
- 45. Conserved Domain Database (CDD) 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 46. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 47. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 48. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 49. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 50. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 51. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 52. БД Structure, на домашней странице NCBI 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
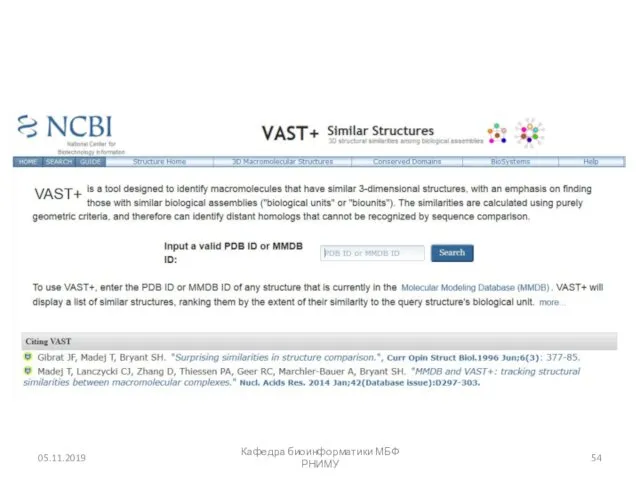
- 53. VAST Vector Alignment Search Tool - является компьютерным алгоритмом, разработанным в NCBI и используется для выявления
- 54. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 55. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
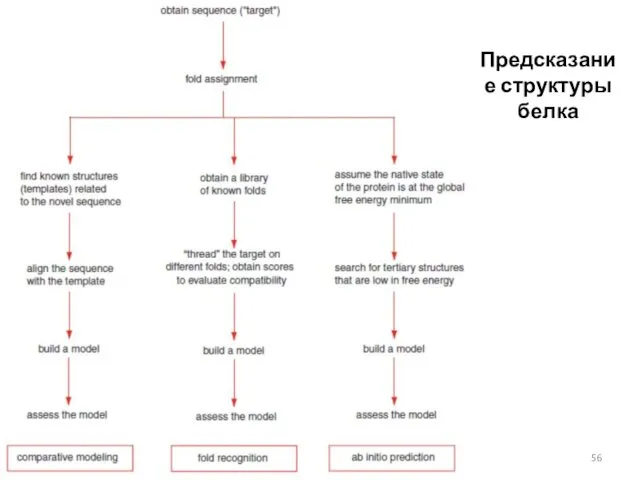
- 56. Предсказание структуры белка 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
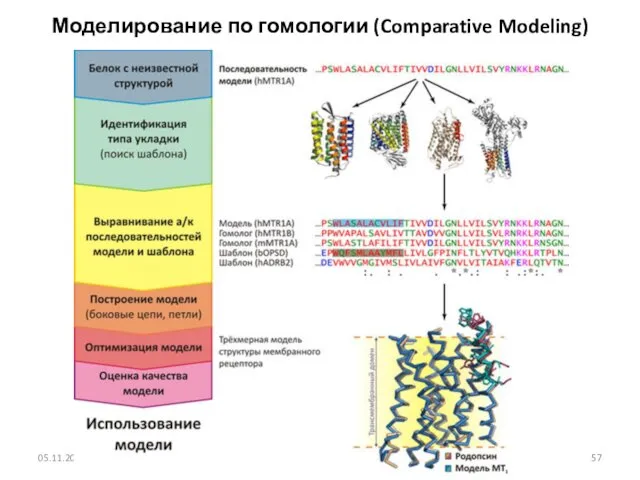
- 57. Моделирование по гомологии (Comparative Modeling) 05.11.2019 Кафедра биоинформатики МБФ РНИМУ
- 58. Есть несколько основных типов ошибок, которые происходят при моделировании по гомологии Ошибки в упаковке боковой цепи
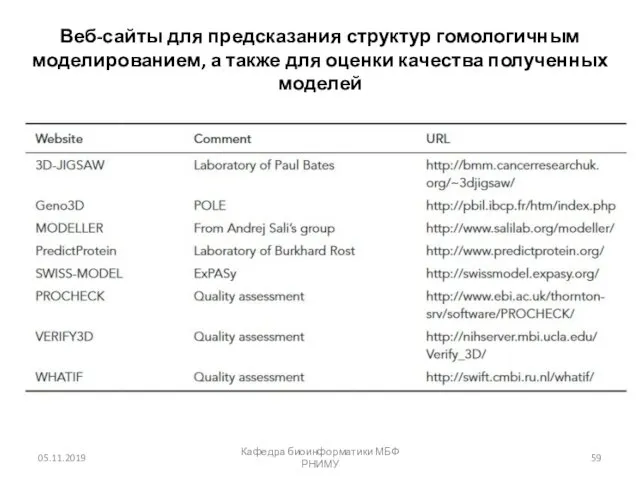
- 59. Веб-сайты для предсказания структур гомологичным моделированием, а также для оценки качества полученных моделей 05.11.2019 Кафедра биоинформатики
- 60. Распознавание фолдинга (Threading) Хотя в настоящее время в PDB более 100 000 записей, может быть лишь
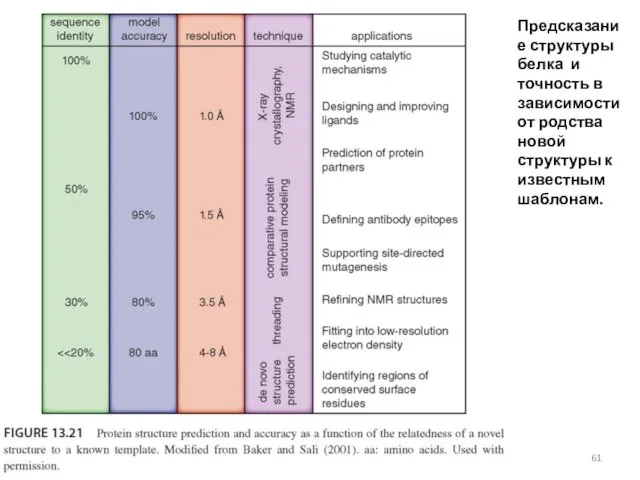
- 61. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Предсказание структуры белка и точность в зависимости от родства новой структуры
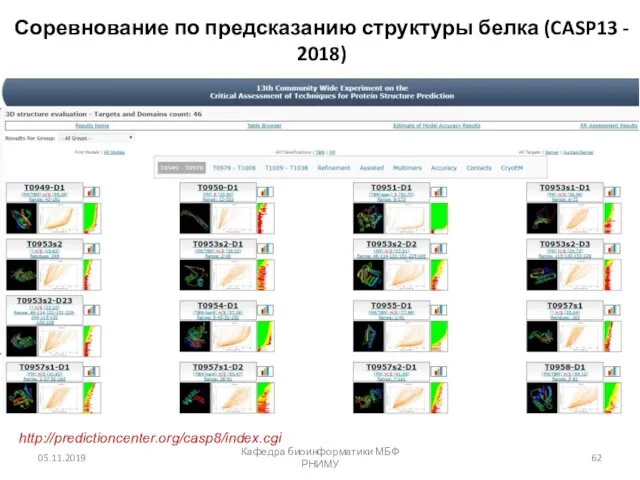
- 62. Соревнование по предсказанию структуры белка (CASP13 - 2018) 05.11.2019 Кафедра биоинформатики МБФ РНИМУ http://predictioncenter.org/casp8/index.cgi
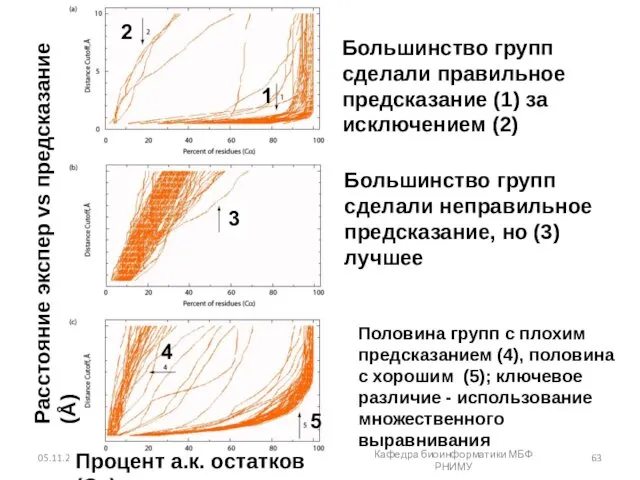
- 63. 05.11.2019 Кафедра биоинформатики МБФ РНИМУ Процент а.к. остатков (Cα) Расстояние экспер vs предсказание (Å) Большинство групп
- 65. Скачать презентацию




![Третичная структура белка: сворачивание белка Основные подходы: [1] Экспериментальное определение (Рентгеновская](/_ipx/f_webp&q_80&fit_contain&s_1440x1080/imagesDir/jpg/498360/slide-13.jpg)




















 Птицы – особенности класса Птицы представляют собой хорошо обособленную группу теплокровных позвоночных животных. Одним из наиб
Птицы – особенности класса Птицы представляют собой хорошо обособленную группу теплокровных позвоночных животных. Одним из наиб Ассортиментная ведомость и нормы посадки растений
Ассортиментная ведомость и нормы посадки растений Сердечно-сосудистая система человека. Сердце
Сердечно-сосудистая система человека. Сердце Презентация на тему "Генетика пола. Наследование, сцепленное с полом" - скачать презентации по Биологии
Презентация на тему "Генетика пола. Наследование, сцепленное с полом" - скачать презентации по Биологии Анатомия и топография черепных нервов
Анатомия и топография черепных нервов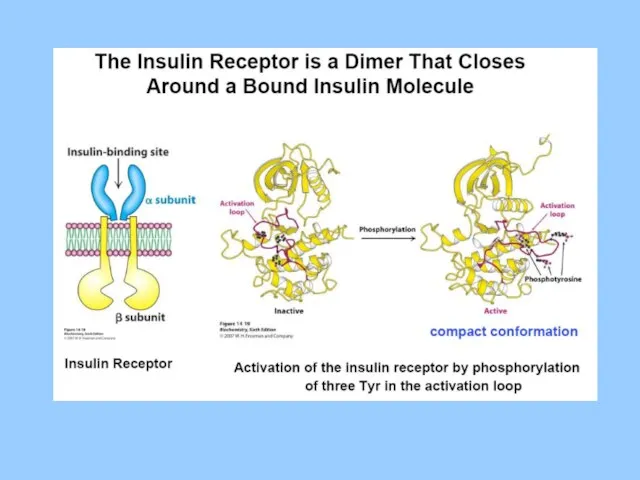 The Insulin Receptor
The Insulin Receptor Систематика птиц
Систематика птиц Эмбриология. Дробление
Эмбриология. Дробление Амилоидоздың патогенезі
Амилоидоздың патогенезі Деление клеток
Деление клеток Human Body
Human Body Скорость прорастания семян гороха в зависимости от температуры
Скорость прорастания семян гороха в зависимости от температуры Мейоз. Механизм мейоза
Мейоз. Механизм мейоза Тварини минулого
Тварини минулого ,,Чудо-Юдо”
,,Чудо-Юдо” Вставить слова в общей характеристике: Надкласс рыбы – это самый многочисленный надкласс подтипа … Рыб разделяют на класс …. и класс … Среда обитания рыб - … Форма тела у них - … Тело покрыто … и … Органы дыхания - … Имеют …. круг кровообращения, сер
Вставить слова в общей характеристике: Надкласс рыбы – это самый многочисленный надкласс подтипа … Рыб разделяют на класс …. и класс … Среда обитания рыб - … Форма тела у них - … Тело покрыто … и … Органы дыхания - … Имеют …. круг кровообращения, сер Самые длинные водные животные Живые существа, обитающие в воде, очень и очень разнообразны по форме, размерам, образу жизни, способу питания и многим другим критериям. Длина тела – один из показателей, отличающий обитателей морей и океанов друг от
Самые длинные водные животные Живые существа, обитающие в воде, очень и очень разнообразны по форме, размерам, образу жизни, способу питания и многим другим критериям. Длина тела – один из показателей, отличающий обитателей морей и океанов друг от  Загадка человека. Тема 2
Загадка человека. Тема 2 Общие сведения о нервной системе
Общие сведения о нервной системе Зір людини. Офтальмологія
Зір людини. Офтальмологія Презентация на тему "Щитовидная железа" - скачать бесплатно презентации по Биологии
Презентация на тему "Щитовидная железа" - скачать бесплатно презентации по Биологии Дефекты и болезни хлеба. Требования к качеству хлеба
Дефекты и болезни хлеба. Требования к качеству хлеба Семейство Розоцветные
Семейство Розоцветные Сцепленное наследование признаков Закон Томаса Моргана 9 класс Подготовила учитель МОУ СОШ №3 с. Китаевского Липадкина Г.И.
Сцепленное наследование признаков Закон Томаса Моргана 9 класс Подготовила учитель МОУ СОШ №3 с. Китаевского Липадкина Г.И. Найдите лишнее растение
Найдите лишнее растение Интегрированный урок в 6 классе по биологии , русскому языку и литературе
Интегрированный урок в 6 классе по биологии , русскому языку и литературе Клуб увлеченных мам представляет Мое тело
Клуб увлеченных мам представляет Мое тело Красная книга России
Красная книга России