Алгоритмы и методы глобального выравнивания последовательностей. Множественное выравнивание. Рекомбинационный анализ
Содержание
- 2. Выравнивание последовательностей —метод, основанный на размещении двух или более последовательностей ДНК, РНК или белков друг под
- 3. Глобальное выравнивание Полагается, что последовательности обладают достаточным сходством по всей длине Можно разделить на: попарное (выравнивание
- 6. Алгоритм Нидлмана — Вунша Был предложен в 1970 году является примером динамического программирования, и он оказался
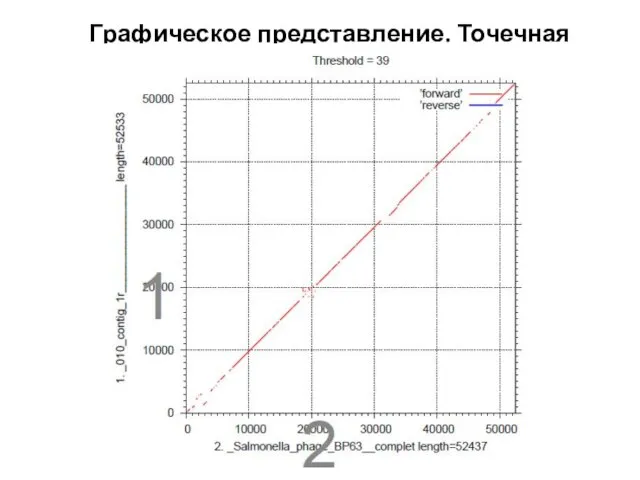
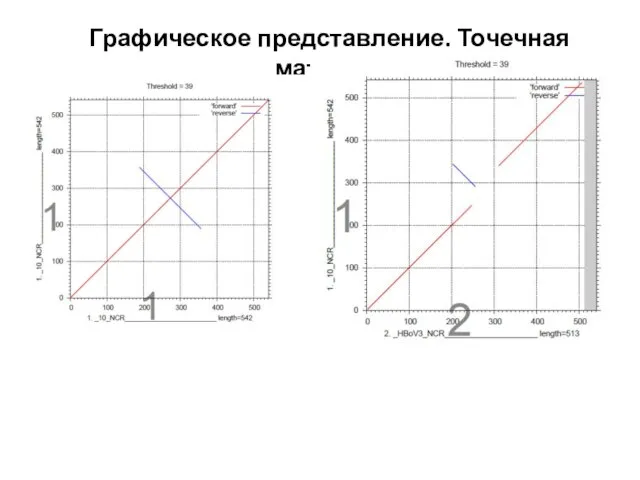
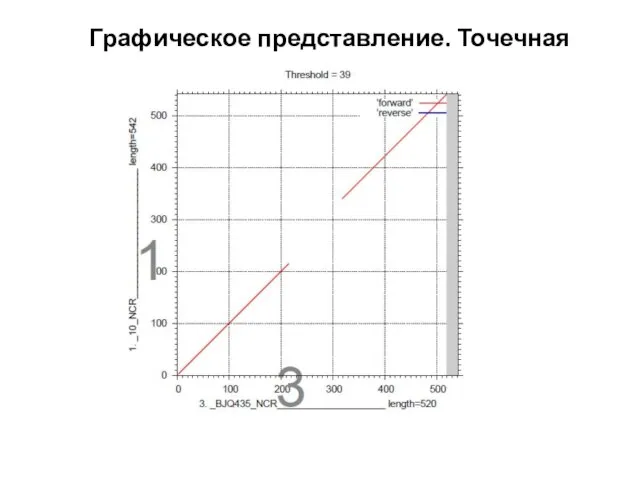
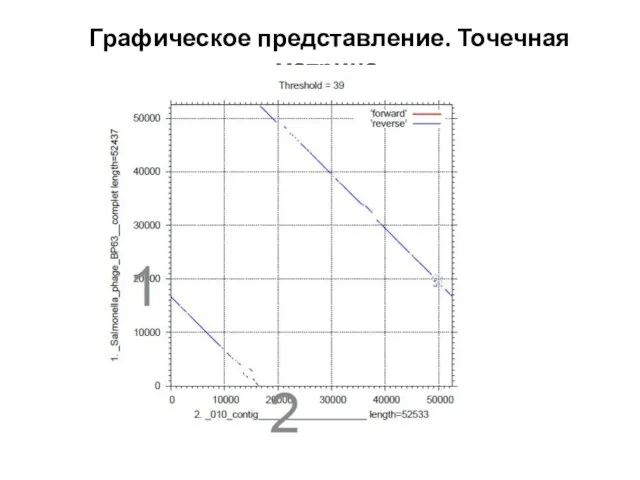
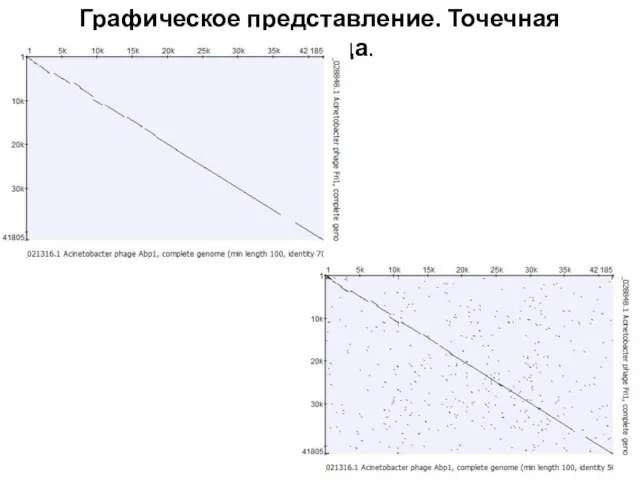
- 7. Графическое представление. Точечная матрица.
- 8. Графическое представление. Точечная матрица.
- 9. Графическое представление. Точечная матрица.
- 10. Графическое представление. Точечная матрица.
- 11. Графическое представление. Точечная матрица.
- 12. Графическое представление. Точечная матрица.
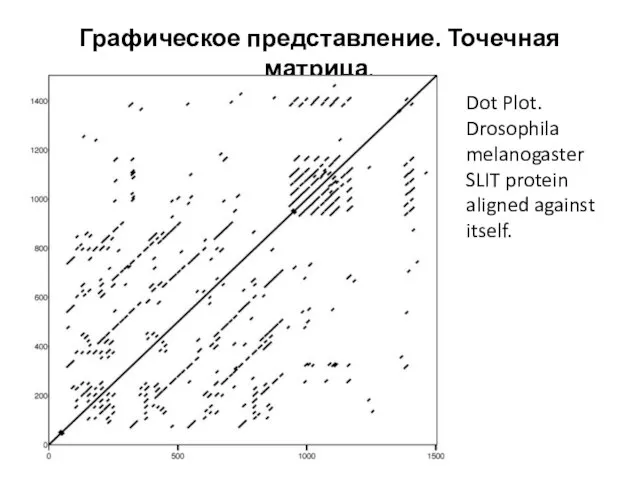
- 13. Графическое представление. Точечная матрица. Dot Plot. Drosophila melanogaster SLIT protein aligned against itself.
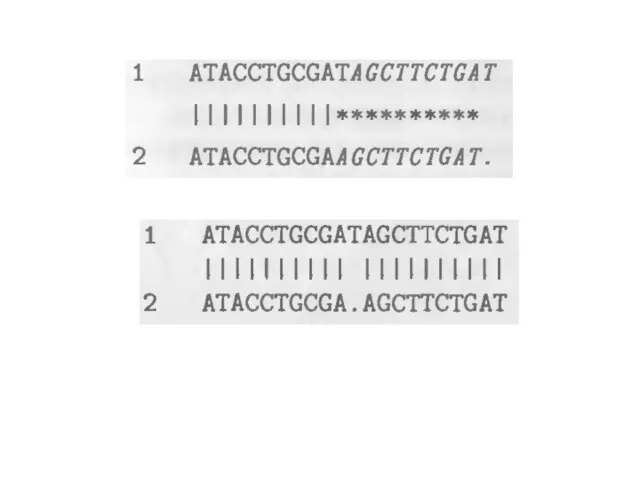
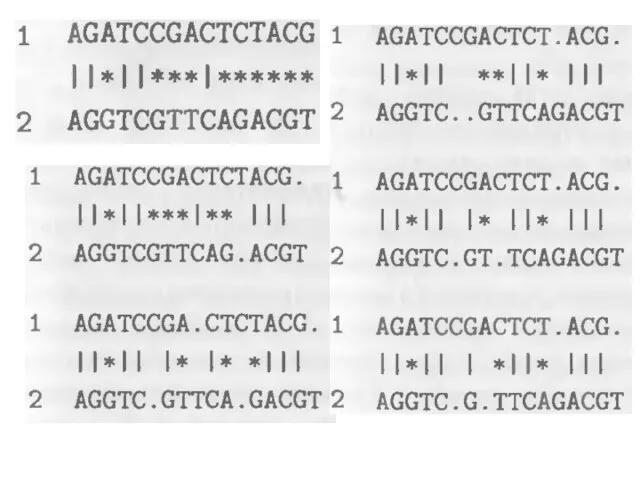
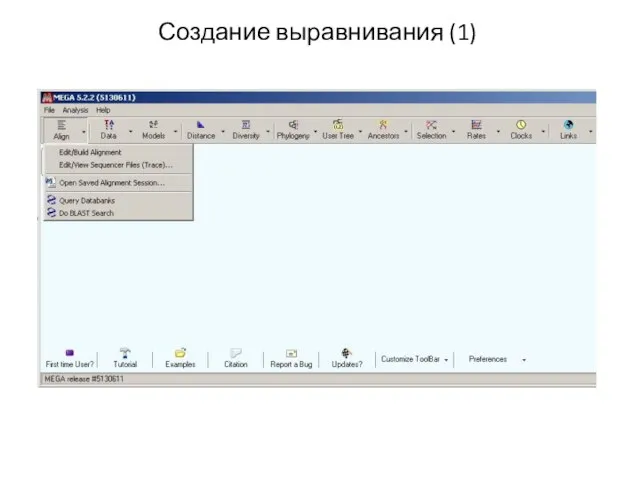
- 14. Создание выравнивания (1)
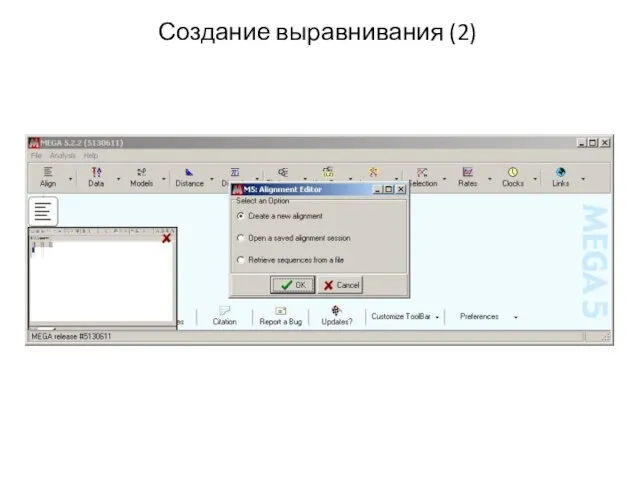
- 15. Создание выравнивания (2)
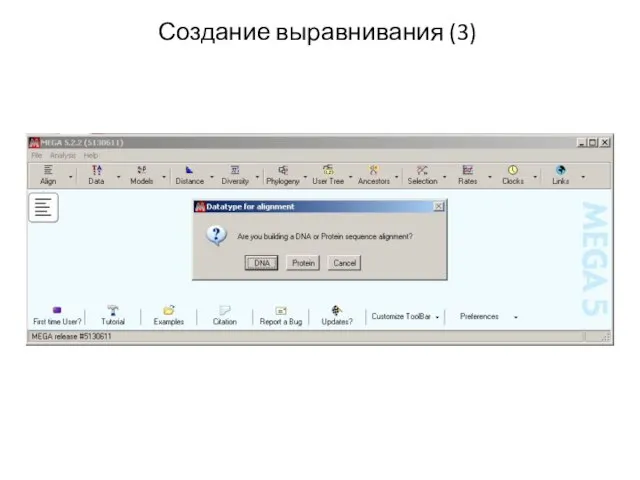
- 16. Создание выравнивания (3)
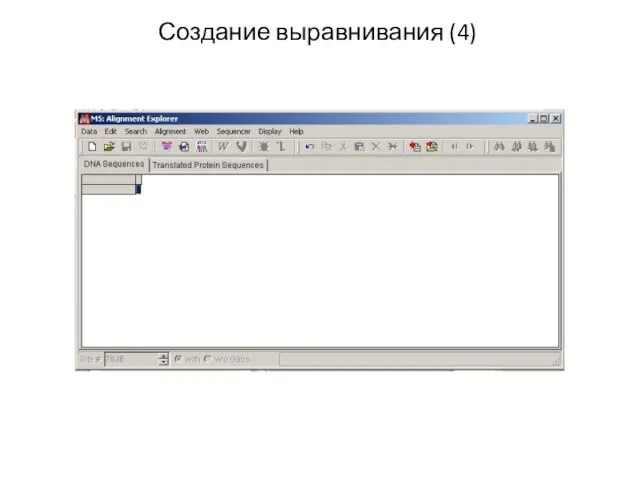
- 17. Создание выравнивания (4)
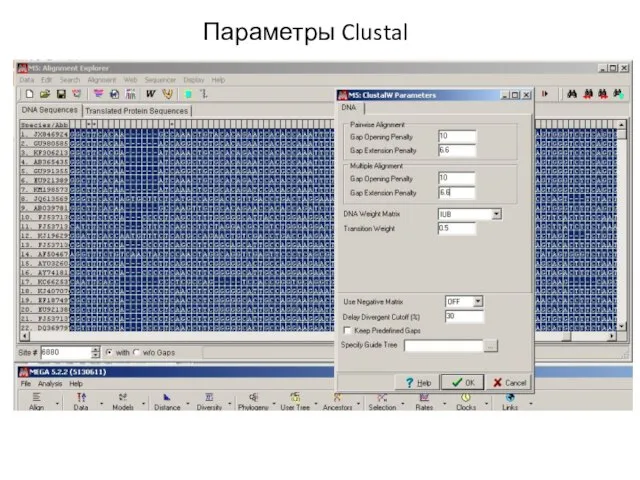
- 18. Параметры Clustal
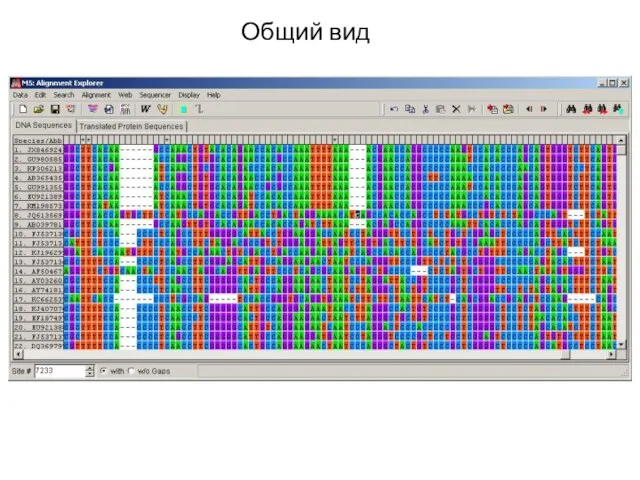
- 19. Общий вид
- 20. https://mafft.cbrc.jp/ MAFFT http://www.tcoffee.org/ T-Coffee
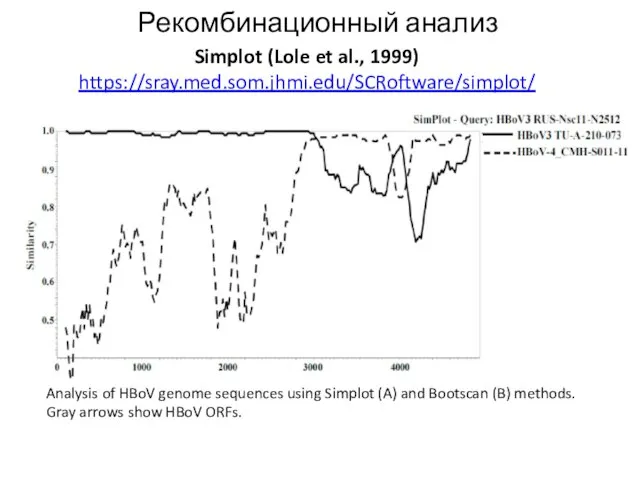
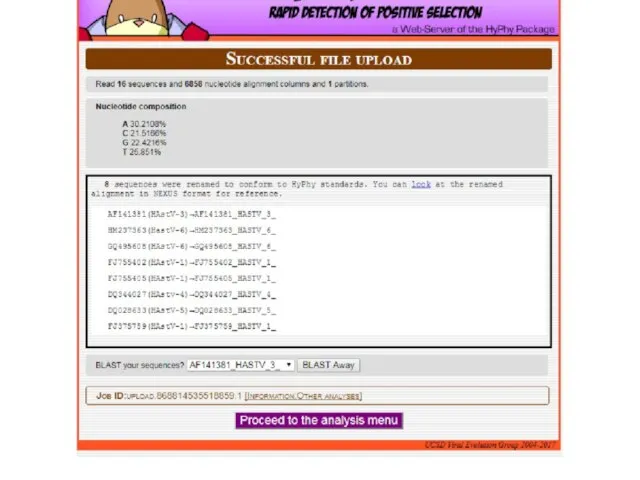
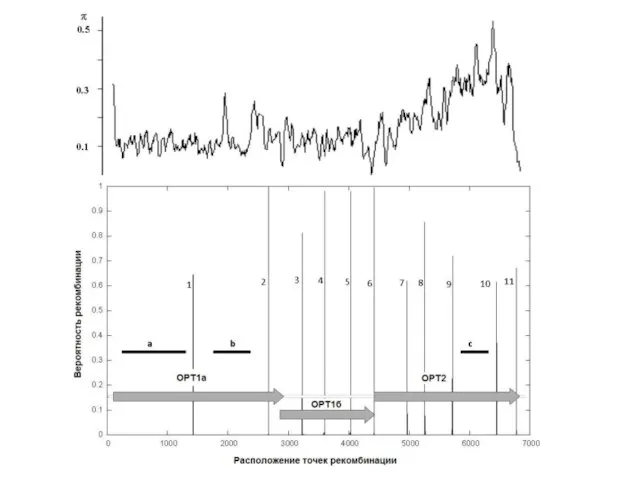
- 21. Рекомбинационный анализ Simplot (Lole et al., 1999) https://sray.med.som.jhmi.edu/SCRoftware/simplot/ Analysis of HBoV genome sequences using Simplot (A)
- 22. Рекомбинационный анализ Simplot (Lole et al., 1999) https://sray.med.som.jhmi.edu/SCRoftware/simplot/ Analysis of HBoV genome sequences using Simplot (A)
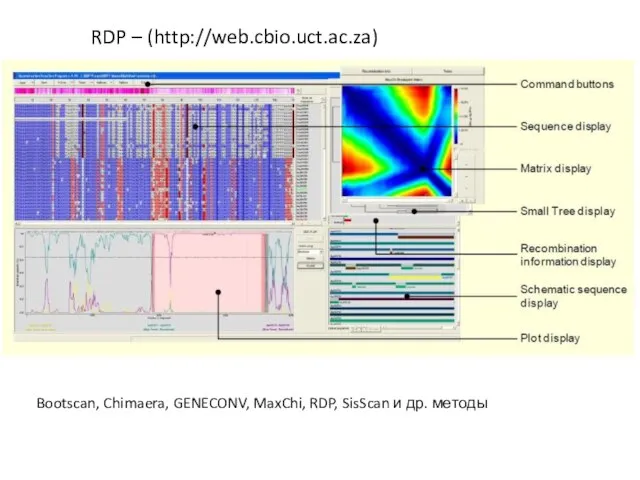
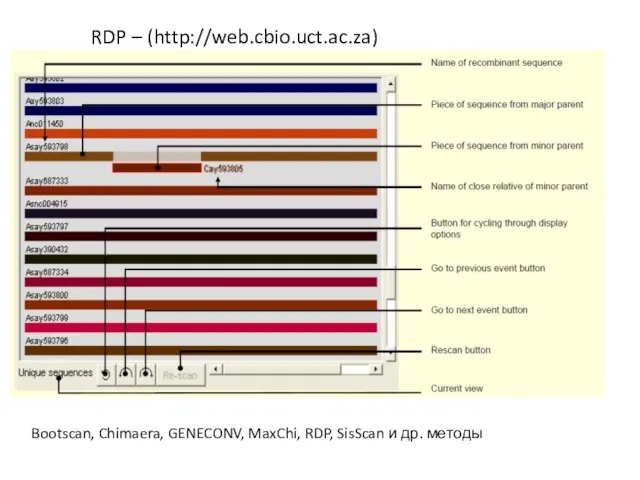
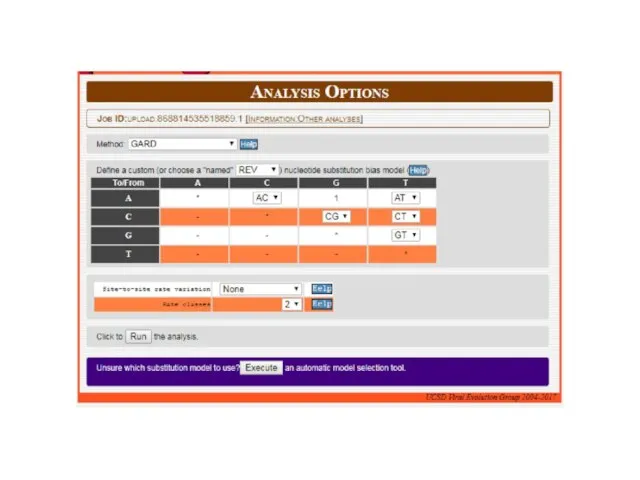
- 23. Bootscan, Chimaera, GENECONV, MaxChi, RDP, SisScan и др. методы RDP – (http://web.cbio.uct.ac.za)
- 24. Bootscan, Chimaera, GENECONV, MaxChi, RDP, SisScan и др. методы RDP – (http://web.cbio.uct.ac.za)
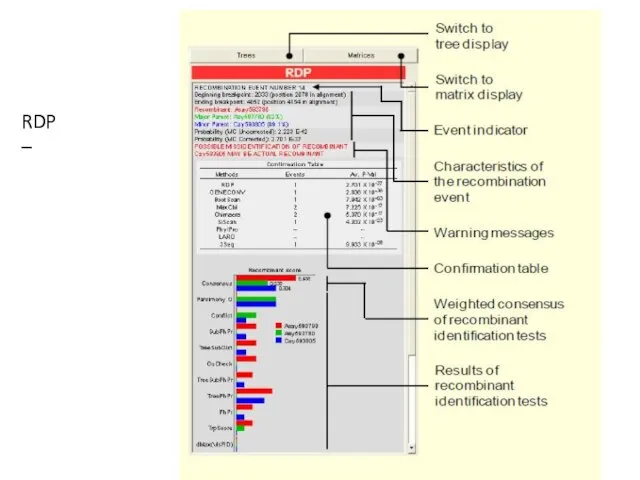
- 25. RDP –
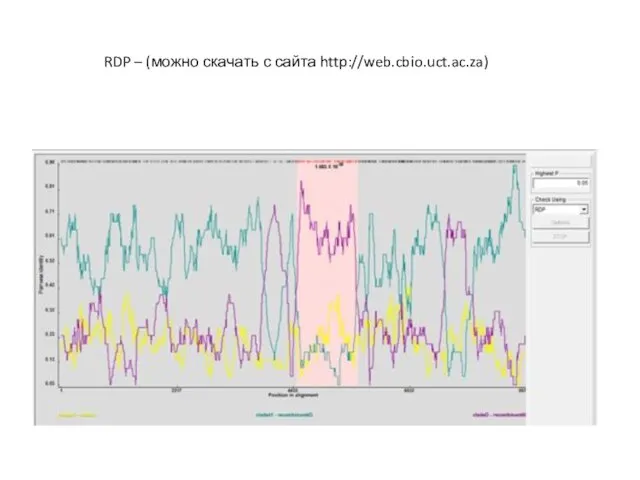
- 26. RDP – (можно скачать с сайта http://web.cbio.uct.ac.za)
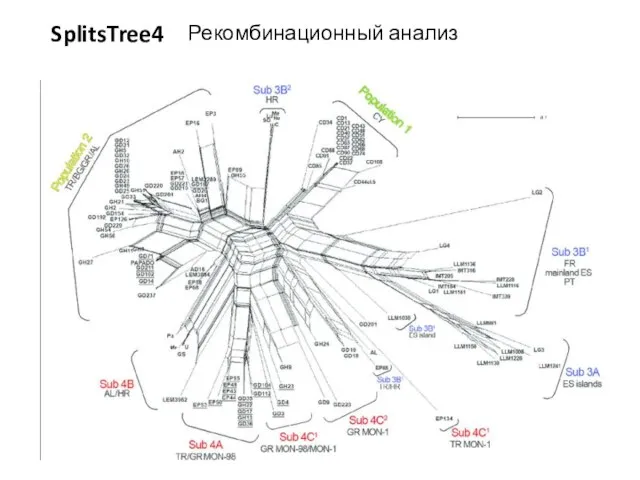
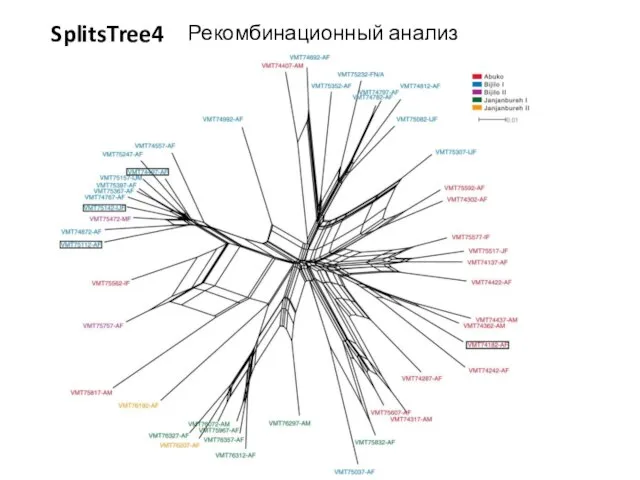
- 31. Рекомбинационный анализ SplitsTree4
- 32. Рекомбинационный анализ SplitsTree4
- 34. Скачать презентацию






 Грузовые операции на необорудованном берегу
Грузовые операции на необорудованном берегу Русский язык
Русский язык Measurement of liquid level using a reflex-radar level meter
Measurement of liquid level using a reflex-radar level meter Виды холодильного оборудования розничной торговли
Виды холодильного оборудования розничной торговли Кузнецкий и Канско-Ачинский угольные бассейны
Кузнецкий и Канско-Ачинский угольные бассейны ПРЕЗЕНТАЦИЯ ДОМАШНИЕ ПТИЦЫ
ПРЕЗЕНТАЦИЯ ДОМАШНИЕ ПТИЦЫ Боб Айгер
Боб Айгер Исполнение токовых цифровых защит. Микропроцессорные средства управления
Исполнение токовых цифровых защит. Микропроцессорные средства управления Металлические конструкции
Металлические конструкции 20141219_igra
20141219_igra Павлодар облысы агроөнеркәсіптік кешенінің төлқұжаты
Павлодар облысы агроөнеркәсіптік кешенінің төлқұжаты Организация грузовых перевозок бумаги в пачках и консервов по маршруту №2431
Организация грузовых перевозок бумаги в пачках и консервов по маршруту №2431 презентация Матвеева
презентация Матвеева 20 апреля. Знакомство с творчеством поэта Ю.Тувима. Чудеса
20 апреля. Знакомство с творчеством поэта Ю.Тувима. Чудеса Группа компаний Пенетрон-Россия. Материалы для строительства, ремонта, восстановления и гидроизоляции строительных конструкций
Группа компаний Пенетрон-Россия. Материалы для строительства, ремонта, восстановления и гидроизоляции строительных конструкций Цитология щитовидной железы
Цитология щитовидной железы Гражданство Латвии
Гражданство Латвии Modlitwa Jana Pawła II o powołania
Modlitwa Jana Pawła II o powołania Эко-архитектура общественных зданий
Эко-архитектура общественных зданий Оценка и совершенствование технологии капельного орошения картофеля на южных черноземах Северо-Казахстанской области
Оценка и совершенствование технологии капельного орошения картофеля на южных черноземах Северо-Казахстанской области Советские Женщины Великой Отечественной войны 1941 – 1945 годов
Советские Женщины Великой Отечественной войны 1941 – 1945 годов Animal husbandry
Animal husbandry креп-тех-1
креп-тех-1 Этюды Цветущий май
Этюды Цветущий май La conjugaison du présent de l’indicatif
La conjugaison du présent de l’indicatif Строение древнерусского храма
Строение древнерусского храма Совершенствование системы технического сервиса сельскохозяйственной техники (на примере Саратовской области)
Совершенствование системы технического сервиса сельскохозяйственной техники (на примере Саратовской области) Производство, передача и использование электроэнергии
Производство, передача и использование электроэнергии