Содержание
- 2. 70 Образование аминоацил-тРНК Aminoacyl tRNA synthetase for aspartic acid (Class II aaRS). It is a dimer
- 3. Состав рибосом Прокариоты Эукариоты Selected views of the 70S ribosome of E. coli as obtained by
- 4. 72 Строение рибосом A P E К анимации: 5S(50S) - желтый; 23S(50S) и 16S(30S)-красный; белки -
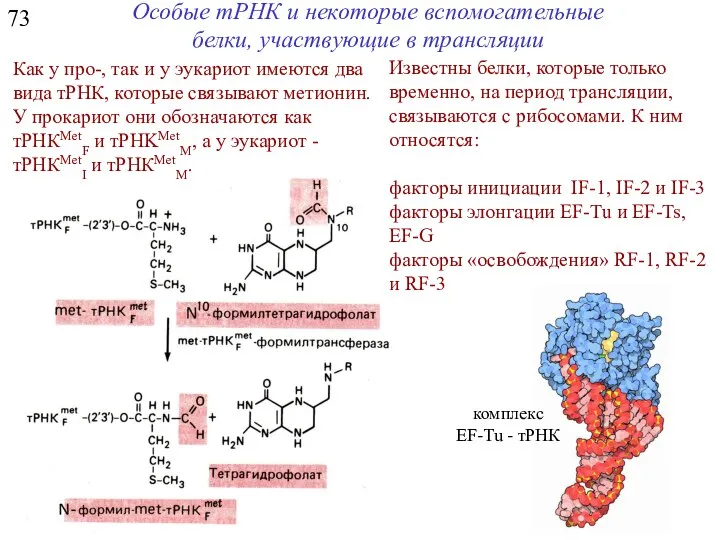
- 5. 73 Особые тРНК и некоторые вспомогательные белки, участвующие в трансляции Как у про-, так и у
- 6. 74 ТРАНСЛЯЦИЯ мРНК У ПРОКАРИОТ Полипептидные цепи синтезируются однонаправленно: с N-конца к C-концу. При этом карбоксильная
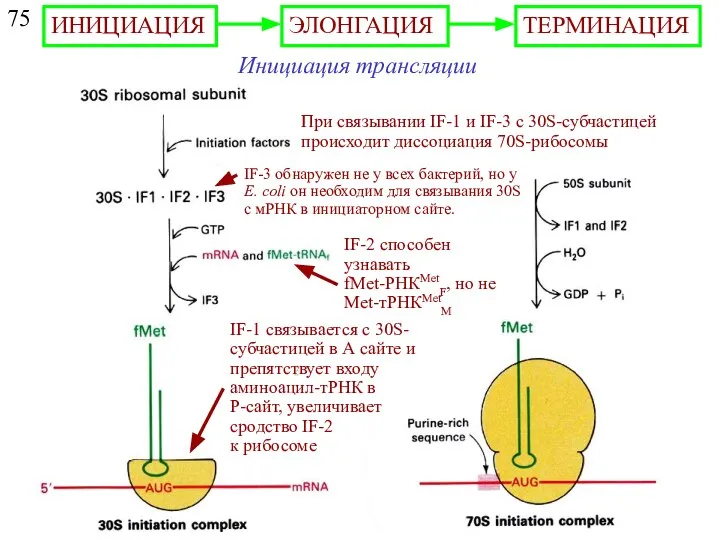
- 7. 75 Инициация трансляции IF-2 способен узнавать fMet-РНКMetF, но не Met-тРНКMetM При связывании IF-1 и IF-3 с
- 8. 76 Инициация трансляции Богатая пуринами последовательности из пяти-восьми нуклеотидов, последовательность Шайна-Дальгарно или SD-последовательность
- 9. 77 Элонгация полипептидной цепи На рибосоме три сайта связывания тРНК (Aminoacyl) (Peptidyl) (Exit)
- 10. 78 Элонгация полипептидной цепи Связывание аминоацил-тРНК в А-сайте и освобождение тРНК из Е-сайта
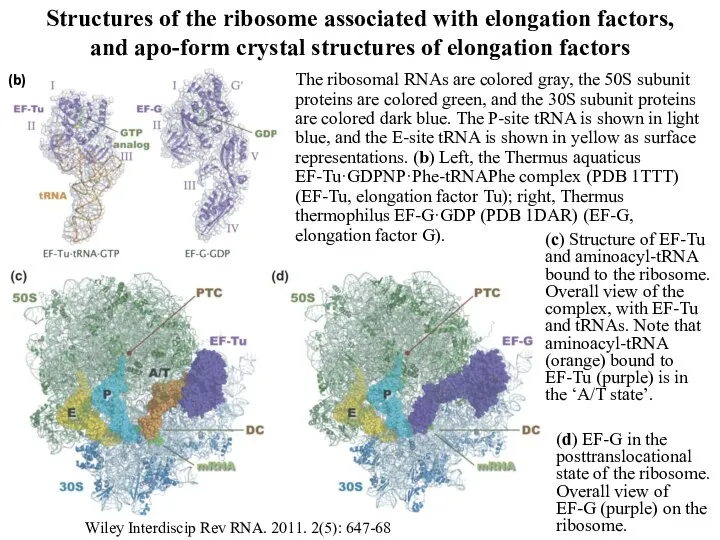
- 11. Structures of the ribosome associated with elongation factors, and apo-form crystal structures of elongation factors The
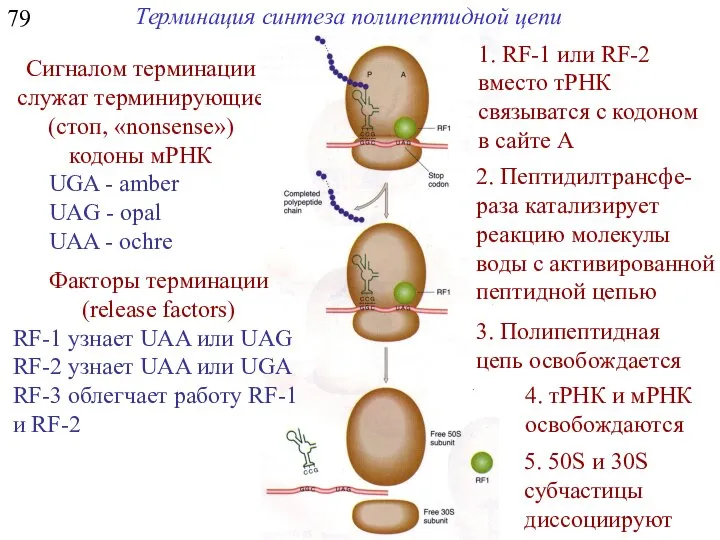
- 12. 79 Терминация синтеза полипептидной цепи Сигналом терминации служат терминирующие (стоп, «nonsense») кодоны мРНК UGA - amber
- 13. Essential steps in translation. The 30S (blue) and 50S (green) subunits harboring the decoding center (DC)
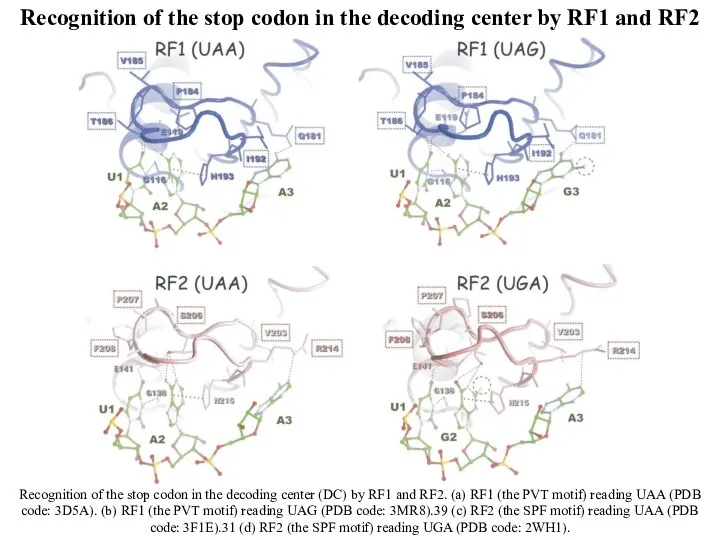
- 14. Recognition of the stop codon in the decoding center (DC) by RF1 and RF2. (a) RF1
- 15. 80 ТРАНСЛЯЦИЯ После трансляции кодирующей последовательности рибосомы начинают новый раунд. Диссоциация субчастиц контролируется фактором инициации IF-3
- 16. 81 ОБЩИЕ ОСОБЕННОСТИ ПРОЦЕССА ТРАНСЛЯЦИИ Одновременная трансляция молекулы мРНК более чем одной рибосомой Образование полирибосомы. После
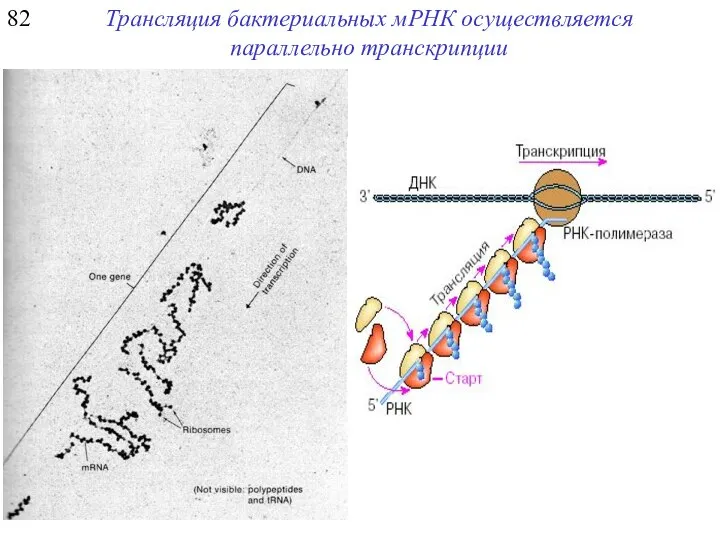
- 17. 82 Трансляция бактериальных мРНК осуществляется параллельно транскрипции
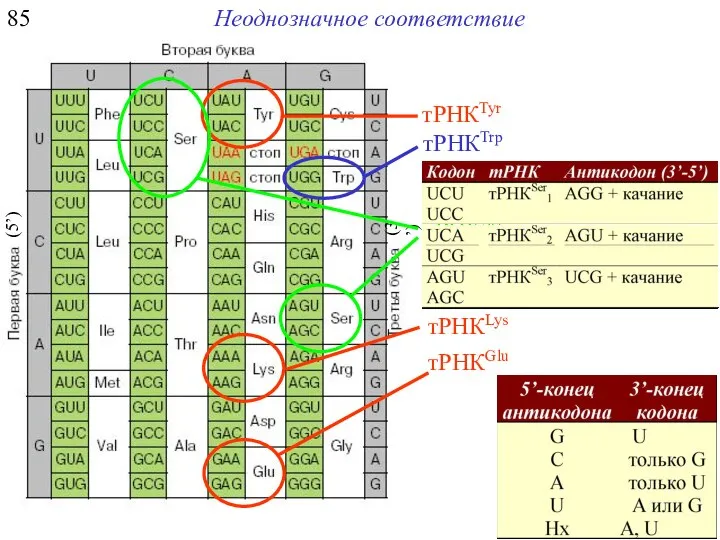
- 18. 83 Взаимодействие кодона и антикодона. Неоднозначное соответствие тРНКTyr тРНКGlu тРНКLys тРНКTrp тРНКSer1 тРНКSer2 тРНКSer3
- 19. 84 Неоднозначное соответствие 1. Некоторые аминокислоты могут доставляться к рибосоме несколькими типами тРНК, имеющими разные антикодоны.
- 20. 85 Неоднозначное соответствие тРНКTyr тРНКGlu тРНКLys тРНКTrp тРНКSer1 тРНКSer2 тРНКSer3
- 21. 86 Мутации в кодонах и антикодонах Трансляционная супрессия терминирующих кодонов Трансляционная супрессия миссенс-мутаций AUC CUA ААА
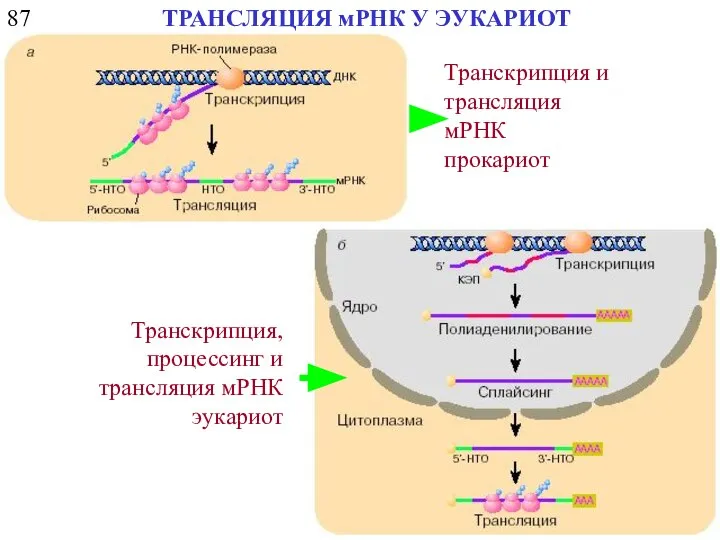
- 22. 87 ТРАНСЛЯЦИЯ мРНК У ЭУКАРИОТ Транскрипция и трансляция мРНК прокариот Транскрипция, процессинг и трансляция мРНК эукариот
- 23. 136 Структура эукариотической мРНК 7-метилгуаниловая кислота (кэп) Первичный транскрипт Последовательность Козак
- 24. Каноническая инициация трансляции у эукариот Схема предполагаемого расположения факторов инициации на 40S рибосомной субчастице. Вид со
- 26. Скачать презентацию















 Водорастворимые витамины
Водорастворимые витамины Репрезентативность геоботанических описаний лесных сообществ природного парка Кумысная поляна
Репрезентативность геоботанических описаний лесных сообществ природного парка Кумысная поляна Кошки и собаки
Кошки и собаки Аттестационнная работа. Развитие творческого потенциала учащихся через организацию исследовательской деятельности
Аттестационнная работа. Развитие творческого потенциала учащихся через организацию исследовательской деятельности Строение цветков однодольных растений
Строение цветков однодольных растений Коротко об акуле
Коротко об акуле Что такое кровь
Что такое кровь Пресмыкающиеся, особенности строения, многообразие
Пресмыкающиеся, особенности строения, многообразие Различия и сходства растительной и животной клетки
Различия и сходства растительной и животной клетки Ссавці. Парнокопитні. Жирафові. Жирафа степова
Ссавці. Парнокопитні. Жирафові. Жирафа степова  Электрон - транспортная цепь митохондрий
Электрон - транспортная цепь митохондрий Учения о клетке. (Лекция 14)
Учения о клетке. (Лекция 14) Основы культуры здоровья. Биологический возраст человека
Основы культуры здоровья. Биологический возраст человека Морфофункциональная характеристика скелета и аппарата движения верхних конечностей
Морфофункциональная характеристика скелета и аппарата движения верхних конечностей Биосинтез белков. Фактор терминации трансляции тРНК. (Лекция 14)
Биосинтез белков. Фактор терминации трансляции тРНК. (Лекция 14) Презентация Врожденные и приобретенные программы поведения
Презентация Врожденные и приобретенные программы поведения Настоящие папоротники
Настоящие папоротники Человек
Человек Віруси, пріони
Віруси, пріони Орсодакниды
Орсодакниды Вегетативная нервная система
Вегетативная нервная система Роль тепловых явлений в жизни живых организмов
Роль тепловых явлений в жизни живых организмов Интерактивное пособие по теме «Кровь и кровообращение» Часть 1 «Внутренняя среда организма» (для подготовки к ЕГЭ,ГИА и 8 класс
Интерактивное пособие по теме «Кровь и кровообращение» Часть 1 «Внутренняя среда организма» (для подготовки к ЕГЭ,ГИА и 8 класс Среды жизни планета Земля
Среды жизни планета Земля Многообразие водорослей
Многообразие водорослей  Позитивные взаимоотношения – симбиоз
Позитивные взаимоотношения – симбиоз ОСНОВЫ БИОТЕХНОЛОГИИ КЛЕТОЧНАЯ ИНЖЕНЕРИЯ. РАСТЕНИЯ. Лекция 8
ОСНОВЫ БИОТЕХНОЛОГИИ КЛЕТОЧНАЯ ИНЖЕНЕРИЯ. РАСТЕНИЯ. Лекция 8  Агрономия. Классификация севооборотов
Агрономия. Классификация севооборотов