Содержание
- 2. PCR “polymerase chain reaction” Descrizione della tecnica, metodo, componenti, variabili, strumenti = termociclatori Tecnica: amplificazione esponenziale
- 3. Come si fa la PCR, in cosa consiste amplificazione tramite sintesi de-novo di nuovi filamenti di
- 4. 1 denaturazione del DNA a 94°C 2 appaiamento (“annealing”) dei primers al ssDNA templato, temperatura misurata
- 5. applicazione del sistema naturale la sintesi del DNA è semiconservativa (duplicazione) le DNA polimerasi e anche
- 6. l’invenzione geniale dalla amplificazione lineare a quella esponenziale: la sintesi in vitro di DNA già era
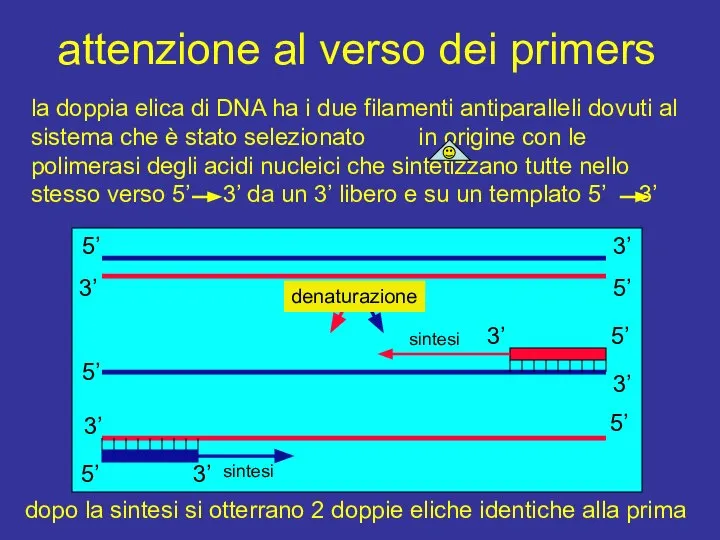
- 7. attenzione al verso dei primers la doppia elica di DNA ha i due filamenti antiparalleli dovuti
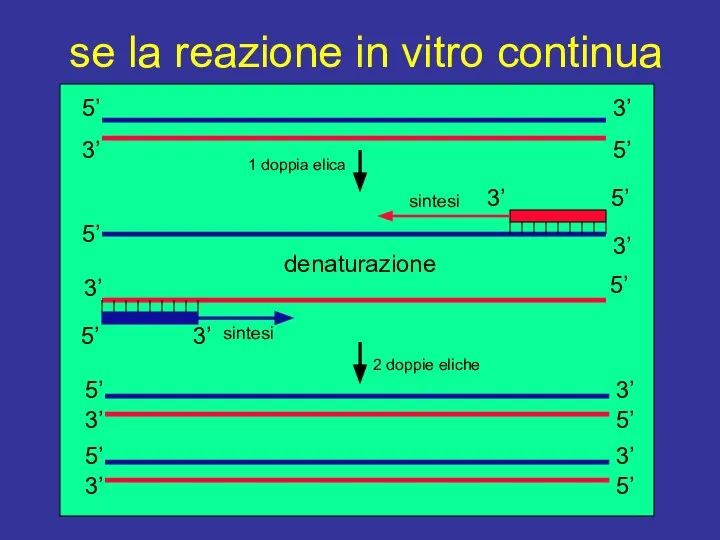
- 8. se la reazione in vitro continua 1 doppia elica 2 doppie eliche
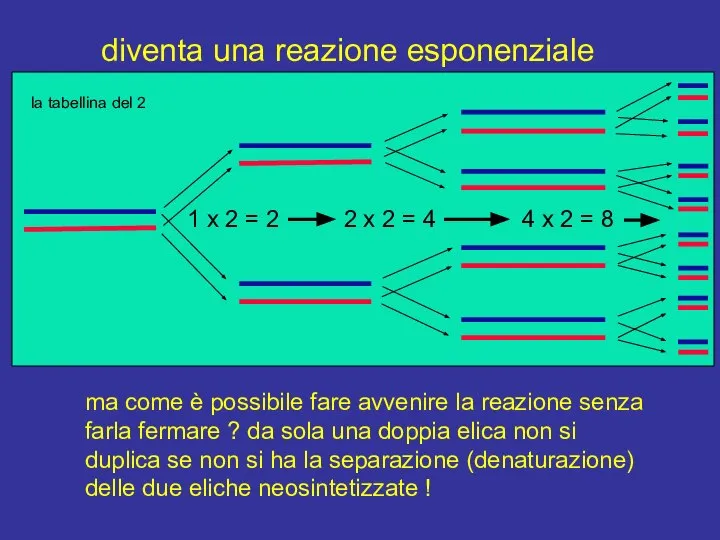
- 9. diventa una reazione esponenziale ma come è possibile fare avvenire la reazione senza farla fermare ?
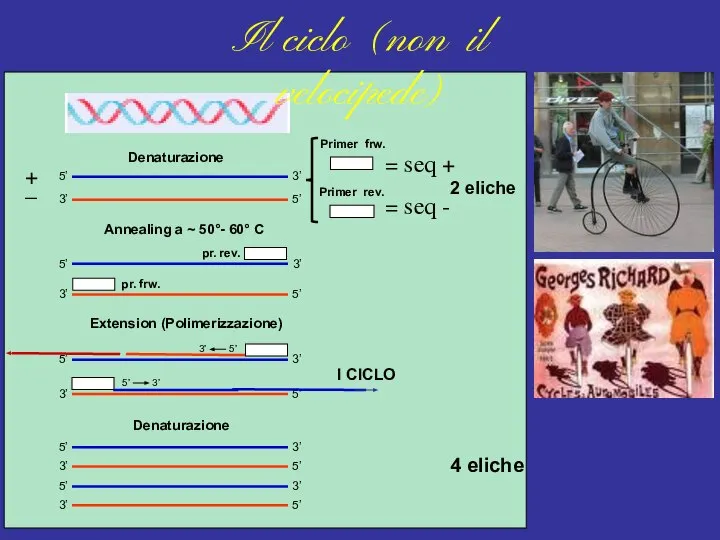
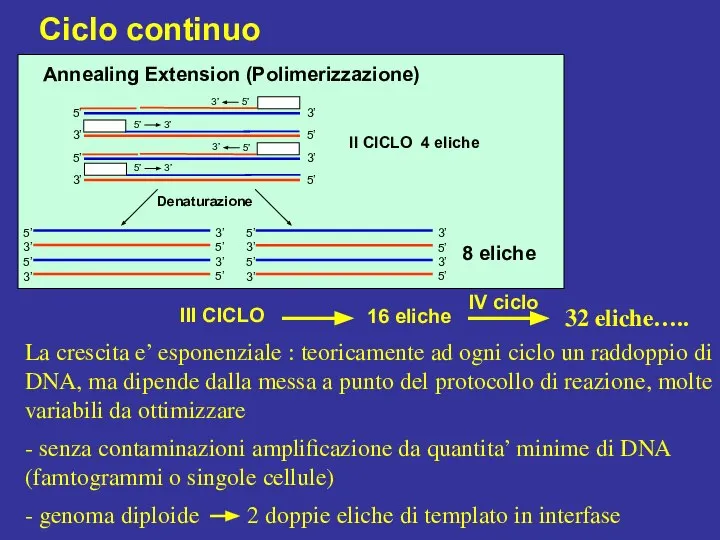
- 10. 5’ 5’ 5’ 3’ 3’ 3’ 3’ 3’ 3’ 5’ 5’ 5’ Denaturazione Extension (Polimerizzazione) Annealing
- 11. Annealing Extension (Polimerizzazione) La crescita e’ esponenziale : teoricamente ad ogni ciclo un raddoppio di DNA,
- 12. vanno determinate per i tempi, temperature e quantita’(conc.) ogni ciclo = 3 passaggi (fasi) I passaggio
- 13. l’enzima sarà una Taq polimerasi (ne esistono diverse ottenute da mutanti che aumentano la specificità, efficienza
- 14. Volume di reazione: da 10 a 50 μl (max 100 μl) per una preparativa. Il DNA
- 15. la PCR si utilizza per amplificare i frammenti random da sequenziare a cui sono stati attaccati
- 16. applicazioni della PCR RT-PCR, nested PCR PCR inversa RACE 3’ e RACE 5’ variazioni sul tema
- 17. Ricerca di un vettore Per inserzione random nel genoma Per inserzione sito specifica nel genoma Cosa
- 18. Cambia la regione limitrofa Gene targeting deve avere regioni limitrofe note a) Random recombination ha regioni
- 19. Altre possibilità di analisi tramite PCR perfezionamenti delle tecniche e degli enzimi - nuove macchine con
- 20. Altre possibili applicazioni della PCR - abbiamo visto RT-PCR tramite reverse transcriptase da mRNA RT-PCR è
- 21. applicazione RT-PCR nuovo esercizio: se devo retrotrascrivere un mRNA per ottenere un un cDNA devo fare
- 22. La reverse trascrittasi RT L’uso della reverse trascrittasi risale a quando furono scoperti i meccanismi molecolari
- 23. RT-PCR: cosa si analizza = analisi della trascrizione di un gene o isolamento di un cDNA
- 24. stratagemmi della RT-PCR Accorgimento: quando si estrae l’RNA si deve evitare il DNA e si puo’
- 25. la retrotrascrizione Per RT si intende reverse transcriptase su templato di RNA Per avere un cDNA
- 26. vantaggi della RT-PCR Analisi della trascrizione tramite PCR Analisi della trascrizione e non determinazione del PM
- 27. come si fa una RT-PCR Si deve ottenere il retrotrascritto cioè il cDNA ( DNA complementare
- 28. RT-PCR dal II filamento in poi Accorgimenti e controlli della RT-PCR Prima di retrotrascrivere il cDNA
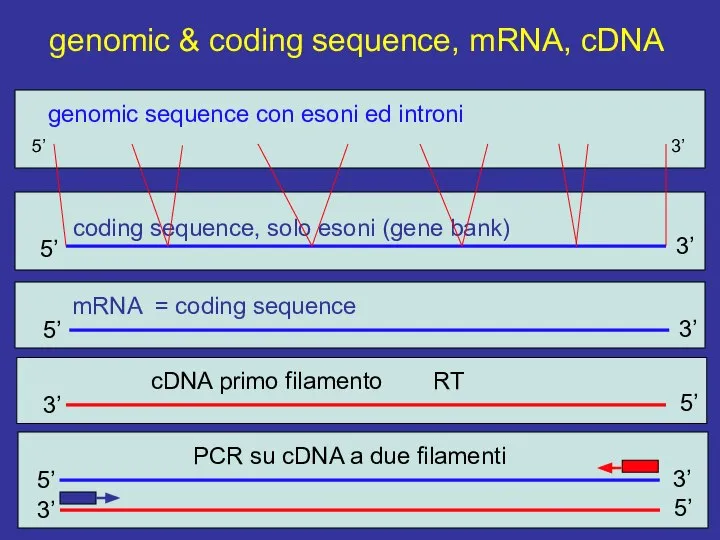
- 29. genomic & coding sequence, mRNA, cDNA 5’ 3’ 5’ 3’ PCR su cDNA a due filamenti
- 30. Correzione parametri di una PCR La PCR deve dare dei prodotti che corrispondono agli attesi Quando
- 31. sequenza di un cDNA dalla banca dati per aumentare specificità? facciamo una doppia PCR : la
- 32. la sequenza 5’- 3’ di un cDNA GGATCCCTGT TCCTGATCAC TGATCTCTGG TTCTTTTATT ATGCATATTC 50 ATTTTGAAAT CTGATTCCTT TTCTGAGCAT
- 33. una nested PCR 1 gatcacaggt ctatcaccct attaaccact cacgggagct ctccatgcat ttggtatttt 61 cgtctggggg gtgtgcacgc gatagcattg cgagacgctg gagccggagc
- 34. i controlli essenziali Controlli, negativi, positivi, (i controlli ci fanno capire se l’esperimento è venuto bene)
- 35. procedure la Rev Transcript virale a 37°C, mutanti max 65°C oligo dT, random priming con esanucleotidi,
- 36. precauzioni estrarre RNA eliminando DNA genomico che falsifica il risultato cosa si vuole vedere con RT-PCR:
- 37. può essere quantitativa? la RT-PCR può essere quantitativa l’amplificazione è proporzionale alla quantità di templato l’amplificazione
- 38. perchè quantitativa ? l’amplificazione è proporzionale al templato iniziale, perchè si conserva la proporzionalità anche dopo
- 39. la rivelazione su gel dopo elettroforesi su gel di agarosio si rivela il DNA amplificato come
- 40. controllo di RT-PCR altro controllo negativo : assenza di amplificazione sui campioni di RNA non retrotrascritti
- 41. Controlli della PCR Controlli di estrazione: quali? ripetibilità della amplificazione ripetibilità su campioni indipendenti univocità di
- 42. R.A.C.E. Con la RT-PCR si amplifica solo un frammento del cDNA Se si vuole identificare l’intero
- 43. (Rapid Amplification of cDNA Ends) La RACE è una tecnica per l’amplificazione di sequenze nucleotidiche, usando
- 44. Differenze nella ricerca di 5’ o 3’ ignoti mRNA AAAAA 5’ 3’ poly TTTTT 5’ 3’
- 45. Cerchiamo il 3’ sconosciuto In questo caso si usa un oligo dT per sintetizzare il cDNA
- 46. Dalle banche EST (expressed sequence tags); Da studi di funzione (per esempio EXON e PROMOTER TRAPPING);
- 47. mRNA poly A 5’ noto 3’ ignoto sintesi del I filamento di cDNA con RT con
- 48. Scelta dei primers per la RACE 3’ Il primo primer obbligato è quello per la RT
- 49. RACE 3’ TTT…..TTT 5’ 5’ mRNA poly(A) tail 1 - Annealing tra la coda di polyA
- 50. Nested PCR o PCR interna cDNA 5’ 3’ TTTTT regione nota regione ignota I primer specifico
- 51. un trucco che inganna la polimerasi 3’ 5’ 5’ 3’ primer la polimerase estende da un
- 52. 2 - Degradazione del templato di RNA 3 - Amplificazione per PCR usando un primer specifico
- 53. GSP 2 TTT…..TTT 5’ 3’ AUAP UAP 3’ 5’ La SPECIFICITA’ può essere garantita da un
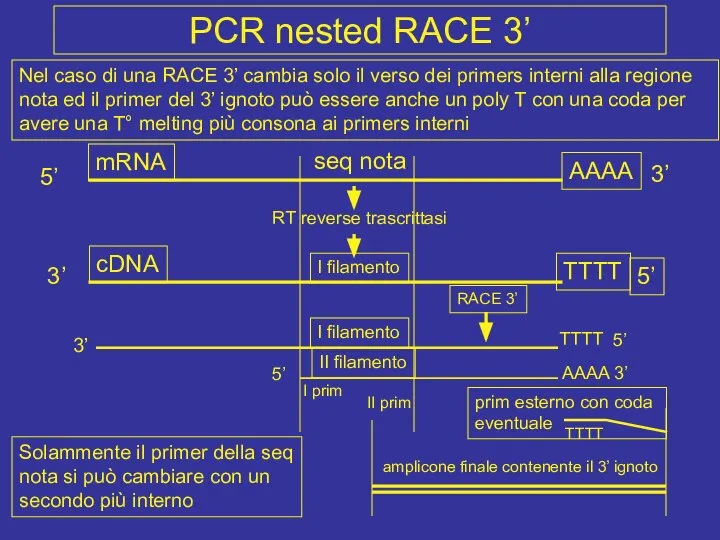
- 54. PCR nested RACE 3’ Nel caso di una RACE 3’ cambia solo il verso dei primers
- 55. RACE 5’ Cerchiamo il 5’ ignoto Dobbiamo comunque ottenere il cDNA ed utiliziamo gli stessi metodi
- 56. 5’ mRNA poly(A) tail 1 - Annealing tra una regione interna dell’mRNA e un primer gene
- 57. 2 - Retrotrascrizione e tailing all’estremità 3’ del cDNA 5’ AAA….AAA n 5’ 3’ Degradazione mRNA
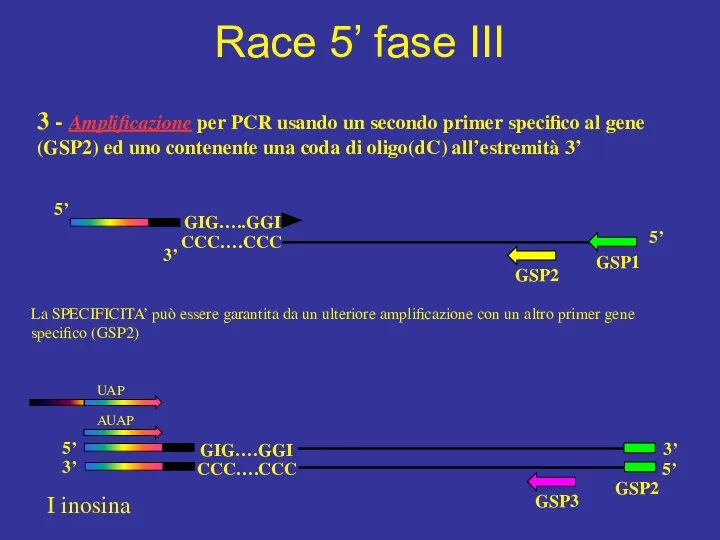
- 58. Race 5’ fase III I inosina
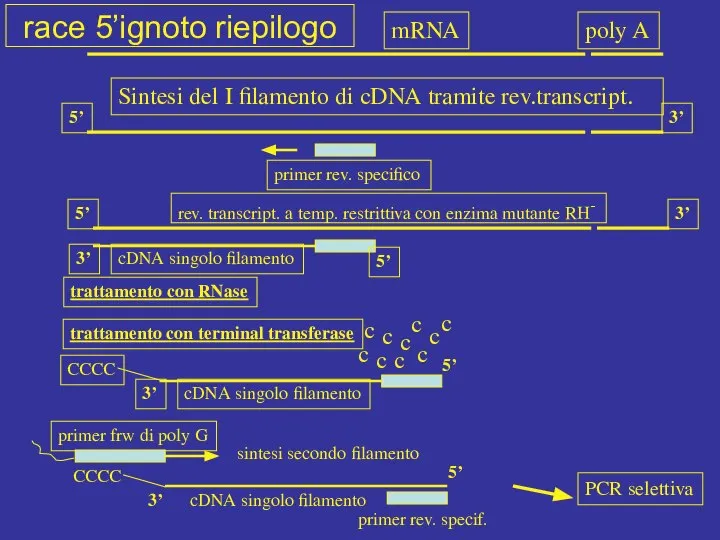
- 59. mRNA poly A Sintesi del I filamento di cDNA tramite rev.transcript. primer rev. specifico 5’ 3’
- 60. dopo la sintesi del I filam. di cDNA e la terminal transferase, PCR selettiva con II
- 62. Скачать презентацию


















































 Деление клетки. Митоз. Подготовила учитель химии и биологии Родионова Н.Г. МОБУ «Лобасковская ООШ»
Деление клетки. Митоз. Подготовила учитель химии и биологии Родионова Н.Г. МОБУ «Лобасковская ООШ»  Коммуникация у кошек
Коммуникация у кошек В мире насекомых
В мире насекомых ИММУНИТЕТ от лат. immunitas — освобождение, избавление
ИММУНИТЕТ от лат. immunitas — освобождение, избавление  Одноклеточные ядерные организмы
Одноклеточные ядерные организмы Плесневые грибы и дрожжи
Плесневые грибы и дрожжи Презентация на тему "Рыбы" - скачать бесплатно презентации по Биологии
Презентация на тему "Рыбы" - скачать бесплатно презентации по Биологии Фолдинг белка. Физикохимические свойства белков
Фолдинг белка. Физикохимические свойства белков Деление клетки
Деление клетки Физиология цнс. Промежуточный мозг. Базальные ганглии. Кора больших полушарий головного мозга
Физиология цнс. Промежуточный мозг. Базальные ганглии. Кора больших полушарий головного мозга Презентация на тему "Цветение и опыление растений" - презентации по Биологии
Презентация на тему "Цветение и опыление растений" - презентации по Биологии ДНК в генной инженерии
ДНК в генной инженерии Биотехнологии в пищевой промышленности
Биотехнологии в пищевой промышленности Класс Амфибии (Земноводные)
Класс Амфибии (Земноводные) Роль К.Ф. Фукса в истории города (Комбинированная экскурсия для гостей Казани по местам, связанным с К.Ф.Фуксом на 3 часа) Подгот
Роль К.Ф. Фукса в истории города (Комбинированная экскурсия для гостей Казани по местам, связанным с К.Ф.Фуксом на 3 часа) Подгот Кровообрщение
Кровообрщение Птицы. 7 класс
Птицы. 7 класс Регуляция и патология липидного обмена
Регуляция и патология липидного обмена Многообразие и значение грибов
Многообразие и значение грибов Тема урока: Эволюция кровеносной системы.
Тема урока: Эволюция кровеносной системы. Тип Плоские черви. Вы уже изучили И много нового для себя открыли Симметрия их тела, Скажите без промедления, имеет: Двусторо
Тип Плоские черви. Вы уже изучили И много нового для себя открыли Симметрия их тела, Скажите без промедления, имеет: Двусторо Урок по предмету Байкаловедение. Тема: «Нерпа». 6кл. МОУ СОШ №11 г. Иркутск. Учитель высшей квалификационно
Урок по предмету Байкаловедение. Тема: «Нерпа». 6кл. МОУ СОШ №11 г. Иркутск. Учитель высшей квалификационно Общая характеристика грибов
Общая характеристика грибов Жизненный цикл клетки
Жизненный цикл клетки Презентация на тему "Использование информационно-коммуникационных технологий на уроках биологии" - презентации по Биологии
Презентация на тему "Использование информационно-коммуникационных технологий на уроках биологии" - презентации по Биологии Тип Членистоногие
Тип Членистоногие Популяция. Генетическая единица вида
Популяция. Генетическая единица вида Семейство Астровые или Сложноцветые (Asteraceae или Compositae)
Семейство Астровые или Сложноцветые (Asteraceae или Compositae)