Содержание
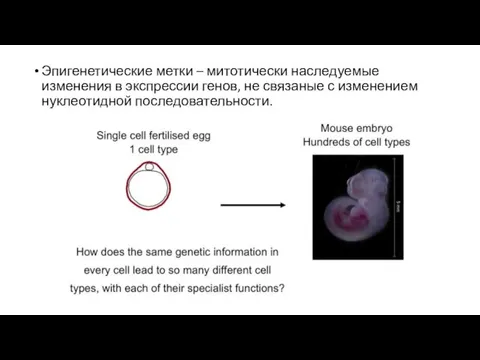
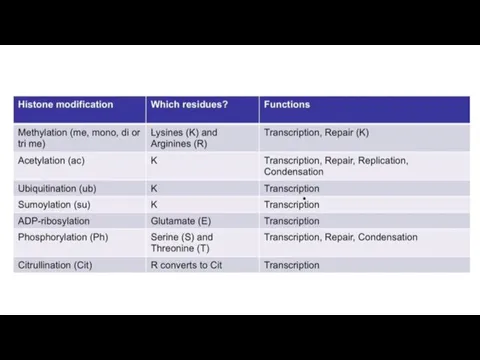
- 2. Эпигенетические метки – митотически наследуемые изменения в экспрессии генов, не связаные с изменением нуклеотидной последовательности.
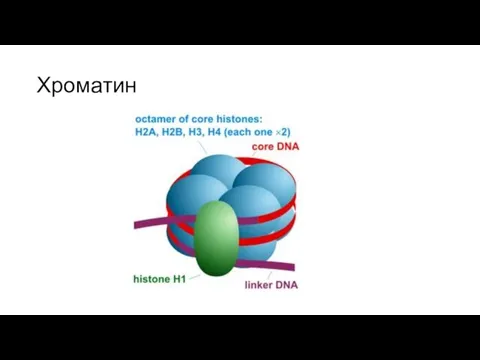
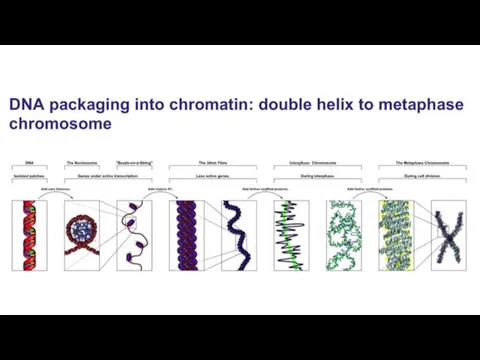
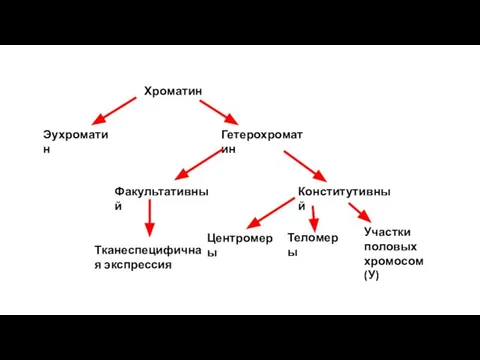
- 3. Хроматин
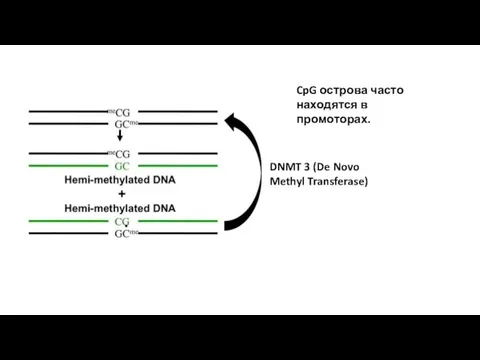
- 8. CpG острова часто находятся в промоторах.
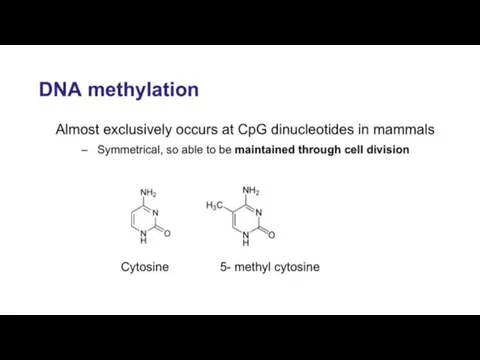
- 12. Метилирование СрG мутагенно В геноме CG пар меньше, чем можно было ожидать вероятностно, т.к. metC переходит
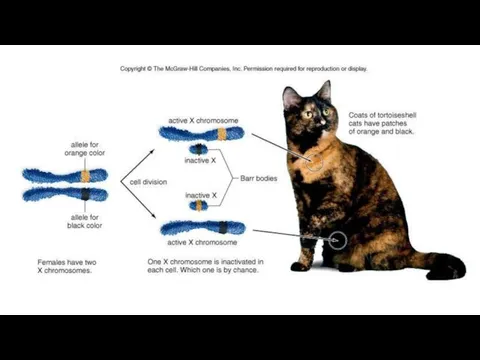
- 15. Тельце Барра
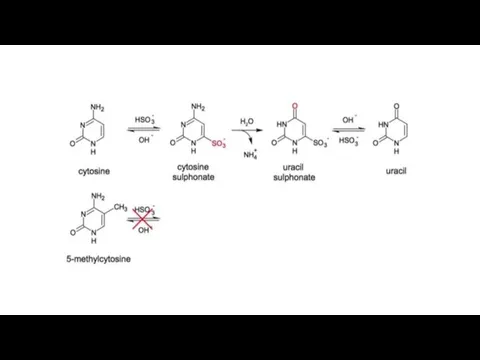
- 16. Бисульфитное секвенирование – определение метилированных CpG
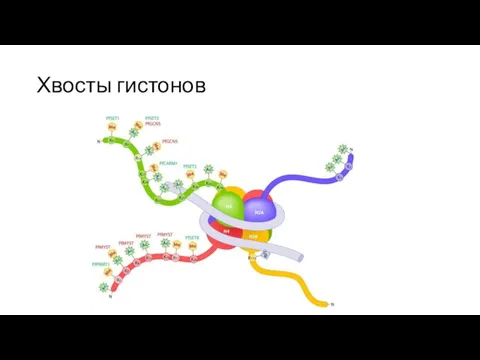
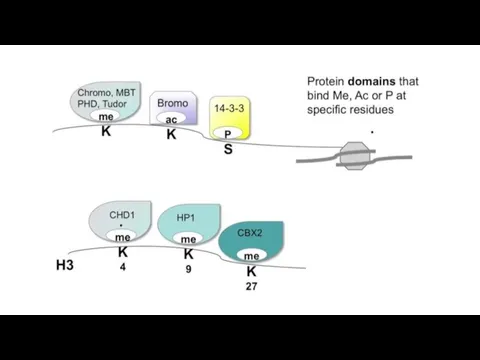
- 18. Хвосты гистонов
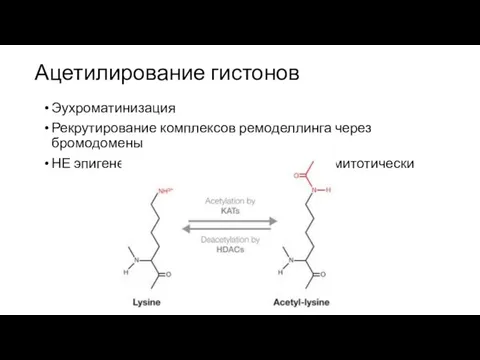
- 20. Ацетилирование гистонов Эухроматинизация Рекрутирование комплексов ремоделлинга через бромодомены НЕ эпигенетическая метка – не передается митотически
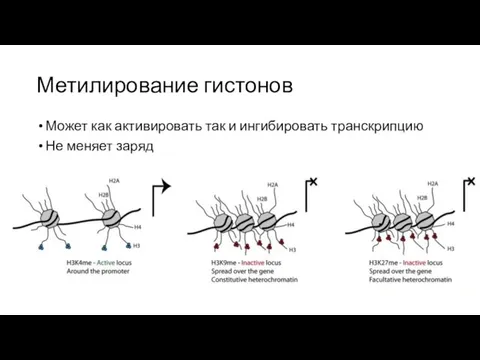
- 21. Метилирование гистонов Может как активировать так и ингибировать транскрипцию Не меняет заряд
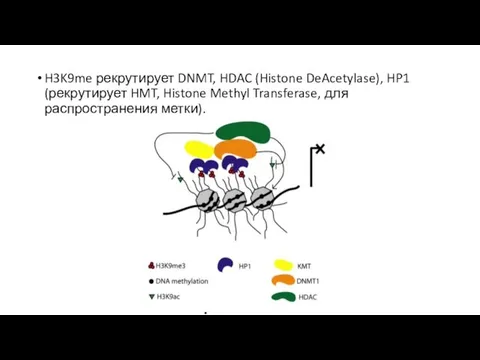
- 23. H3K9me рекрутирует DNMT, HDAC (Histone DeAcetylase), HP1 (рекрутирует HMT, Histone Methyl Transferase, для распространения метки).
- 24. Комплексы ремоделлинга – АТФ-зависимое передвижение нуклеосом
- 25. Варианты гистонов Гистоны Н2А, Н3 и Н1 бывают в виде нескольких вариантов. CENP-A (centromere specific histone)
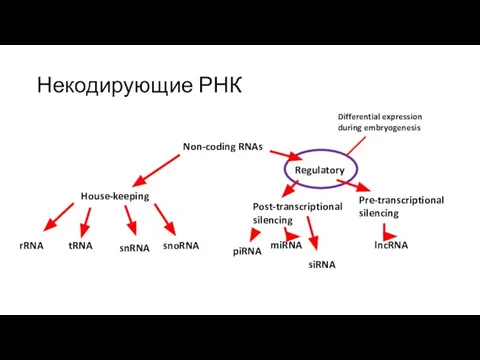
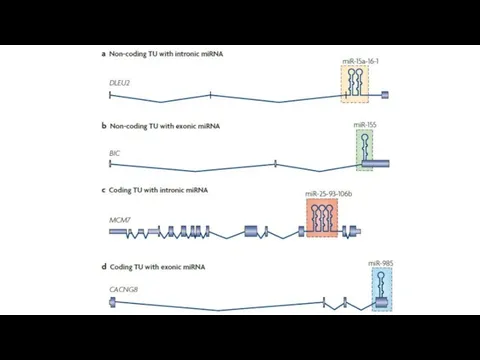
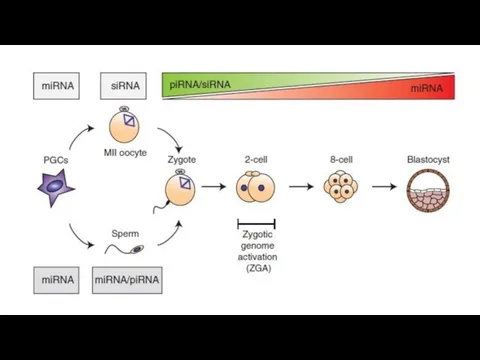
- 26. Некодирующие РНК
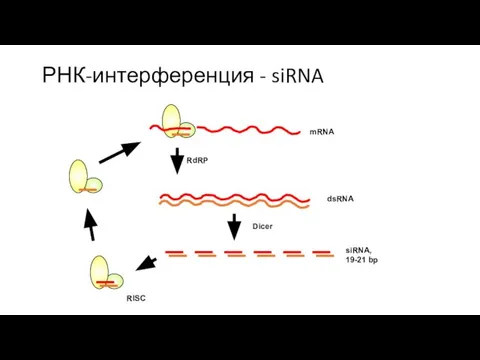
- 27. РНК-интерференция - siRNA
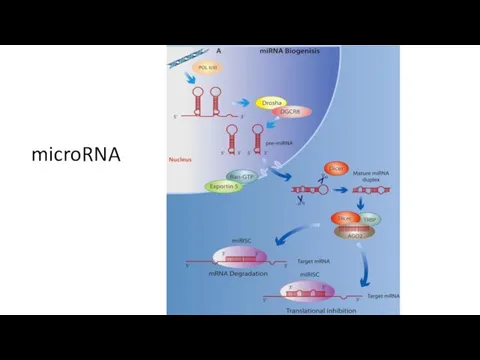
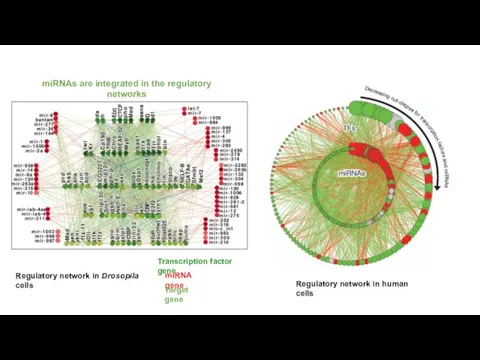
- 28. microRNA
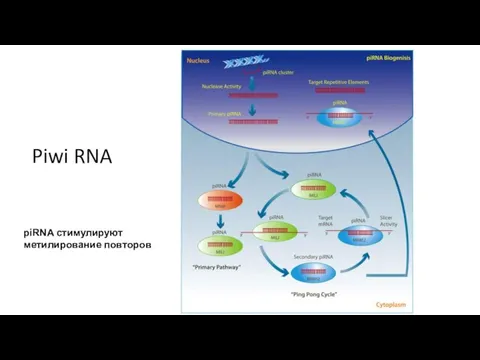
- 30. Piwi RNA piRNA стимулируют метилирование повторов
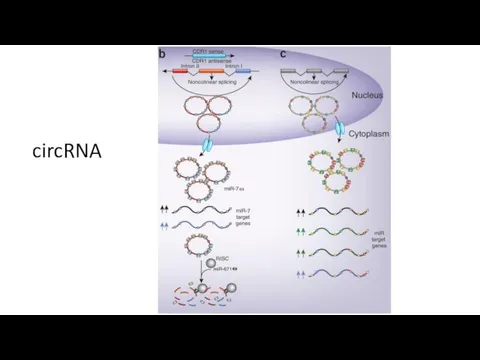
- 33. circRNA
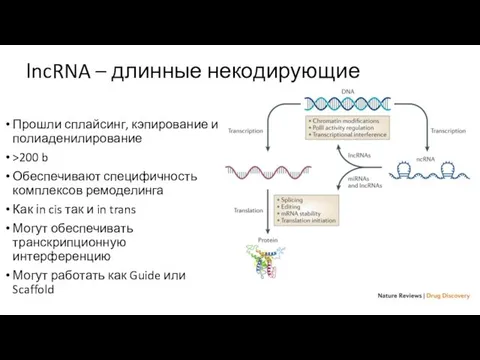
- 35. lncRNA – длинные некодирующие Прошли сплайсинг, кэпирование и полиаденилирование >200 b Обеспечивают специфичность комплексов ремоделинга Как
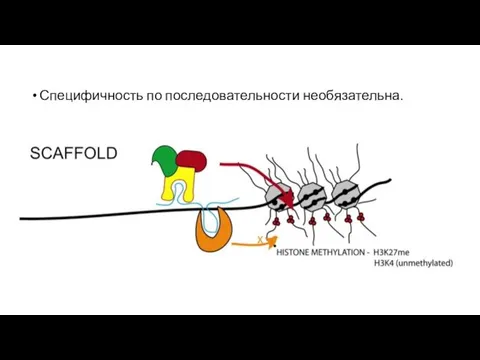
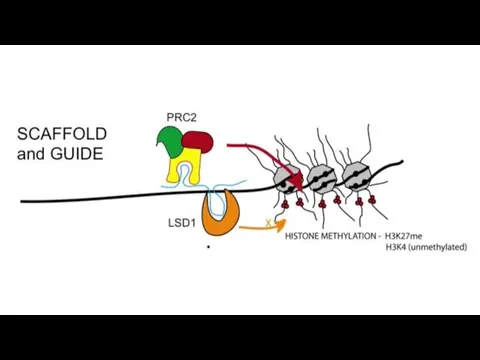
- 37. Специфичность по последовательности необязательна.
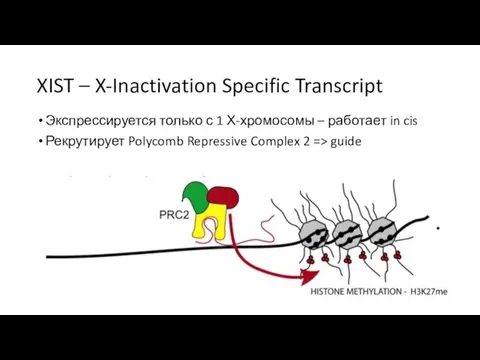
- 38. XIST – X-Inactivation Specific Transcript Экспрессируется только с 1 Х-хромосомы – работает in cis Рекрутирует Polycomb
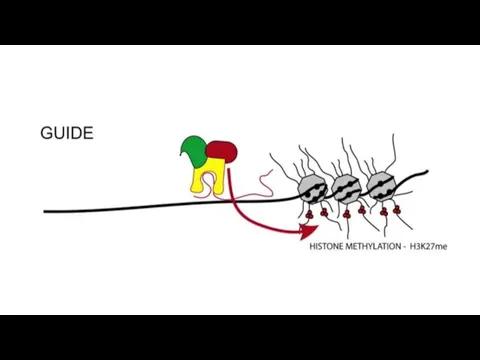
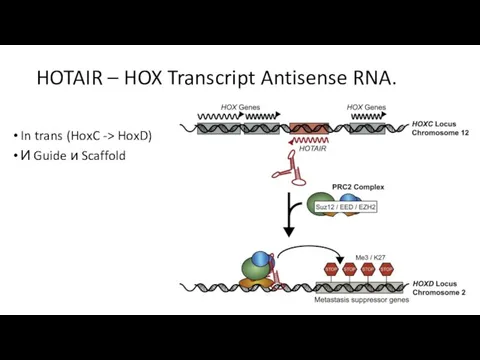
- 39. HOTAIR – HOX Transcript Antisense RNA. In trans (HoxC -> HoxD) И Guide и Scaffold
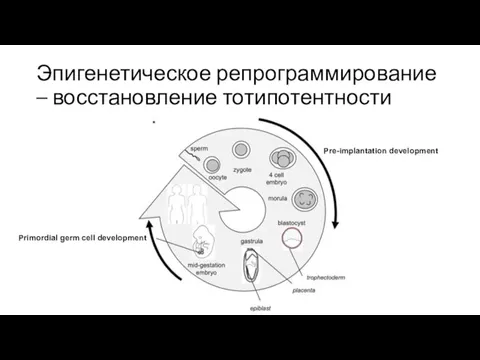
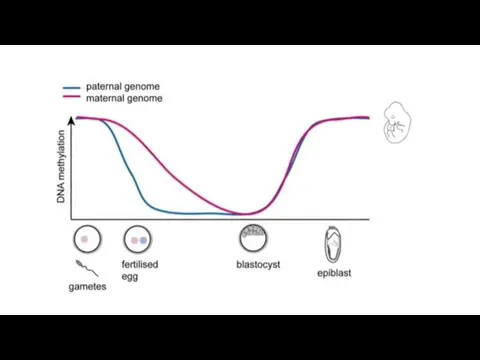
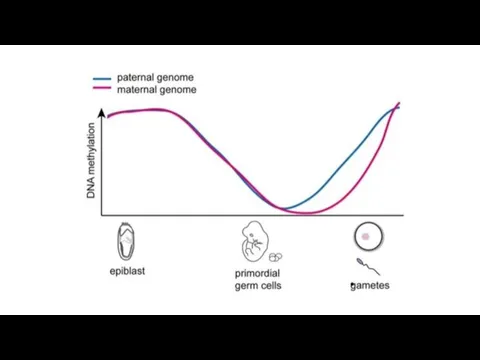
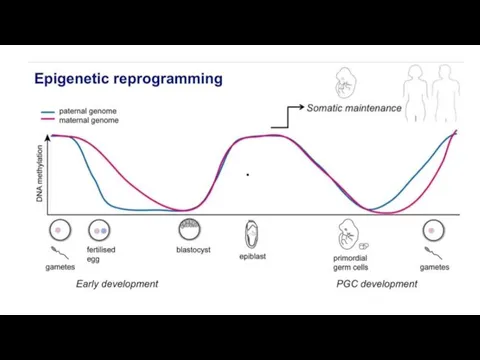
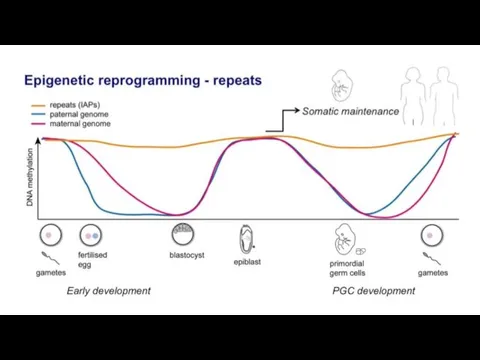
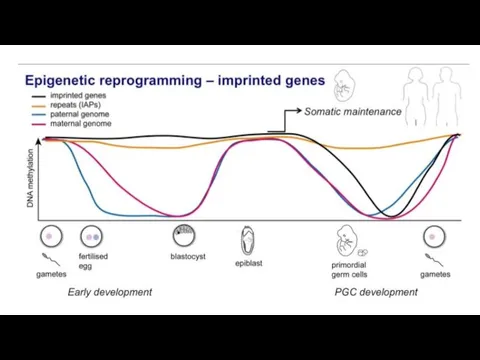
- 41. Эпигенетическое репрограммирование – восстановление тотипотентности
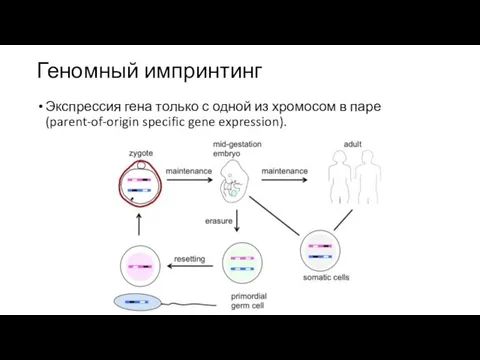
- 46. Геномный импринтинг Экспрессия гена только с одной из хромосом в паре (parent-of-origin specific gene expression).
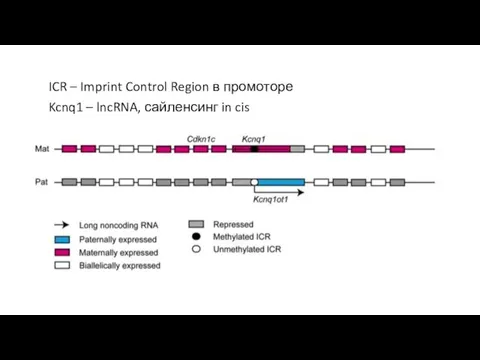
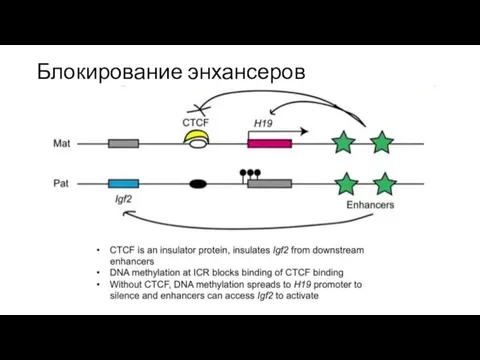
- 48. ICR – Imprint Control Region в промоторе Kcnq1 – lncRNA, сайленсинг in cis
- 49. Блокирование энхансеров
- 50. Эволюция импринтинга Разделение на социальные группы – родство не 1, а 1/2
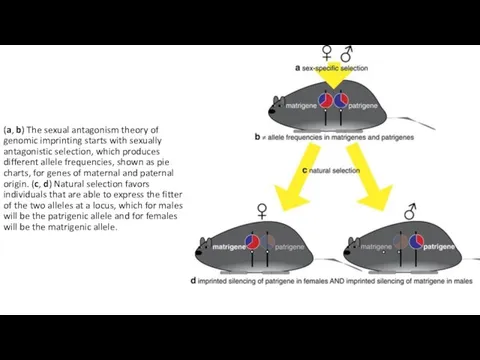
- 51. (a, b) The sexual antagonism theory of genomic imprinting starts with sexually antagonistic selection, which produces
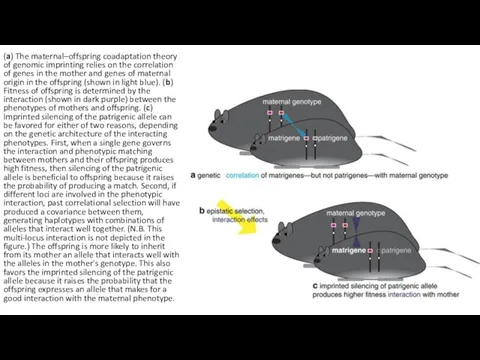
- 52. (a) The maternal–offspring coadaptation theory of genomic imprinting relies on the correlation of genes in the
- 54. Скачать презентацию












 Лекция 18. Фармакологическая регуляция гемостаза
Лекция 18. Фармакологическая регуляция гемостаза Работа врачей общей практики в г. Гродно
Работа врачей общей практики в г. Гродно Любовь или созависимость
Любовь или созависимость Общая и частная психопатология
Общая и частная психопатология Зарубежная психология периода открытого кризиса (10-е - середина 30-х годов 20 века)
Зарубежная психология периода открытого кризиса (10-е - середина 30-х годов 20 века)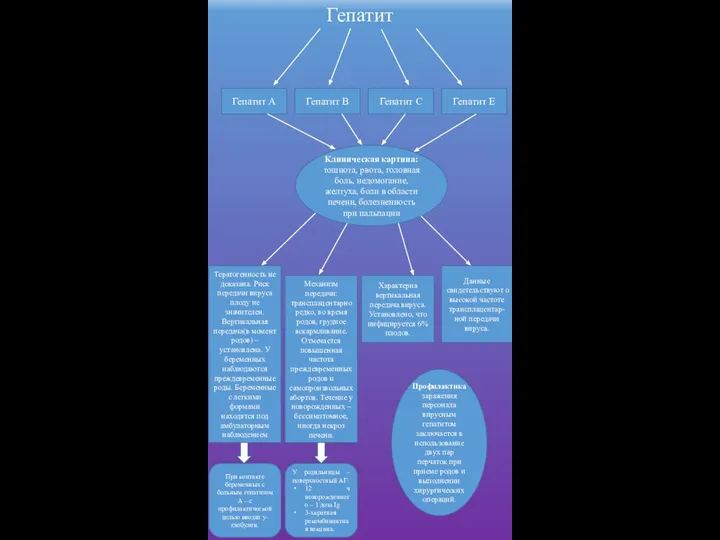 Гепатит
Гепатит Правильное питание
Правильное питание Психологическая классификация профессий (специальностей)
Психологическая классификация профессий (специальностей) Ведение пациентов с внебольничной пневмонией
Ведение пациентов с внебольничной пневмонией ЭТИОЛОГИЯ
ЭТИОЛОГИЯ Одонтогенные опухоли
Одонтогенные опухоли Причины конфликтов
Причины конфликтов презентация_авт
презентация_авт Расстройства воли и влечений, двигательные нарушения
Расстройства воли и влечений, двигательные нарушения Особенности Профессиональной гигиены у детей и подростков
Особенности Профессиональной гигиены у детей и подростков Лечебная физкультура при нарушениях осанки
Лечебная физкультура при нарушениях осанки Практики выявления и исцеления психосоматических заболеваний
Практики выявления и исцеления психосоматических заболеваний Диагностика заболеваний глотки и гортани. Аденоиды
Диагностика заболеваний глотки и гортани. Аденоиды Венерические болезни. Раздел 4. Гонорея у мужчин, женщин и детей
Венерические болезни. Раздел 4. Гонорея у мужчин, женщин и детей Гонококковая инфекция у женщин
Гонококковая инфекция у женщин Антигипертензивные лс. Мочегонные лс ( диуретики) . Лс повышающие артериальное давление (гипертензивные лс)
Антигипертензивные лс. Мочегонные лс ( диуретики) . Лс повышающие артериальное давление (гипертензивные лс) Влияние излучения, исходящего из сотового телефона на организм человека
Влияние излучения, исходящего из сотового телефона на организм человека Презентация по медицине Гнойные заболевания костей и суставов
Презентация по медицине Гнойные заболевания костей и суставов  Острое воспаление
Острое воспаление Искусственные органы
Искусственные органы Отчет о производственной практике в стоматологической поликлинике
Отчет о производственной практике в стоматологической поликлинике Инфекциялық иммунология негіздері. Иммунитет түрлері және формалары. Организмнің бейспецификалық қорғаныс
Инфекциялық иммунология негіздері. Иммунитет түрлері және формалары. Организмнің бейспецификалық қорғаныс 10 аргументов, чтобы прекратить потребление алкоголя
10 аргументов, чтобы прекратить потребление алкоголя