Содержание
- 2. СИНДРОМ - ? ВПС Особенности фенотипа Аномалии соединительной ткани Задержка развития Особенности характера и поведения Задержка
- 3. СИНДРОМ - ? Неонатальная гипотония Нормальный или ускоренный рост Тяжелая задержка развития Нормальная окружность головы Множественные
- 4. 1 970,0 млн USD 469,2 млн USD
- 5. 1 970,0 млн USD 469,2 млн USD dup 17p11.2 del 1p36 del 9q34.3 del 3q29 del
- 6. ХРОМОСОМНАЯ ПАТОЛОГИЯ ОПРЕДЕЛЕНИЕ Хромосомная патология – любое нарушение числа и/или структуры хромосом
- 7. ХРОМОСОМНАЯ ПАТОЛОГИЯ КЛИНИЧЕСКОЕ ЗНАЧЕНИЕ числовые и структурные аномалии хромосом (за исключением сбалансированных) патогенны характеризуются мультисистемным поражением
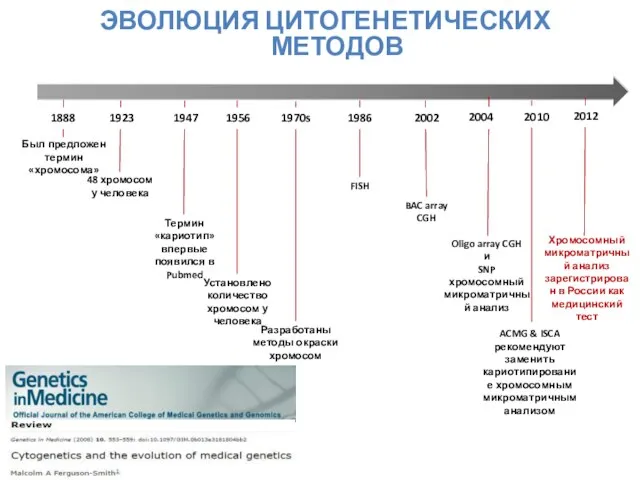
- 8. ЭВОЛЮЦИЯ ЦИТОГЕНЕТИЧЕСКИХ МЕТОДОВ Термин «кариотип» впервые появился в Pubmed Был предложен термин «хромосома» 48 хромосом у
- 9. НА ПУТИ К МОЛЕКУЛЯРНОМУ КАРИОТИПИРОВАНИЮ
- 10. НА ПУТИ К МОЛЕКУЛЯРНОМУ КАРИОТИПИРОВАНИЮ
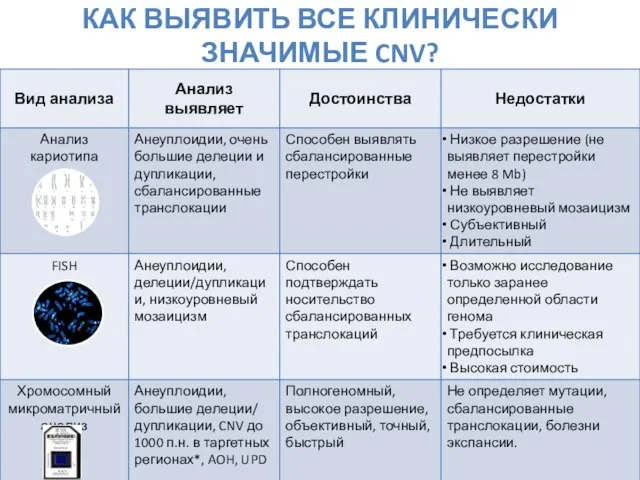
- 11. КАК ВЫЯВИТЬ ВСЕ КЛИНИЧЕСКИ ЗНАЧИМЫЕ CNV?
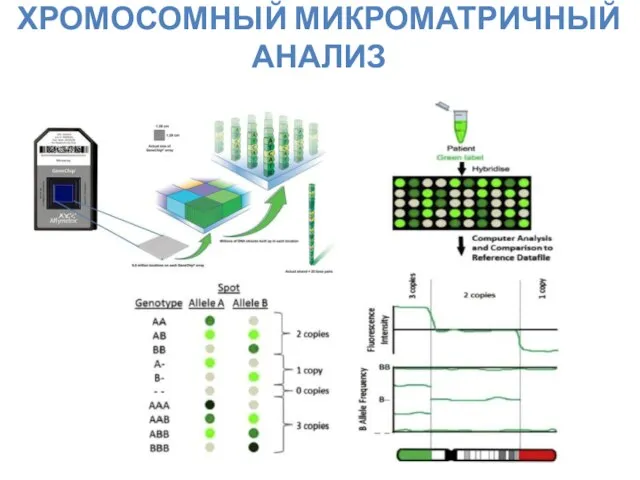
- 12. Хромосомный микроматричный анализ – молекулярно-цитогенетический метод анализа вариаций числа копий ДНК по сравнению с набором проб
- 13. ИСТОРИЯ Первый прототип микроматицы на микроскопном стекле 1993 Создание микроматрицы с общей последовательностью проб >1 млн.
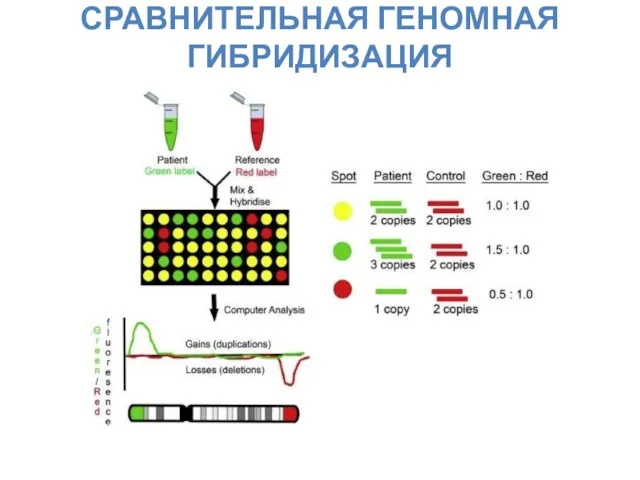
- 14. СРАВНИТЕЛЬНАЯ ГЕНОМНАЯ ГИБРИДИЗАЦИЯ
- 15. ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ
- 16. Normal 2N AA:(0.5+0.5)=+1 AB: 0.5-0.5 = 0 BB: 0-(0.5+0.5)=-1 Loss 1N A:0.5-0 =+0.5 B:0-0.5=-0.5 Mosaic loss
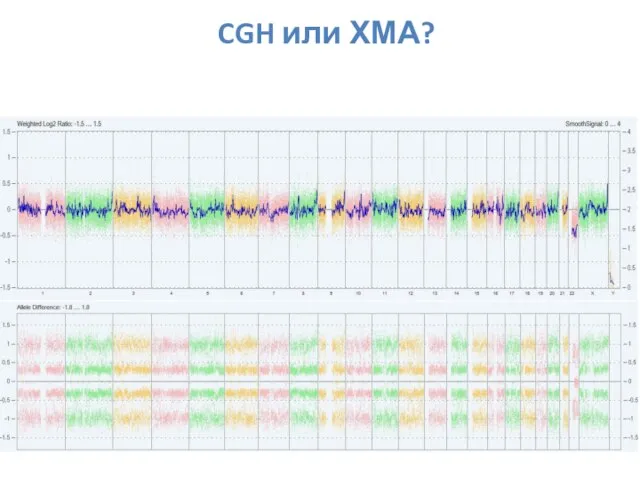
- 17. CGH или ХМА?
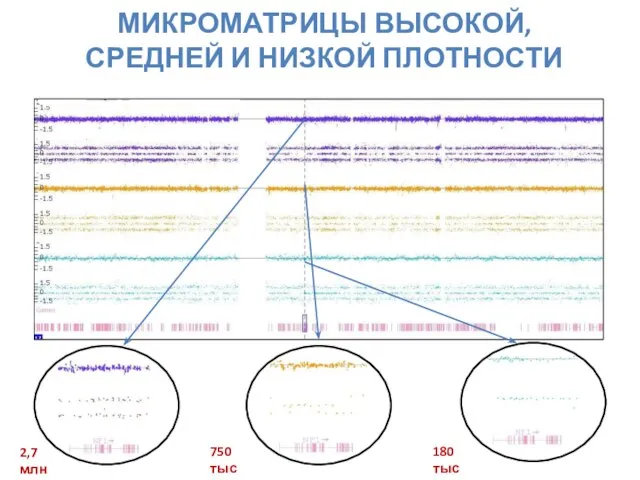
- 18. Распределение проб на микроматрицах разной плотности Микроматрица 400К – 90% Микроматрица 2,67М – 99,6% Клиническая достоверность
- 19. МИКРОМАТРИЦЫ ВЫСОКОЙ, СРЕДНЕЙ И НИЗКОЙ ПЛОТНОСТИ 2,7 млн 750 тыс 180 тыс
- 20. ТЕОРЕТИЧЕСКОЕ ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦ Размер определяемых микроделеций и микродупликаций зависит от параметров маркерных сигналов на
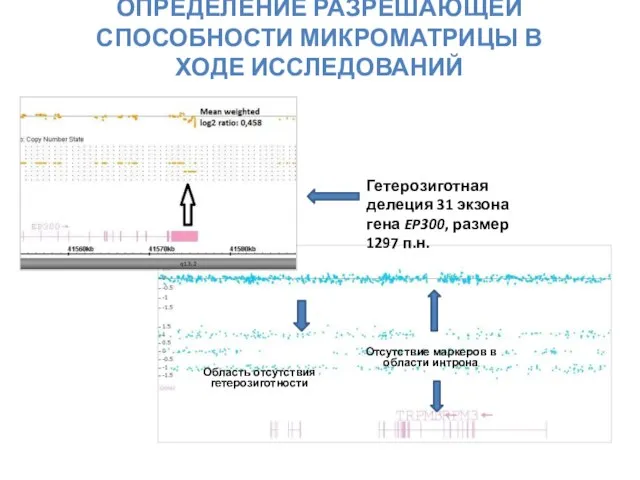
- 21. ОПРЕДЕЛЕНИЕ РАЗРЕШАЮЩЕЙ СПОСОБНОСТИ МИКРОМАТРИЦЫ В ХОДЕ ИССЛЕДОВАНИЙ Отсутствие маркеров в области интрона Область отсутствия гетерозиготности Гетерозиготная
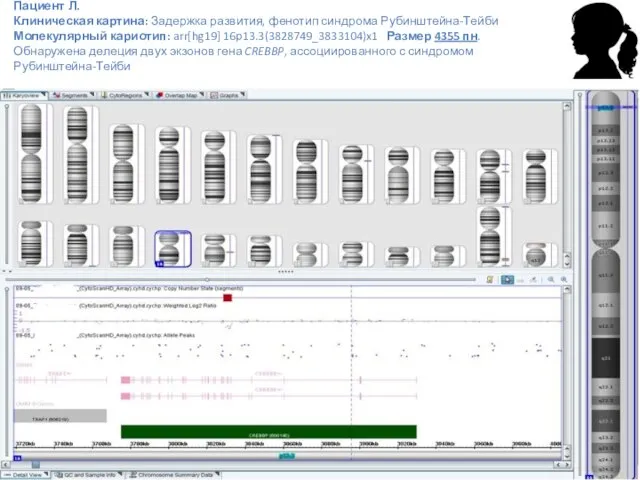
- 22. Пациент Л. Клиническая картина: Задержка развития, фенотип синдрома Рубинштейна-Тейби Молекулярный кариотип: arr[hg19] 16p13.3(3828749_3833104)x1 Размер 4355 пн.
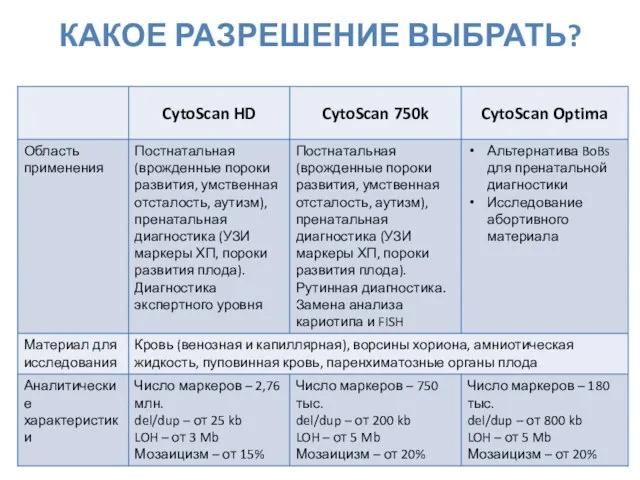
- 23. КАКОЕ РАЗРЕШЕНИЕ ВЫБРАТЬ?
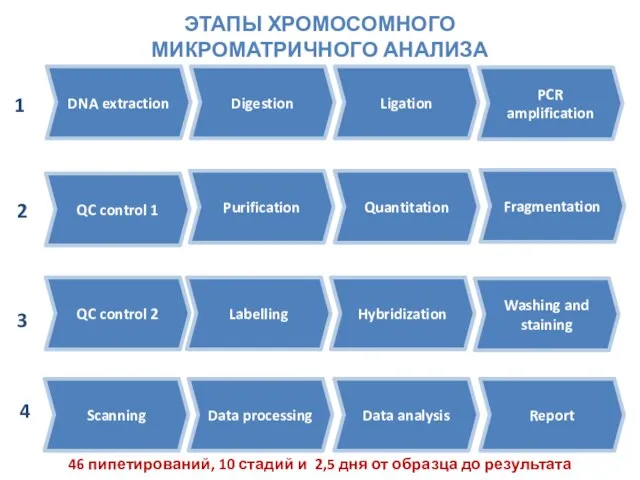
- 24. DNA extraction Digestion Ligation PCR amplification QC control 1 Purification Fragmentation Quantitation QC control 2 Labelling
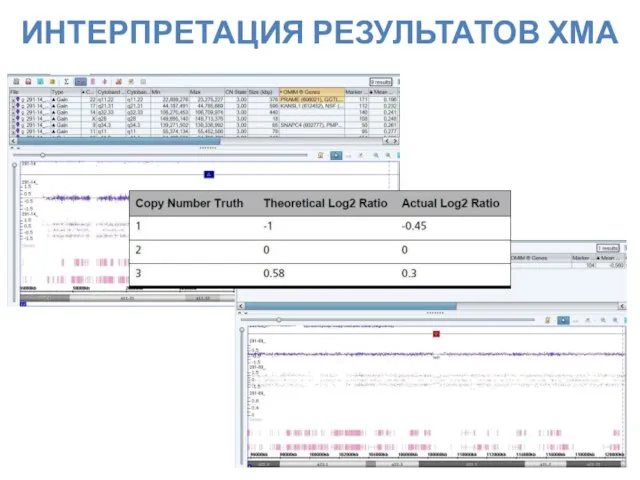
- 25. ИНТЕРПРЕТАЦИЯ РЕЗУЛЬТАТОВ ХМА
- 26. Вариация числа копий (CNV) – это изменение числа копий определенного сегмента ДНК размером, по крайней мере,
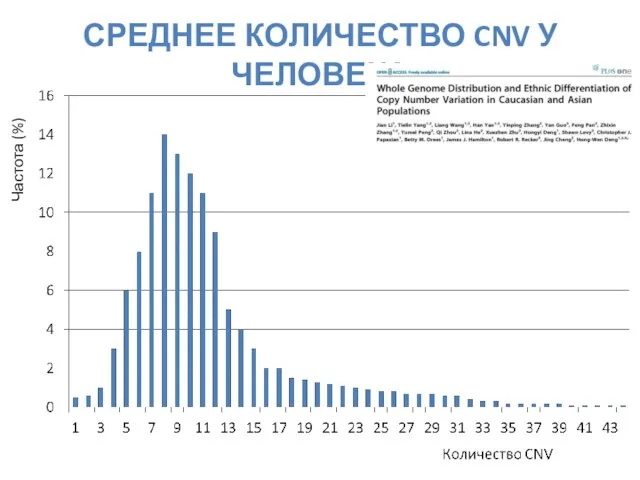
- 27. СРЕДНЕЕ КОЛИЧЕСТВО CNV У ЧЕЛОВЕКА Частота (%)
- 28. ОПРЕДЕЛЕНИЕ КЛИНИЧЕСКОЙ ЗНАЧИМОСТИ (КАУЗАТИВНОСТИ) CNVs Базы данных OMIM, ISCA, DECIPHER, GeneReviews, литературные данные -PubMed
- 29. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
- 30. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
- 31. ПОПУЛЯЦИОННЫЕ БАЗЫ ДАННЫХ CNVs http://dgv.tcag.ca/dgv/app/home
- 32. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
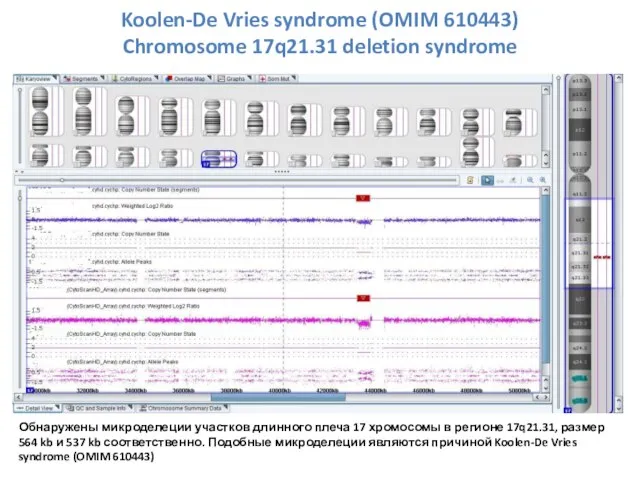
- 33. Обнаружены микроделеции участков длинного плеча 17 хромосомы в регионе 17q21.31, размер 564 kb и 537 kb
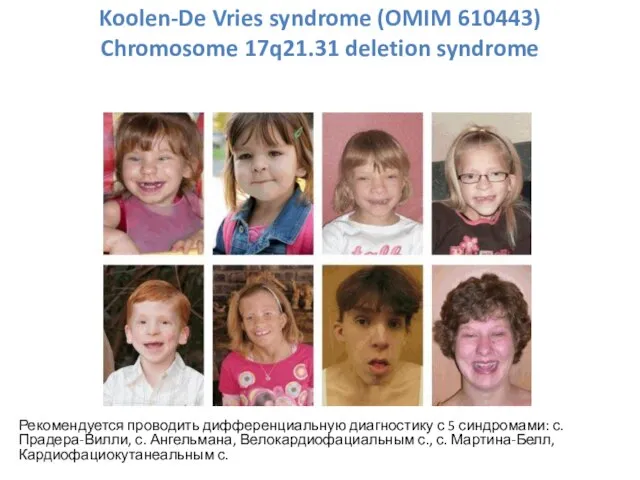
- 34. Рекомендуется проводить дифференциальную диагностику с 5 синдромами: с. Прадера-Вилли, с. Ангельмана, Велокардиофациальным с., с. Мартина-Белл, Кардиофациокутанеальным
- 35. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
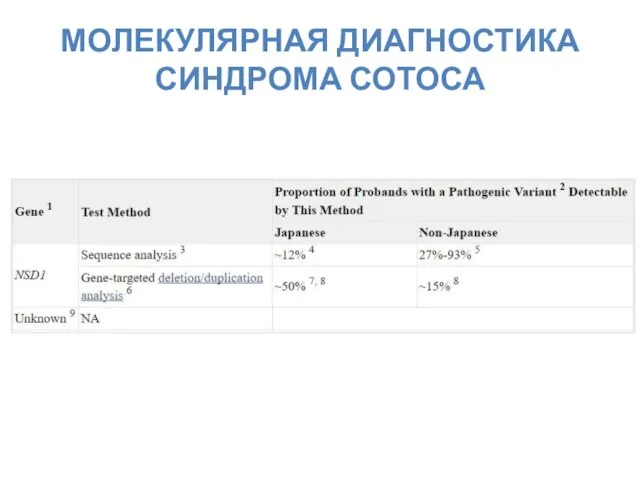
- 36. МОЛЕКУЛЯРНАЯ ДИАГНОСТИКА СИНДРОМА СОТОСА
- 38. ДИФФЕРЕНЦИАЛЬНАЯ ДИАГНОСТИКА СИНДРОМА СОТОСА Синдром Вивера Синдром Беквита-Видемана Синдром Банаян-Райли-Рувалькаба Дупликации 4p Мозаичная трисомия 20p11.2-р12.1 Синдром
- 39. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
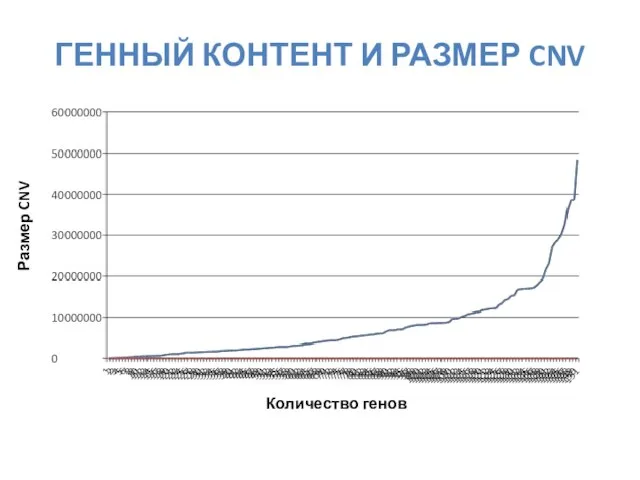
- 40. ГЕННЫЙ КОНТЕНТ И РАЗМЕР CNV Количество генов Размер CNV
- 41. Включена ли CNV в базы данных нормальных вариаций? Содержит ли CNV область, делеция или дупликация которой
- 42. CNV: НАСЛЕДУЕМАЯ ИЛИ DE NOVO? ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ ПОЗВОЛЯЕТ ВЫЯВЛЯТЬ МИКРОДЕЛЕЦИИ… Пациент К., женщина 32 г.
- 43. CNV: НАСЛЕДУЕМАЯ ИЛИ DE NOVO?
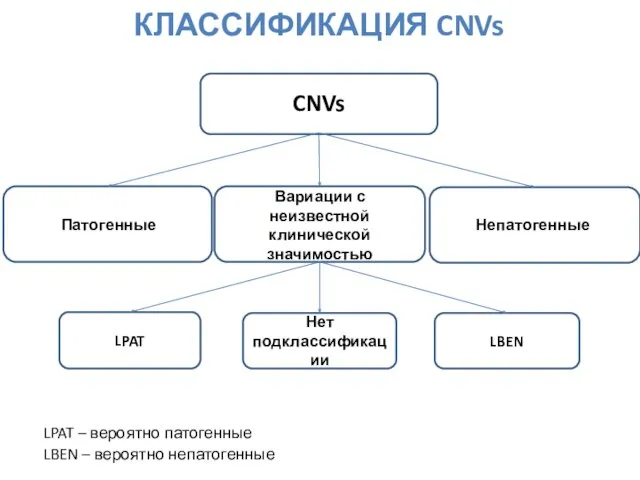
- 44. КЛАССИФИКАЦИЯ CNVs CNVs Патогенные Непатогенные Вариации с неизвестной клинической значимостью LPAT LBEN Нет подклассификации LPAT –
- 45. ПАТОГЕННЫЕ CNVs Описаны в базах OMIM и ORPHANET как делеционный/дупликационный синдром Описаны в 2 и более
- 46. НЕПАТОГЕННЫЕ CNVs CNV была описана во многих рецензируемых публикациях или курируемых базах данных, как непатогенная, особенно
- 47. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО ПАТОГЕННАЯ CNV описана в единственной рецензируемой публикации, но с чёткими
- 48. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, ВЕРОЯТНО НЕПАТОГЕННАЯ В районе CNV отсутствуют гены (но она была включена
- 49. CNVs С НЕИЗВЕСТНОЙ КЛИНИЧЕСКОЙ ЗНАЧИМОСТЬЮ, НЕТ ПОДКЛАССИФИКАЦИИ CNV содержит гены, однако неизвестно, чувствительны ли они к
- 50. НЕОЖИДАННЫЕ КЛИНИЧЕСКИ ЗНАЧИМЫЕ РЕЗУЛЬТАТЫ, НЕ ОТНОСЯЩИЕСЯ К ПРИЧИНЕ НАПРАВЛЕНИЯ Носительство CNVs, связанных с рецессивными заболеваниями Носительство
- 51. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РЕЦЕССИВНЫМИ ЗАБОЛЕВАНИЯМИ Обнаружена мутация в гене, связанном с хорошо описанным в литературе
- 52. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С ЗАБОЛЕВАНИЯМИ С ПОЗДНИМ НАЧАЛОМ Сообщать или нет? Может ли пациент принимать такое
- 53. НОСИТЕЛЬСТВО CNVs, СВЯЗАННЫХ С РИСКОМ НЕОПЛАЗИИ Сообщать или нет? Какие варианты могут быть включены в отчет?
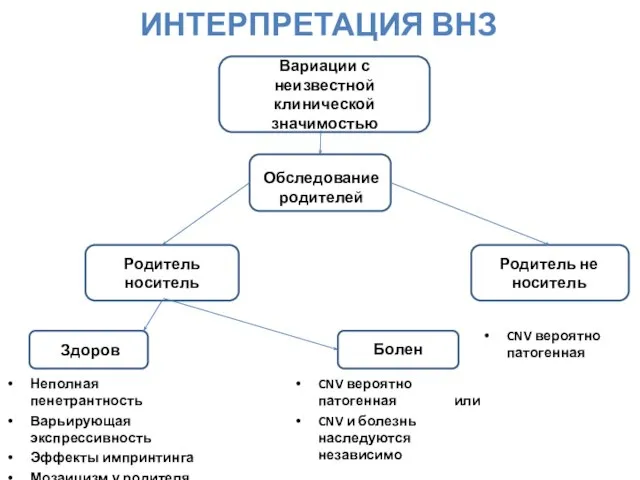
- 54. ИНТЕРПРЕТАЦИЯ ВНЗ Вариации с неизвестной клинической значимостью Обследование родителей Родитель не носитель Родитель носитель Здоров Болен
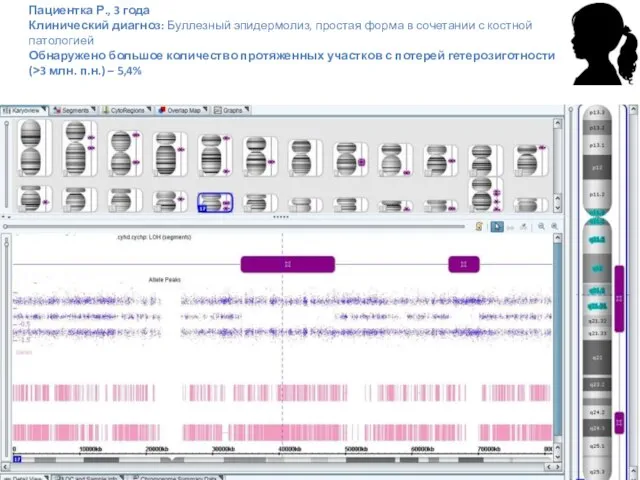
- 55. Пациентка Р., 3 года Клинический диагноз: Буллезный эпидермолиз, простая форма в сочетании с костной патологией Обнаружено
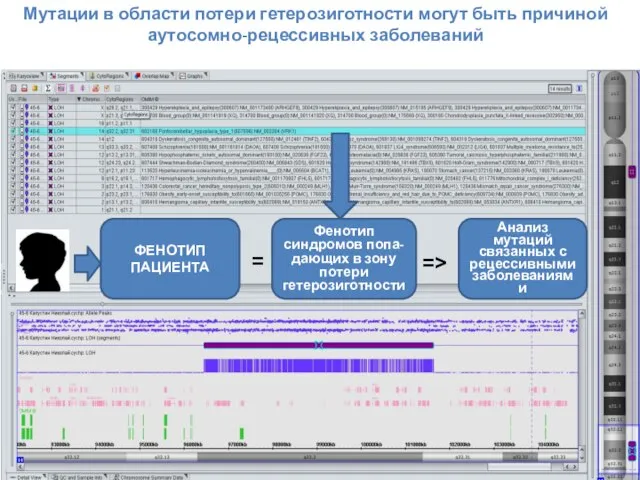
- 56. ФЕНОТИП ПАЦИЕНТА Фенотип синдромов попа-дающих в зону потери гетерозиготности = => Анализ мутаций связанных с рецессивными
- 57. Пациентка Р., 3 года Клинический диагноз: Буллезный эпидермолиз, простая форма в сочетании с костной патологией Обнаружено
- 58. ЗАПИСЬ РЕЗУЛЬТАТОВ ХРОМОСОМНОГО МИКРОМАТРИЧНОГО АНАЛИЗА arr(1-22)x2,(X,Y)x1 – норма, мужской пол arr(1-22,X)x2 – норма, женский пол arr[hg19]
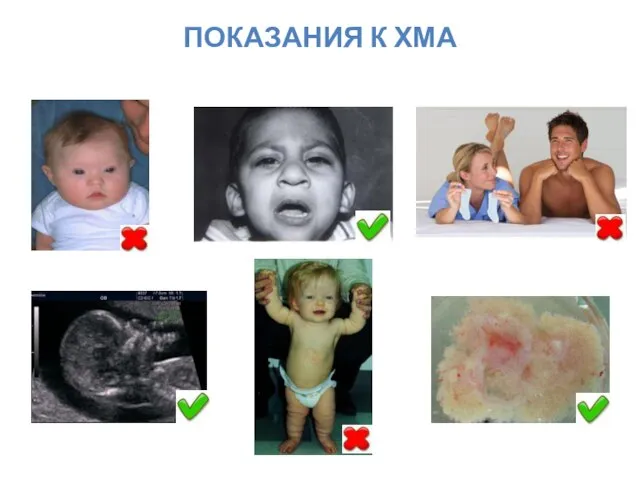
- 59. ПОКАЗАНИЯ К ХМА
- 60. ХМА В ПОСТНАТАЛЬНОЙ ДИАГНОСТИКЕ ХМА является тестом первой линии в постнатальной диагностике причин МВПР с задержкой
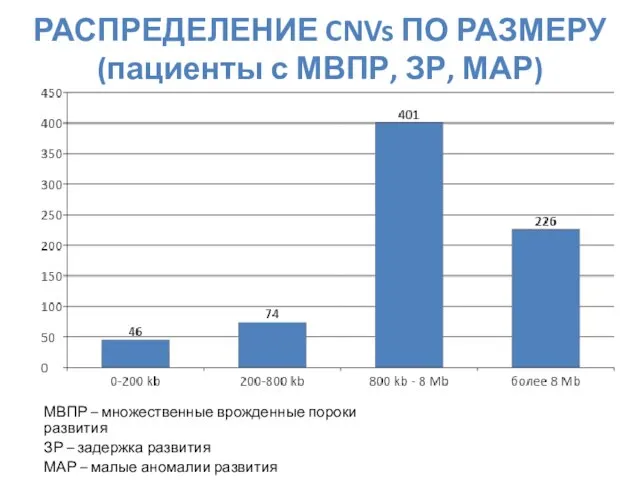
- 61. РАСПРЕДЕЛЕНИЕ CNVs ПО РАЗМЕРУ (пациенты с МВПР, ЗР, МАР) МВПР – множественные врожденные пороки развития ЗР
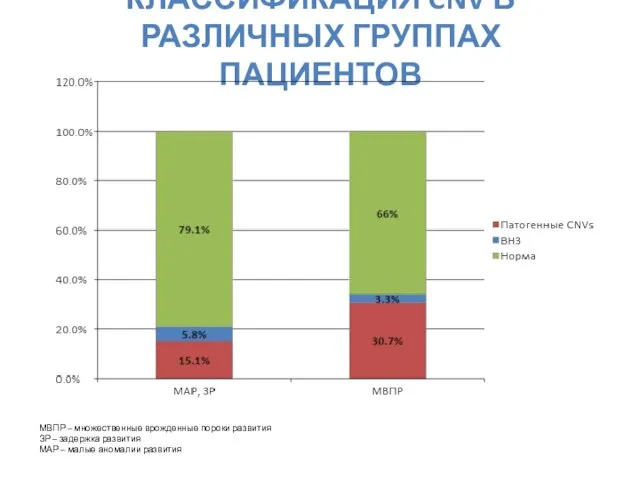
- 62. КЛАССИФИКАЦИЯ CNV В РАЗЛИЧНЫХ ГРУППАХ ПАЦИЕНТОВ МВПР – множественные врожденные пороки развития ЗР – задержка развития
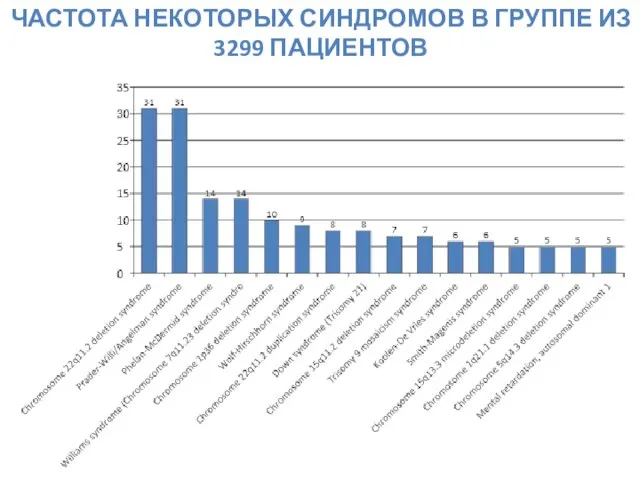
- 63. ЧАСТОТА НЕКОТОРЫХ СИНДРОМОВ В ГРУППЕ ИЗ 3299 ПАЦИЕНТОВ
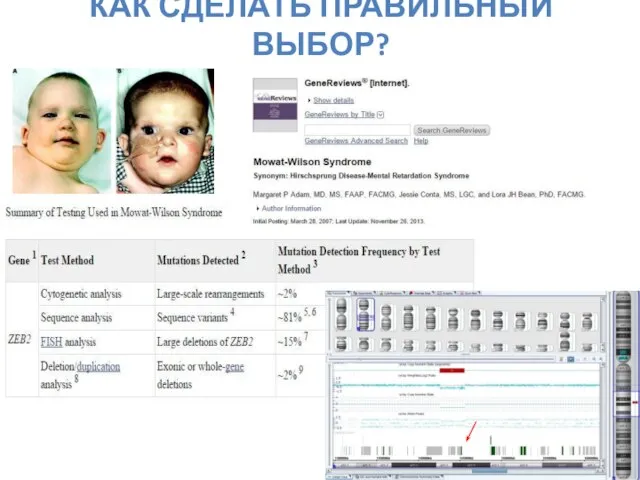
- 64. КАК СДЕЛАТЬ ПРАВИЛЬНЫЙ ВЫБОР?
- 65. Сбалансированные хромосомные перестройки (транслокации, инверсии) Ожидаем ли мы увидеть сбалансированную перестройку у пробанда с МВПР? Точковые
- 66. НЕТ! НУЖНО ЛИ ПОДТВЕРЖДАТЬ РЕЗУЛЬТАТЫ ХМА? Система Геноскан 3000 является единственной апробированной полногеномной технологией и оборудованием,
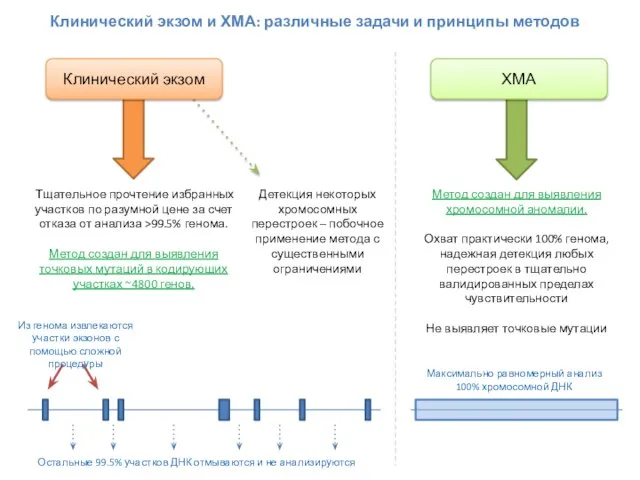
- 67. Клинический экзом Тщательное прочтение избранных участков по разумной цене за счет отказа от анализа >99.5% генома.
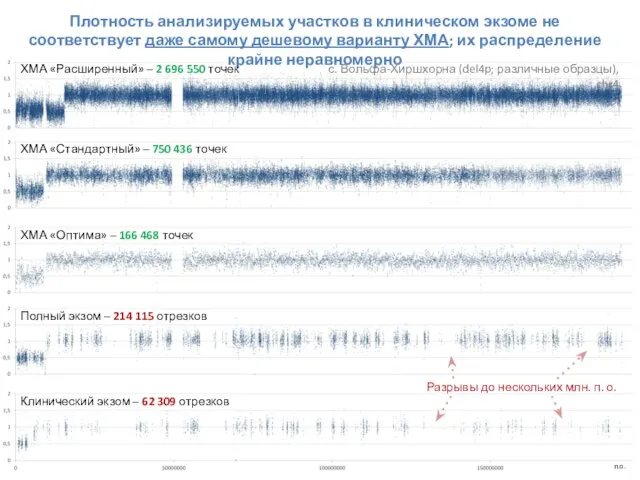
- 68. Плотность анализируемых участков в клиническом экзоме не соответствует даже самому дешевому варианту ХМА; их распределение крайне
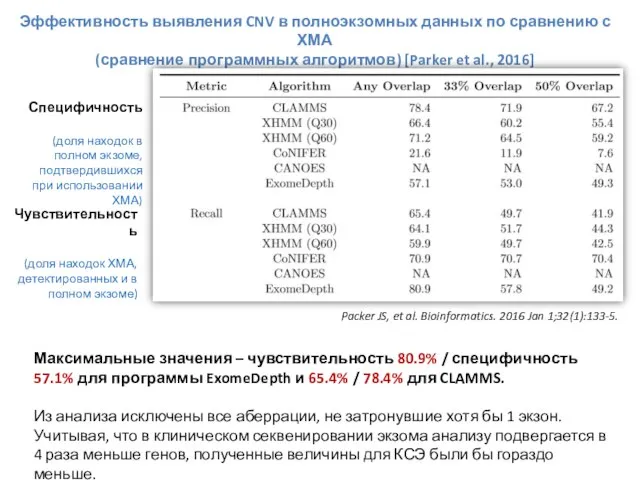
- 69. Эффективность выявления CNV в полноэкзомных данных по сравнению с ХМА (сравнение программных алгоритмов) [Parker et al.,
- 70. Секвенирование экзома, хотя уже подтвердило свою высокую эффективность в выявлении точковых мутаций, пока является сравнительно новым
- 71. ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА ПРОБЛЕМЫ Назначения врача не выполняются Завышенные ожидания от результатов анализа Пациенты
- 72. ГЕНЕТИЧЕСКОЕ КОНСУЛЬТИРОВАНИЕ ПО РЕЗУЛЬТАТАМ ХМА ОСОБЕННОСТИ Некоторые патогенные CNV наследуются Могут иметь неполную пенетрантность Выявление CNV
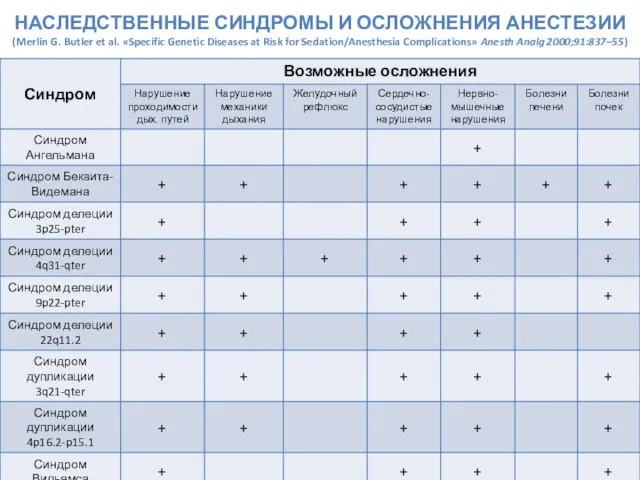
- 73. НАСЛЕДСТВЕННЫЕ СИНДРОМЫ И ОСЛОЖНЕНИЯ АНЕСТЕЗИИ (Merlin G. Butler et al. «Specific Genetic Diseases at Risk for
- 74. ХРОМОСОМНЫЙ МИКРОМАТРИЧНЫЙ АНАЛИЗ В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ ПОКАЗАНИЯ Высокий риск ХП по результатам НИПТ Ребенок с хромосомной
- 75. ХМА в качестве замены анализа кариотипа рекомендован при выявлении пороков развития плода или мертворождении врач должен
- 76. ЭФФЕКТИВНОСТЬ ХМА В ПРЕНАТАЛЬНОЙ ДИАГНОСТИКЕ УЗ-аномалии – 6% Другие причины – 1,7% от общего числа случаев
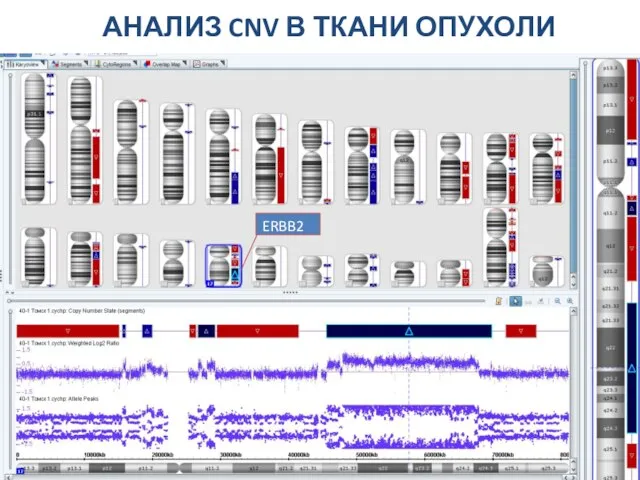
- 77. АНАЛИЗ CNV В ТКАНИ ОПУХОЛИ ERBB2
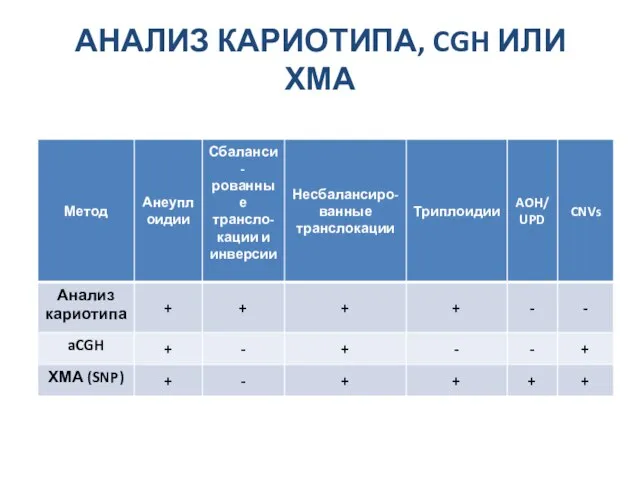
- 78. АНАЛИЗ КАРИОТИПА, CGH ИЛИ ХМА
- 80. Скачать презентацию













































 Созылмалы энтерит
Созылмалы энтерит ЛФК при анкилозирующем спондилоартрите
ЛФК при анкилозирующем спондилоартрите Теория поколений. Поколения X, Y, Z
Теория поколений. Поколения X, Y, Z Иммуноглобулин человека нормальный
Иммуноглобулин человека нормальный Преэклампсия. Факторы риска
Преэклампсия. Факторы риска Правила личной гигиены
Правила личной гигиены Осложнения сахарного диабета
Осложнения сахарного диабета Сестринский уход в педиатрии. Наследственные и врожденные заболевания новорожденных
Сестринский уход в педиатрии. Наследственные и врожденные заболевания новорожденных Американская классическая школа психологии.Теория Г. Эмерсона
Американская классическая школа психологии.Теория Г. Эмерсона Интерннің өзіндік жұмысы
Интерннің өзіндік жұмысы Лабораторная диагностика сердечно-сосудистых заболеваний
Лабораторная диагностика сердечно-сосудистых заболеваний Профессиональный стандарт. Педагог-психолог (психолог в сфере образования)
Профессиональный стандарт. Педагог-психолог (психолог в сфере образования) Балалардағы ауыз қуысының аурулары
Балалардағы ауыз қуысының аурулары Сирингомиелия. Байланысқан сирингомиелия
Сирингомиелия. Байланысқан сирингомиелия Мультисистемные проявления заболеваний органов пищеварения
Мультисистемные проявления заболеваний органов пищеварения Функціональна діагностика захворювань нервової системи
Функціональна діагностика захворювань нервової системи Грибковые заболевания кожи. Микозы волосистой части головы
Грибковые заболевания кожи. Микозы волосистой части головы Гигиена детей и подростков
Гигиена детей и подростков Наружный генитальный эндометриоз. Классификация. Этиопатогенез. Диагностика
Наружный генитальный эндометриоз. Классификация. Этиопатогенез. Диагностика Ишемический инфаркт мозга
Ишемический инфаркт мозга Авторский алгоритм получения предварительных оттисков у пациентов с полным отсутствием зубов
Авторский алгоритм получения предварительных оттисков у пациентов с полным отсутствием зубов Методы восстановительного лечения в акушерстве и гинекологии
Методы восстановительного лечения в акушерстве и гинекологии Бүйрек амилоидозы
Бүйрек амилоидозы Медицина Древней Индии и Китая. Лекция 3
Медицина Древней Индии и Китая. Лекция 3 Оценка тяжести состояния пациентов с острым панкреатитом
Оценка тяжести состояния пациентов с острым панкреатитом Цитокинотерапия. Цитокиндер
Цитокинотерапия. Цитокиндер Anaflakti̇k şok
Anaflakti̇k şok Thinking About Psychology. The Science of Mind and Behavior 3e
Thinking About Psychology. The Science of Mind and Behavior 3e