Содержание
- 2. Существует много программ, интегрирующих различные биоинформационные инструменты и позволяющие молекулярному биологу планировать эксперименты и обрабатывать их
- 3. Геном вируса клещевого энцефалита
- 4. Геном вируса краснухи
- 5. · BioEdit · Genbank · Fasta · NBRF/PIR · Phylip 3.2 / 2 · Phylip 4
- 6. FASTA-формат >Rattus_norvegicus | Rattus norvegicus heat shock 20kDa protein (Loc192245), mRNA GCAGGATGGAGATCCGGGTGCCTGTGCAGCCTTCTTGGCTGCGCCGTGCTTCAGCTCCTTTACCGGGTTTTTCCACTCCGGGACGCCTCTTTGACCAGCGTTTCGGCGAAGGGCTGCTTGAGGCAGAGCTGGCTTC >Homo_sapiens | Homo sapiens
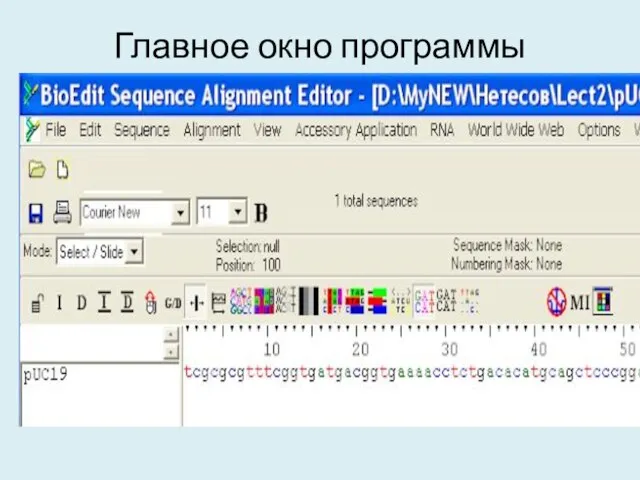
- 7. Главное окно программы
- 8. Меню «Файл»
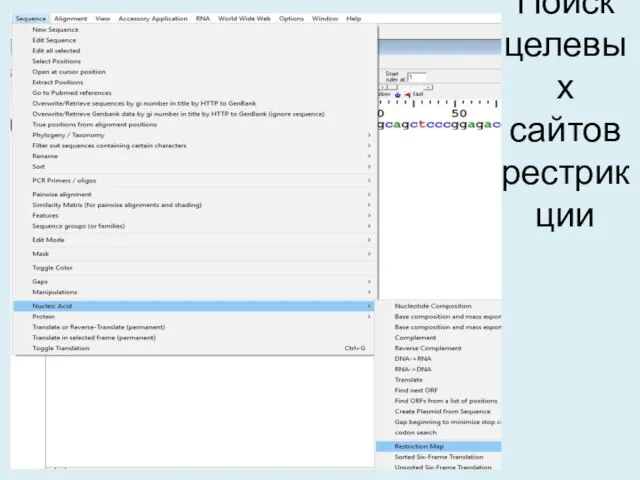
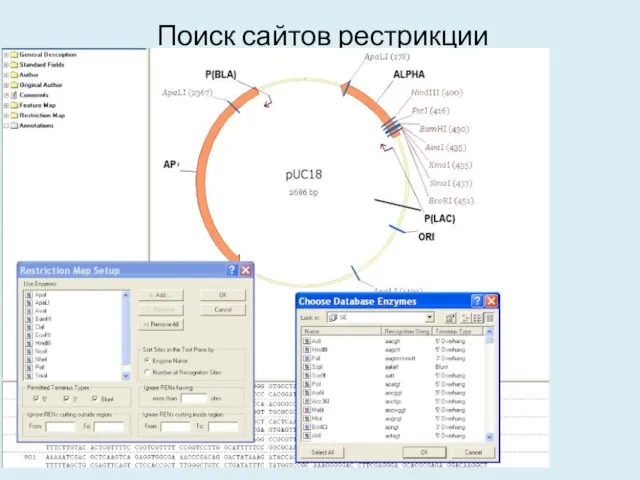
- 9. Поиск целевых сайтов рестрикции
- 10. Поиск целевых сайтов рестрикции
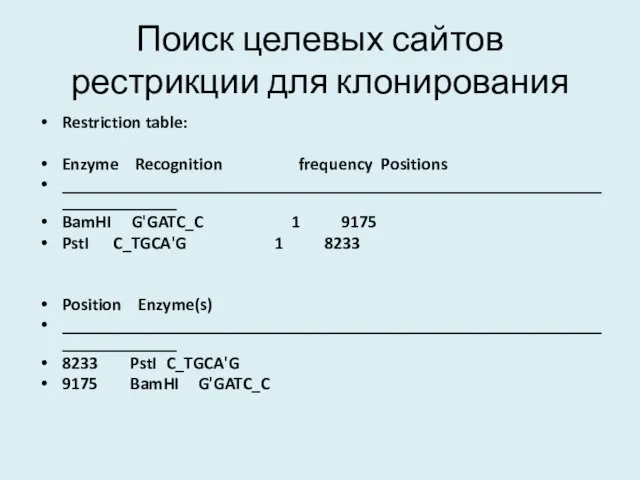
- 11. Поиск целевых сайтов рестрикции для клонирования
- 12. Поиск целевых сайтов рестрикции для клонирования Restriction table: Enzyme Recognition frequency Positions __________________________________________________________________________ BamHI G'GATC_C 1
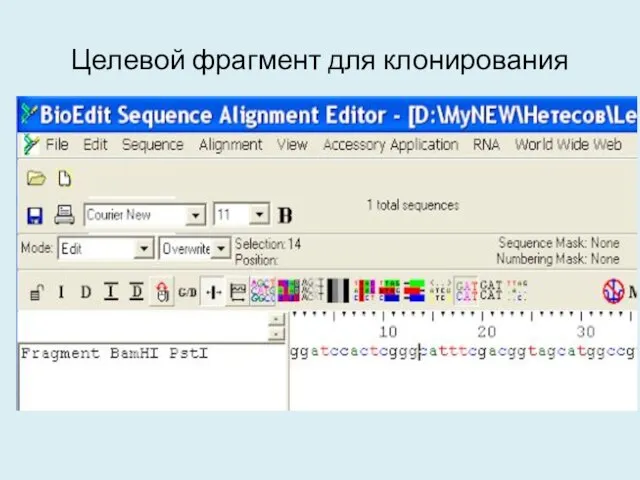
- 13. Целевой фрагмент для клонирования
- 14. Вектор для клонирования
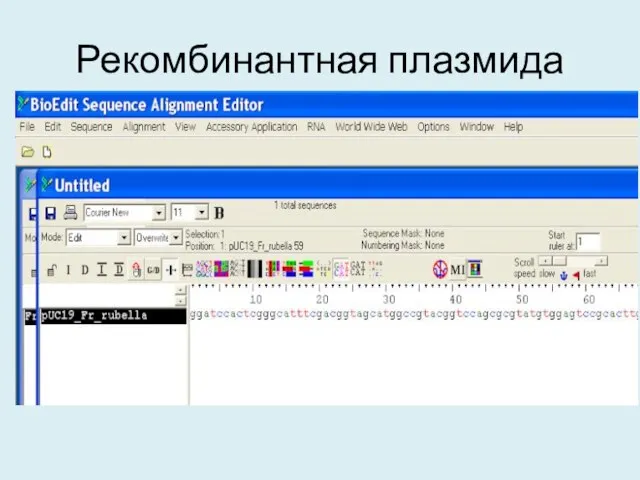
- 15. Рекомбинантная плазмида
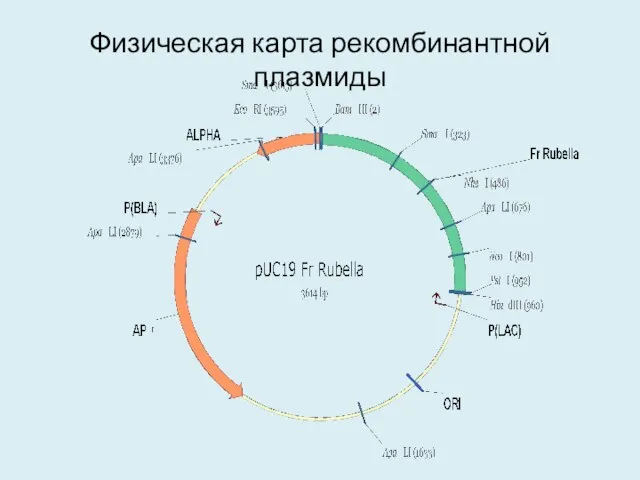
- 16. Физическая карта рекомбинантной плазмиды
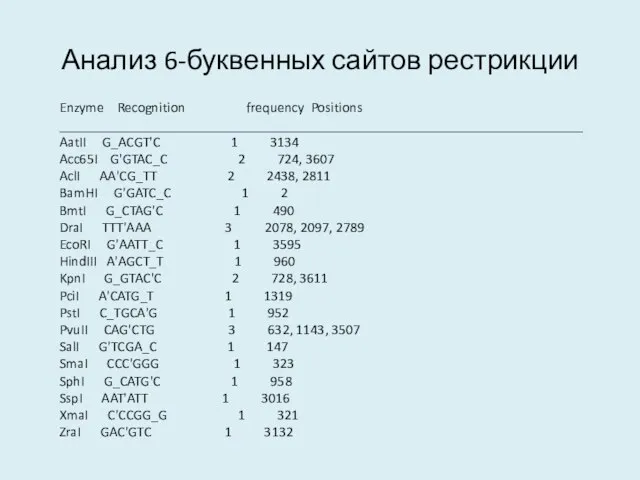
- 17. Анализ 6-буквенных сайтов рестрикции Enzyme Recognition frequency Positions __________________________________________________________________________ AatII G_ACGT'C 1 3134 Acc65I G'GTAC_C 2
- 18. Четыре режима ручного выравнивания. Встраивание и редактирование цвета с помощью отдельных таблиц и полный контроль над
- 19. Сортировать последовательности по имени, LOCUS, DEFINITION, ACCESSION, PID/NID, REFERENCES, COMMENTS или по частоте остатков в выбранной
- 20. Поиск ОРТ с пользовательскими настройками Трансляция последовательностей нуклеиновых кислот с информацией об использовании кодонов, выбор одно-
- 21. Обзор ферментов рестрикции Возможность манипулировать последовательностями длиной не более 4,6 МБ. Six-frame translations capable of raw
- 22. Настройка и запуск вспомогательных приложений через графический интерфейс BioEdit. BioEdit поставляется с: TreeView CAP-ассемблер FastDNml Программы
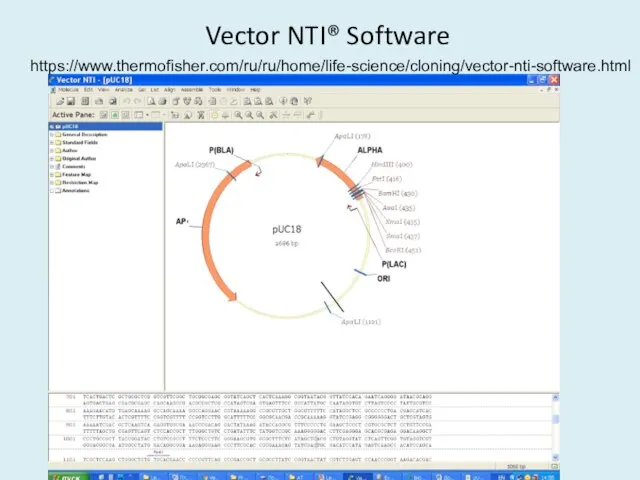
- 23. Vector NTI® Software https://www.thermofisher.com/ru/ru/home/life-science/cloning/vector-nti-software.html
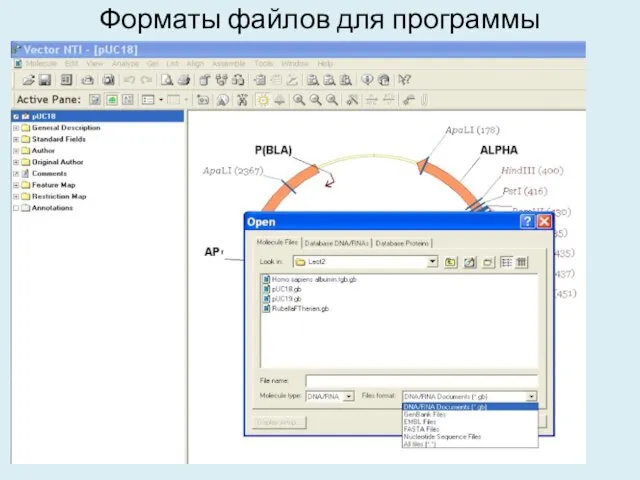
- 25. Форматы файлов для программы
- 26. GenBank-формат LOCUS pUC18 2686 bp DNA circular 13-APR-2013 DEFINITION ATCC 37253: very usable general purpose vector.
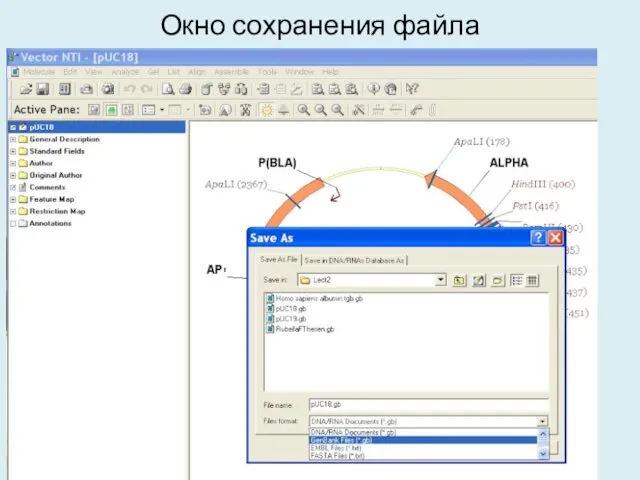
- 27. Окно сохранения файла
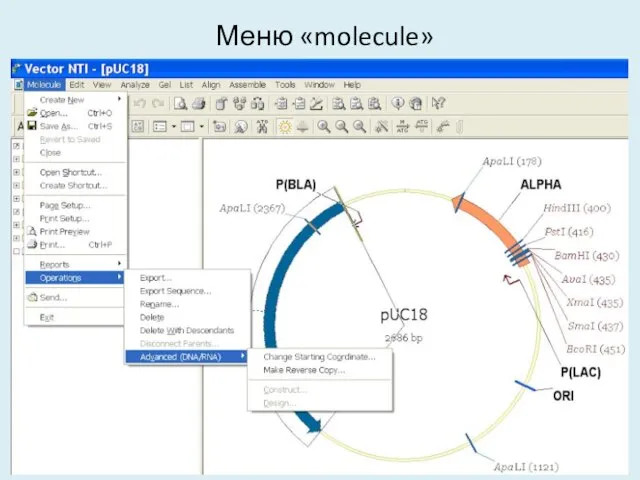
- 28. Меню «molecule»
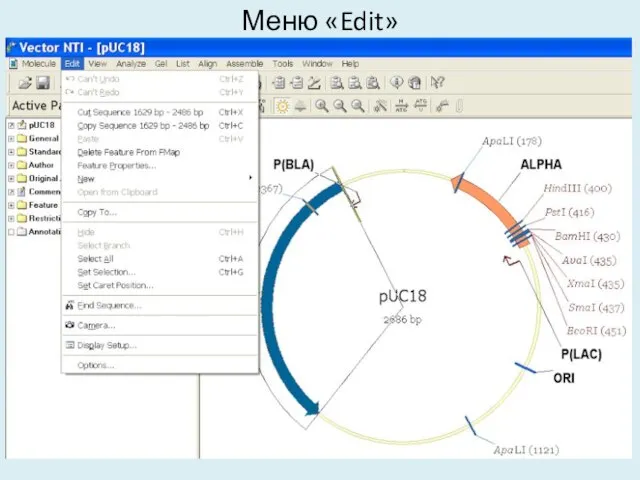
- 29. Меню «Edit»
- 30. Добавление локуса в карту
- 31. Поиск последовательности
- 32. Меню «View»
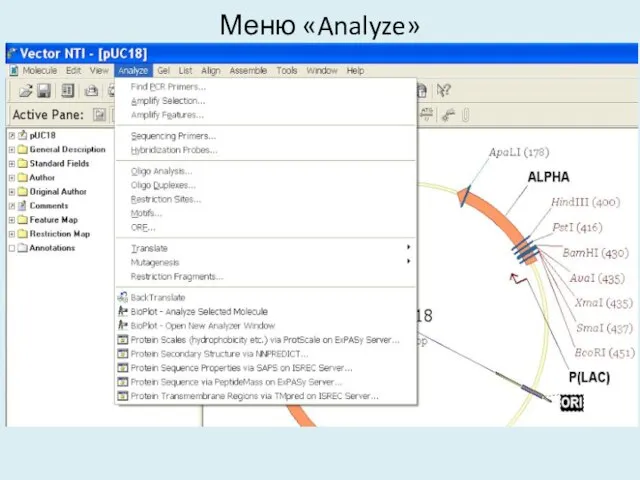
- 33. Меню «Analyze»
- 34. Поиск сайтов рестрикции
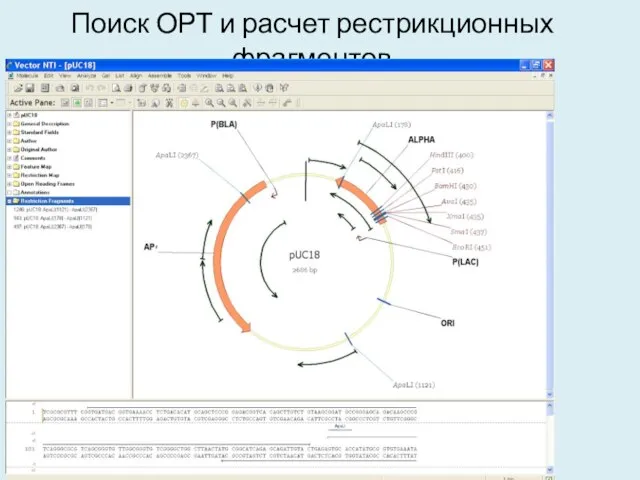
- 35. Поиск ОРТ и расчет рестрикционных фрагментов
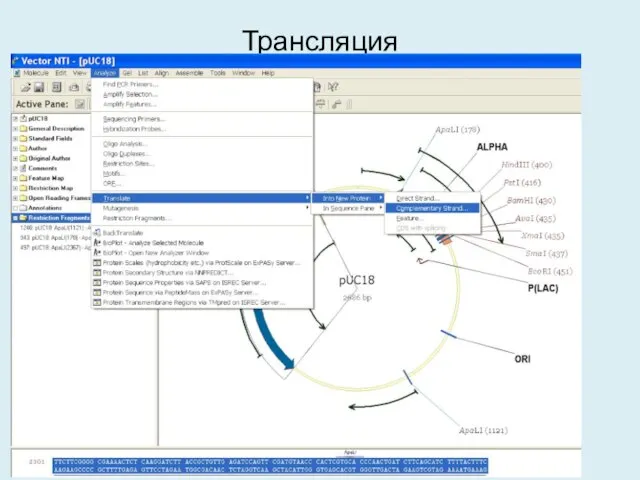
- 36. Трансляция
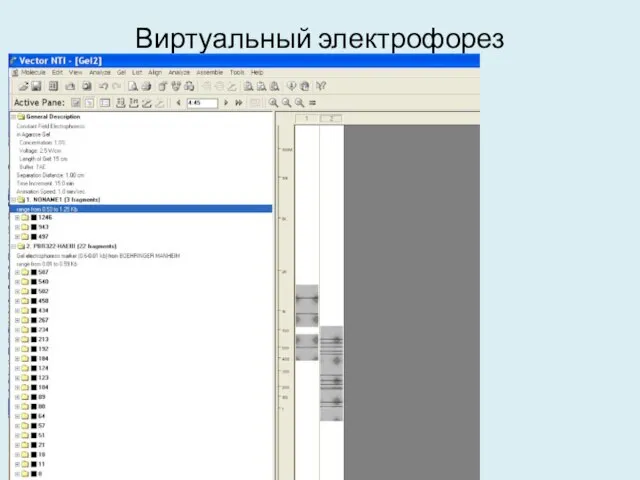
- 37. Виртуальный электрофорез
- 38. Физическая карта рекомбинантной плазмиды
- 39. Физическая карта рекомбинантной плазмиды
- 40. Программы для повседневной молекулярной биологии
- 41. Программы для повседневной молекулярной биологии SimVector Geneious DNASTAR Lasergene Unipro UGENE (free)
- 42. UGENE - Интегрированные инструменты биолога ugene.net/ru/
- 43. Полезные Интернет-сайты: 1. httphttp://http://wwwhttp://www.http://www.ncbihttp://www.ncbi.http://www.ncbi.nlmhttp://www.ncbi.nlm.http://www.ncbi.nlm.nihhttp://www.ncbi.nlm.nih.http://www.ncbi.nlm.nih.govhttp://www.ncbi.nlm.nih.gov/ 2. httphttp://http://wwwhttp://www.http://www.ebihttp://www.ebi.http://www.ebi.achttp://www.ebi.ac.http://www.ebi.ac.ukhttp://www.ebi.ac.uk/ 3. http://web.expasy.org/ 4. httphttp://http://evolutionhttp://evolution.http://evolution.geneticshttp://evolution.genetics.http://evolution.genetics.washingtonhttp://evolution.genetics.washington.http://evolution.genetics.washington.eduhttp://evolution.genetics.washington.edu/http://evolution.genetics.washington.edu/phyliphttp://evolution.genetics.washington.edu/phylip/http://evolution.genetics.washington.edu/phylip/softwarehttp://evolution.genetics.washington.edu/phylip/software.http://evolution.genetics.washington.edu/phylip/software.htmlhttp://evolution.genetics.washington.edu/phylip/software.html#http://evolution.genetics.washington.edu/phylip/software.html#methods 5. httphttp://http://blasthttp://blast.http://blast.ncbihttp://blast.ncbi.http://blast.ncbi.nlmhttp://blast.ncbi.nlm.http://blast.ncbi.nlm.nihhttp://blast.ncbi.nlm.nih.http://blast.ncbi.nlm.nih.govhttp://blast.ncbi.nlm.nih.gov/http://blast.ncbi.nlm.nih.gov/Blasthttp://blast.ncbi.nlm.nih.gov/Blast.http://blast.ncbi.nlm.nih.gov/Blast.cgi 6. httphttp://http://maffthttp://mafft.http://mafft.cbrchttp://mafft.cbrc.http://mafft.cbrc.jphttp://mafft.cbrc.jp/http://mafft.cbrc.jp/alignmenthttp://mafft.cbrc.jp/alignment/http://mafft.cbrc.jp/alignment/serverhttp://mafft.cbrc.jp/alignment/server/http://mafft.cbrc.jp/alignment/server/indexhttp://mafft.cbrc.jp/alignment/server/index.http://mafft.cbrc.jp/alignment/server/index.html 7. httphttp://http://unafoldhttp://unafold.http://unafold.rnahttp://unafold.rna.http://unafold.rna.albanyhttp://unafold.rna.albany.http://unafold.rna.albany.eduhttp://unafold.rna.albany.edu/?http://unafold.rna.albany.edu/?qhttp://unafold.rna.albany.edu/?q=http://unafold.rna.albany.edu/?q=mfoldhttp://unafold.rna.albany.edu/?q=mfold/http://unafold.rna.albany.edu/?q=mfold/DNAhttp://unafold.rna.albany.edu/?q=mfold/DNA-http://unafold.rna.albany.edu/?q=mfold/DNA-Foldinghttp://unafold.rna.albany.edu/?q=mfold/DNA-Folding-http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form
- 45. Скачать презентацию
























 Прием 1. Принцип дробления
Прием 1. Принцип дробления Шыны өндірісіндегі жаңа бағыттар
Шыны өндірісіндегі жаңа бағыттар Продукция химической и связанных с ней отраслей промышленности
Продукция химической и связанных с ней отраслей промышленности Теория графов: основные понятия
Теория графов: основные понятия the_phantom_of_the_opera
the_phantom_of_the_opera Prezentatsia6_1
Prezentatsia6_1 Гидрофизические свойства строительных материалов
Гидрофизические свойства строительных материалов Ремонт гидравлических гасителей колебаний
Ремонт гидравлических гасителей колебаний Происхождение человека
Происхождение человека Современные проблемы науки и производства в области строительства
Современные проблемы науки и производства в области строительства Вводная_ЕГЭ
Вводная_ЕГЭ Правовое регулирование организации и деятельности органов государственной власти. Электронные библиотечные системы
Правовое регулирование организации и деятельности органов государственной власти. Электронные библиотечные системы ЧIал квадармир, ам хуьх. ЧIал руьгьдин девлет я!
ЧIал квадармир, ам хуьх. ЧIал руьгьдин девлет я! UCS. Универсальная зарядная станция
UCS. Универсальная зарядная станция Going shopping
Going shopping Основы физиологии поведения и научения
Основы физиологии поведения и научения Послание к Фессалоникийцам
Послание к Фессалоникийцам Катионит
Катионит Растительный и животный мир. Крым
Растительный и животный мир. Крым 20170212_pvl
20170212_pvl Технология возделывания и уборки сена
Технология возделывания и уборки сена 20131119_drevnerimskaya_literatura
20131119_drevnerimskaya_literatura Головные_уборы
Головные_уборы Игра Мастер своего дела
Игра Мастер своего дела Евгений Яковлевич Шульгин (1873-1937)
Евгений Яковлевич Шульгин (1873-1937) Картотека по экспериментальной деятельности с детьми дошкольного возраста
Картотека по экспериментальной деятельности с детьми дошкольного возраста Павленко презентация
Павленко презентация Личность и Время - Василий Зуев
Личность и Время - Василий Зуев